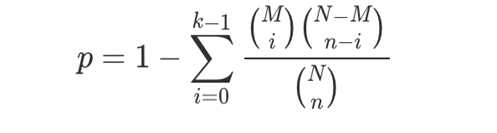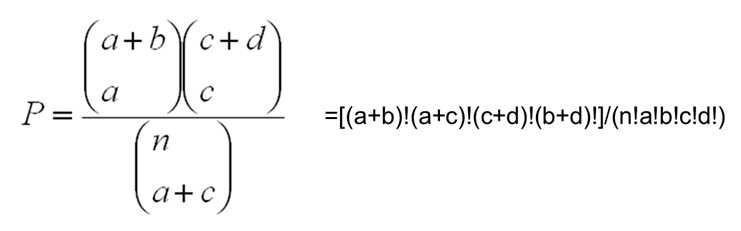目录
一、基因集功能富集分析(Gene Set Enrichment Analysis)
基因功能富集分析,是指借助各类数据库和分析工具进行统计分析,挖掘在数据库中与我们要研究的生物学问题具有显著相关性的基因功能类别。
For example, given a set of genes that are up-regulated under certain conditions, an enrichment analysis will find which Ontology terms are over-represented (or under-represented) using annotations for that gene set.
通俗来说:富集分析是基于一个先验的知识图谱将输入内容进行聚类分析,得到聚类后结果。
上句话中逐个概念解析:
输入内容:一组基因或者基因产物(RNA、蛋白质)
知识图谱:往往是由符号连接的树状结构(DAG有向无环图)。
(1)有可能是描述功能的知识图谱,例如GO:描述“单个基因如何在分子,细胞和生物水平上的生物学贡献”。
(2)也可能是描述代谢通路的知识图谱,例如KEGG:一个整合了基因组、化学和系统功能信息的综合数据库,其中用的最多的数据库是描述基因通路的KEGG pathway
聚类:基于知识图谱进行映射分类
输出:
(1)富集结果。输入内容所映射的分类结果,一般包括数量和p值
(2)可以查看具体的分类的注释信息
(3)具体分类所对应的局部知识图谱
简单来说,就是把我们挑出来的基因归归类,看看哪些基因的功能和我们的研究相关
二、富集分析算法
统计原理是用超几何分布型来检验一组基因(共表达或差异表达)中某个功能类的显著性,通过离散分布的显著性分析、富集度分析和假阳性分析, 得出与实验目的有显著关联的、低假阳性率的及靶向性的基因功能类别。
2.1 超几何分布
Over Representation Analysis (ORA) (Boyle et al. 2004) is a widely used approach to determine whether known biological functions or processes are over-represented (= enriched) in an experimentally-derived gene list, e.g. a list of differentially expressed genes (DEGs). The p-value can be calculated by hypergeometric distribution.
以上公式中:
N为所有基因中具有pathway/GO term注释的基因数目;
n为N中差异表达基因的数目;
M为所有基因中注释为某特定pathway/GO term的基因数目;
m为注释为某特定pathway/GO term的差异表达基因数目。
通过计算得到的P value会进一步经过多重检验校正,得到FDR值。然后以一定的FDR为阈值,满足此条件的pathway/GO term定义为在差异表达基因中显著富集的pathway/GO term。
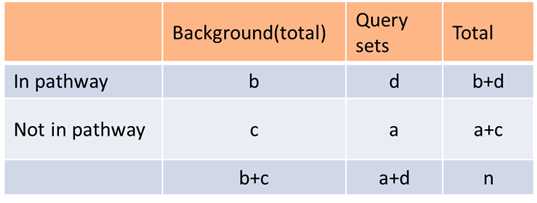
2.2 Fisher精确检验
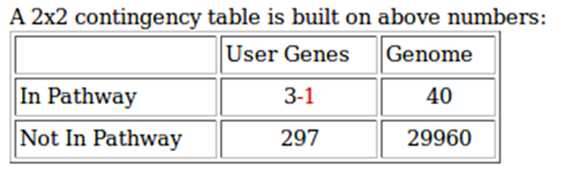
例:In human genome background (30,000 gene total), 40 genes are involved in p53 signaling pathway. A given gene list has found that 3 out of 300 belong to p53 signaling pathway. Then we ask the question if 3/300 is more than random chance comparing to the human background of 40/30000.
三、富集分析工具
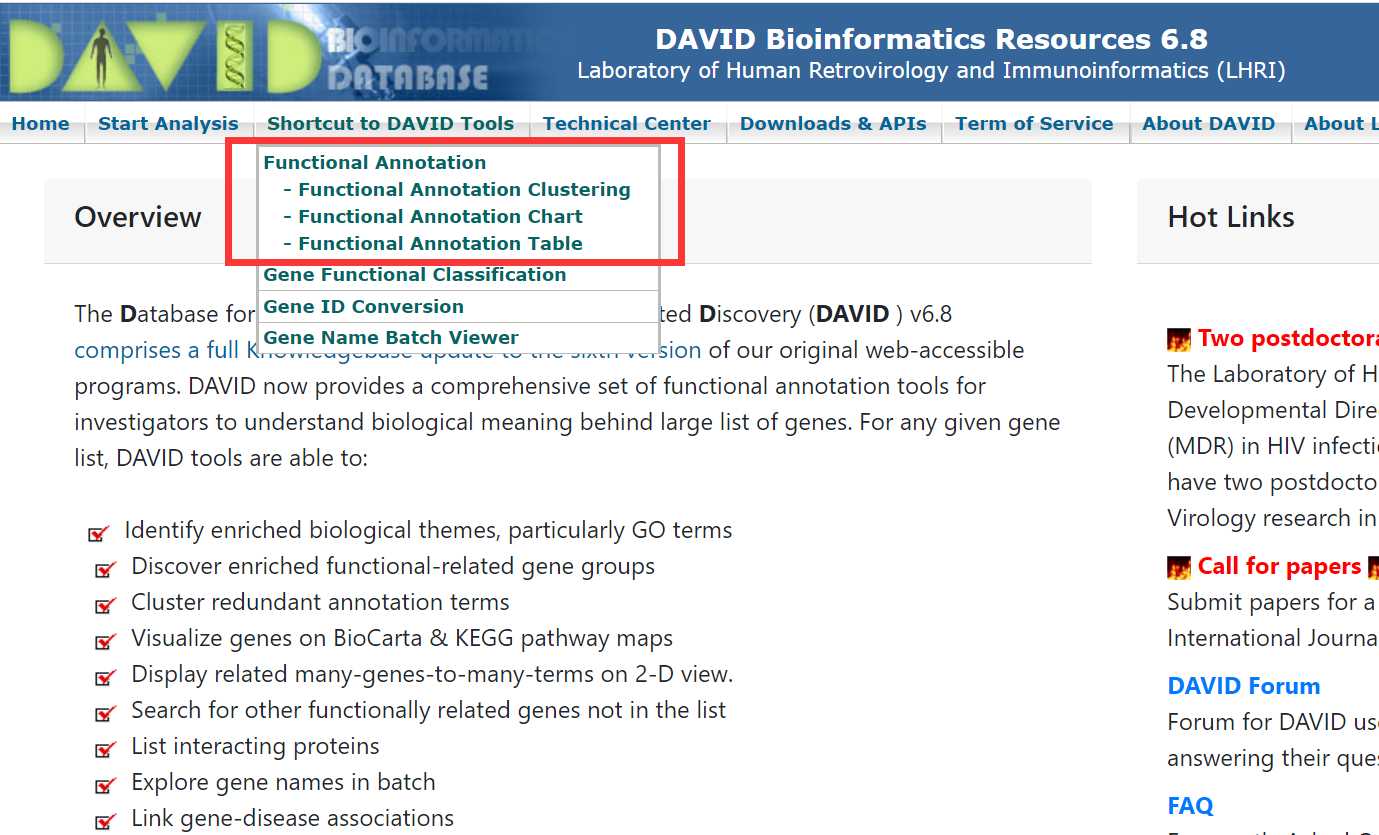
3.1 DAVID
DAVID是一个综合工具,提供基因间ID的转换、基因功能的分类和基因富集分析。DAVID平台是通过Fisher精确检验对gene富集分析。
DAVID网址:
https://david.ncifcrf.gov/
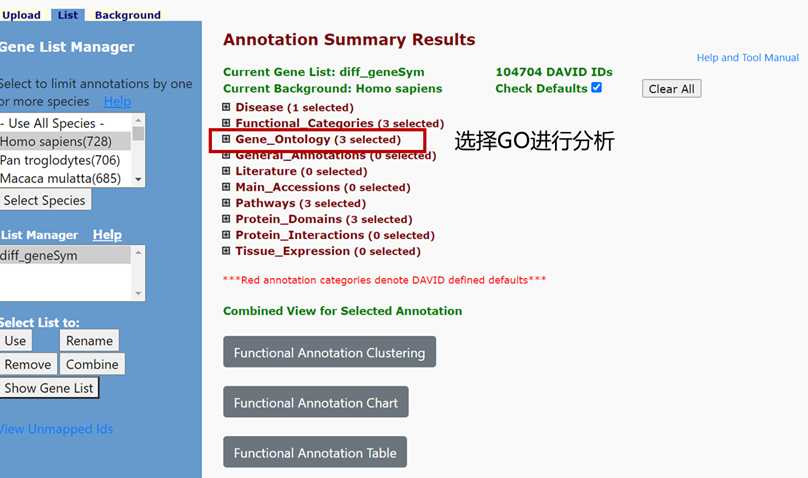
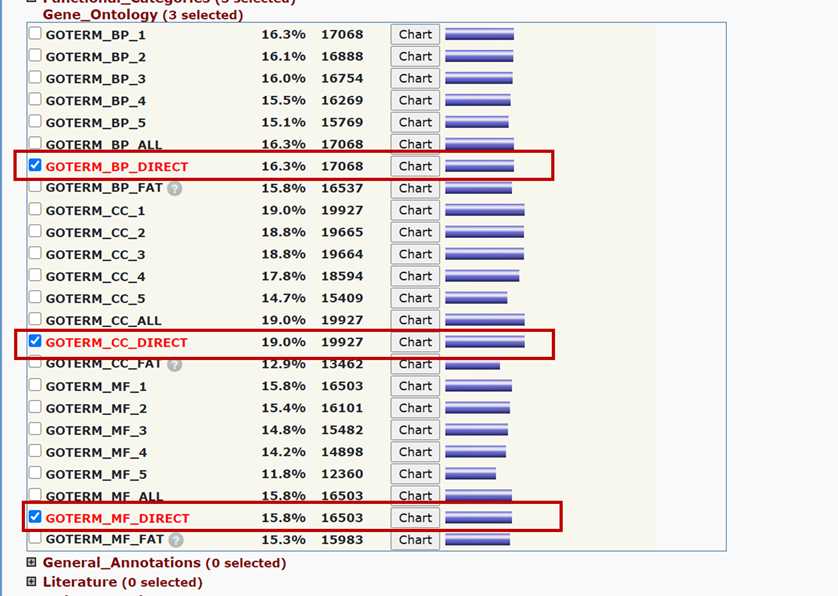
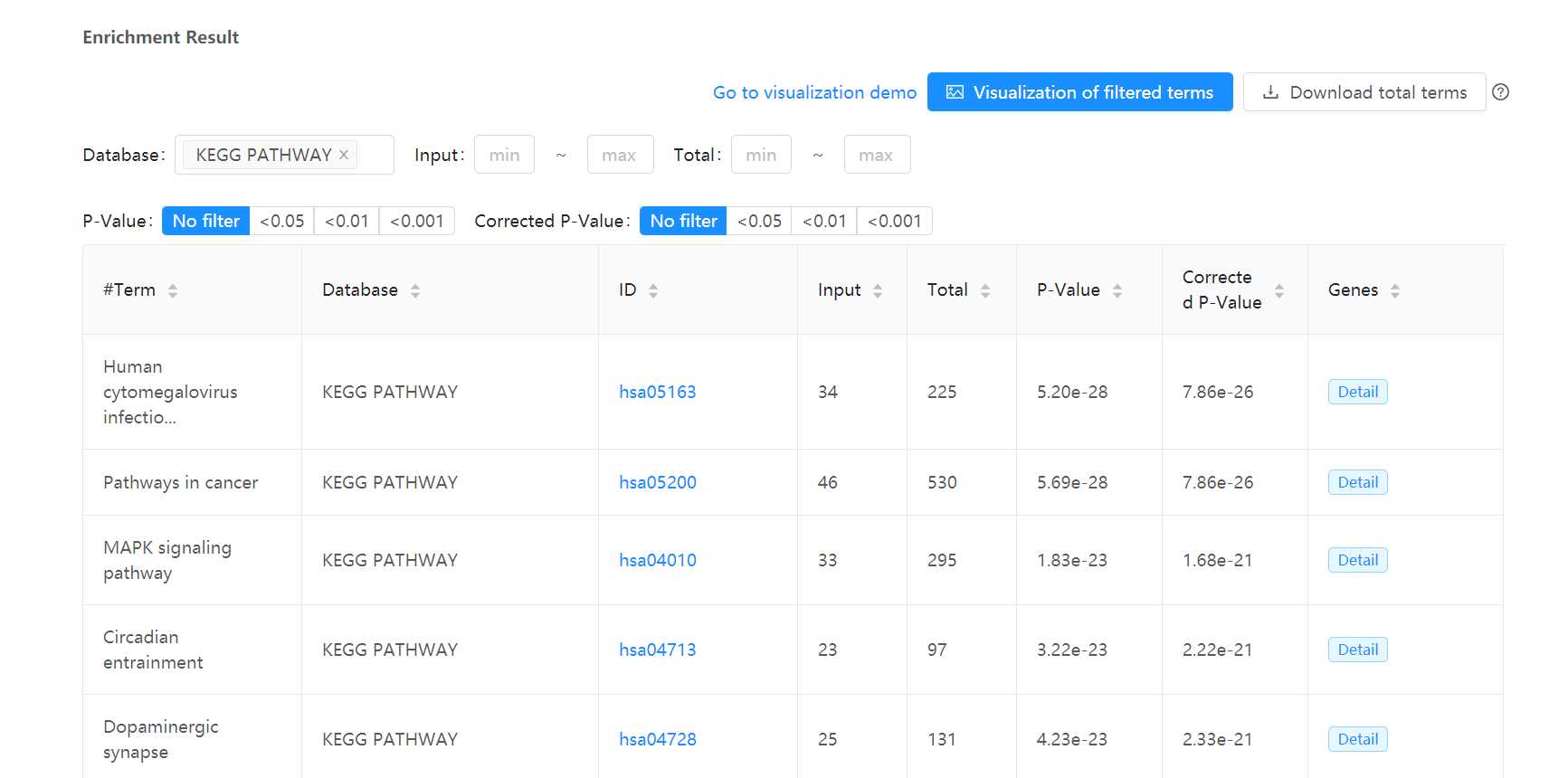
实例分析
一般主要关注它标红的几项:
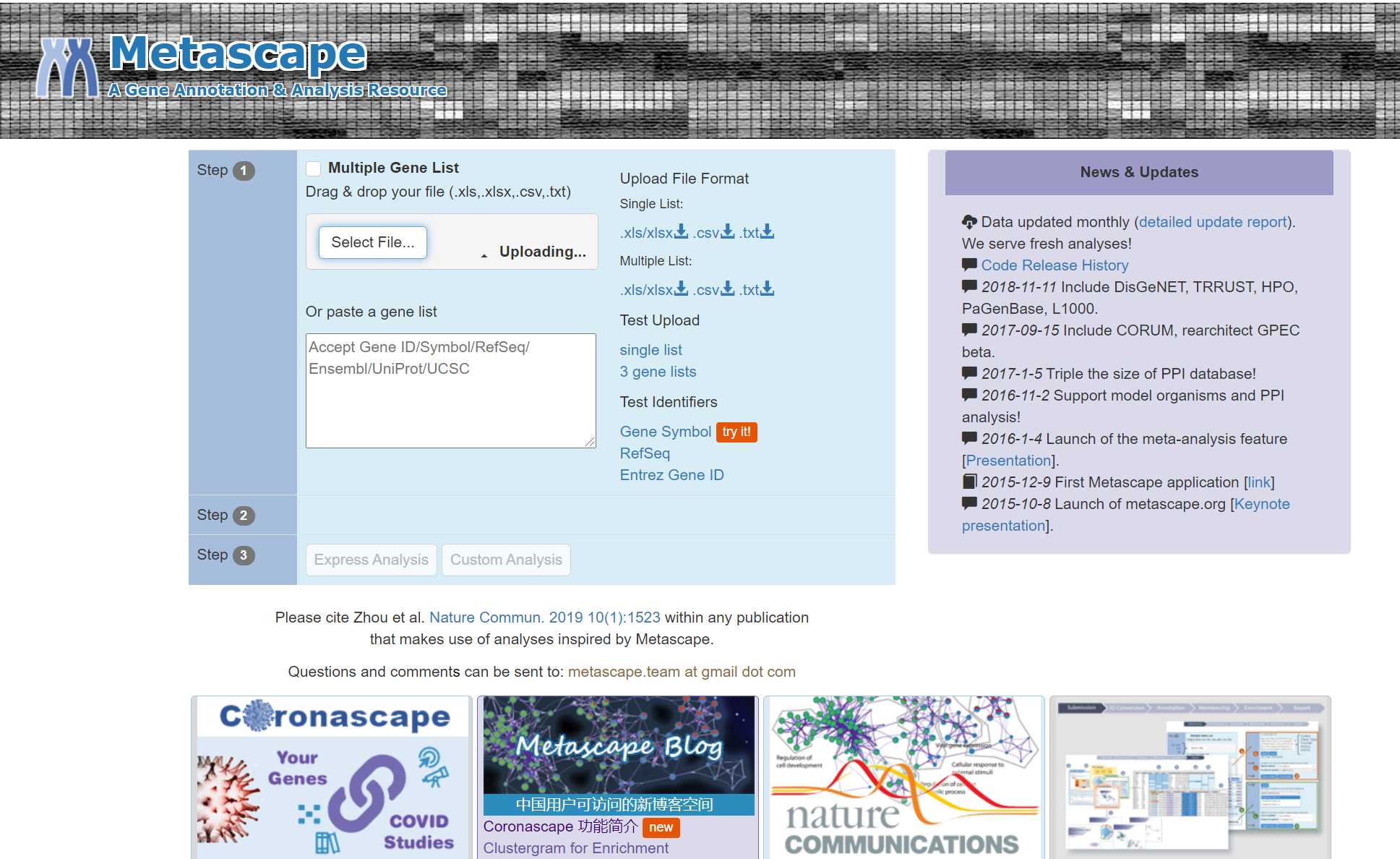
3.2 Metascape
metascape是一个web工具,提供了基因富集分析,蛋白质互作网络分析等多种功能,集成了40多个基因功能注释数据库,并且提供了多样化的可视化方式,目前支持人,小鼠,大鼠等10个物种的基因分析。
对应的文章发表在nature communications上:
Metascape provides a biologist-oriented resource for the analysis of systems-level datasets
Metascape网址:
http://metascape.org/gp/index.html#/main/step1
分析类型:
- ID Conversion: 将基因标识符从流行类型(如Symbol,RefSeq,Ensembl,UniProt,UCSC)转换为人类Entrez基因ID,或反向转换;
- Gene Annotation:从许多功能相关的基因注释中提取,包括蛋白质家族,跨膜/分泌预测,疾病关联,复合关联等。
- Enrichment Analysis:识别丰富的生物学通路,特别是GO,KEGG,Reactome,BioCarta,以及MSigDB中收集的其他通路等。此外,丰富的GO本体术语自动聚类以减少冗余以便于解释。
- Interaction Network Analysis:蛋白质 – 蛋白质相互作用网络基于BioGRID,OmniPath,InWeb_IM构建,并且识别密集组分并进行生物学解释。
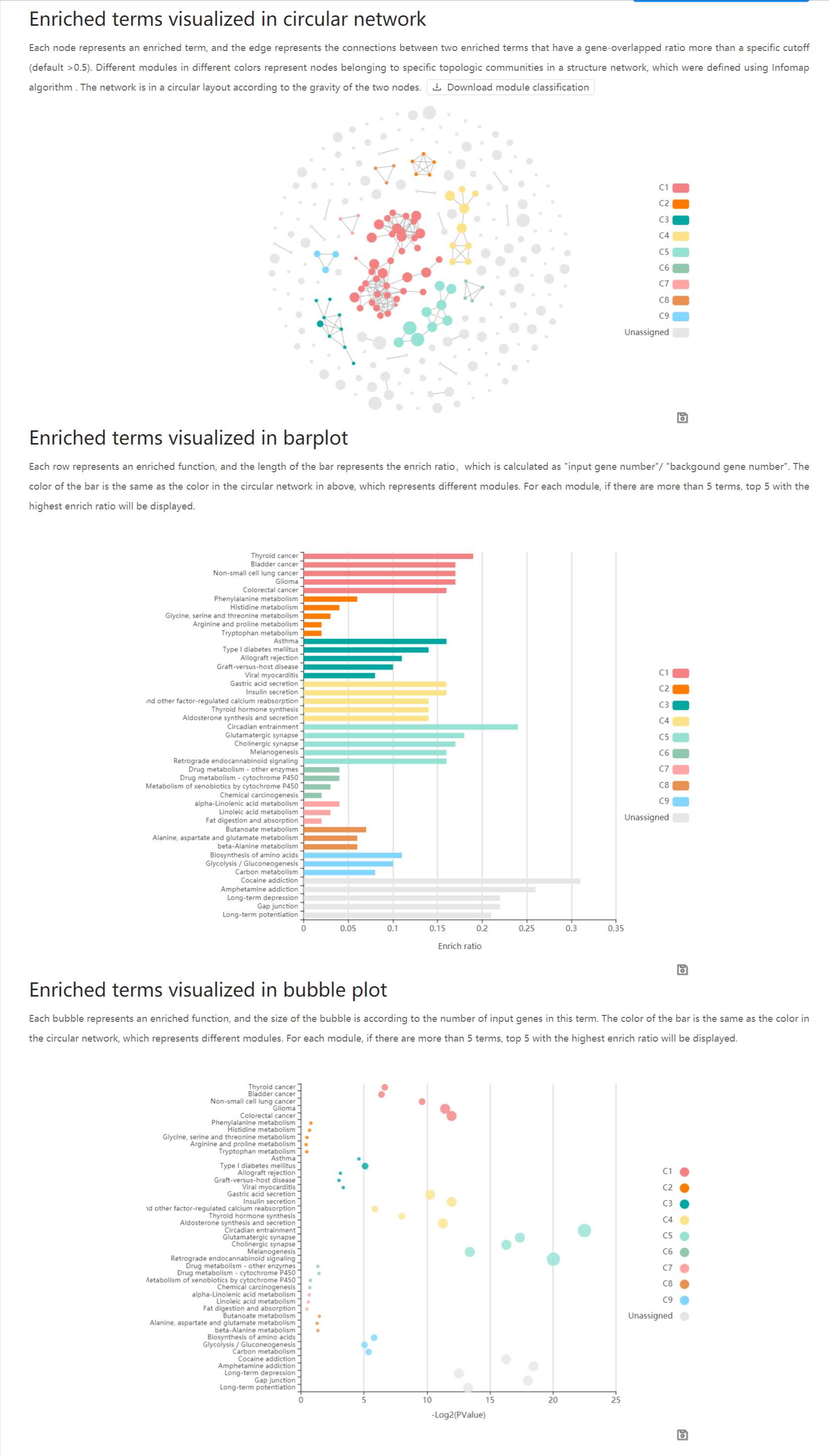
实例分析
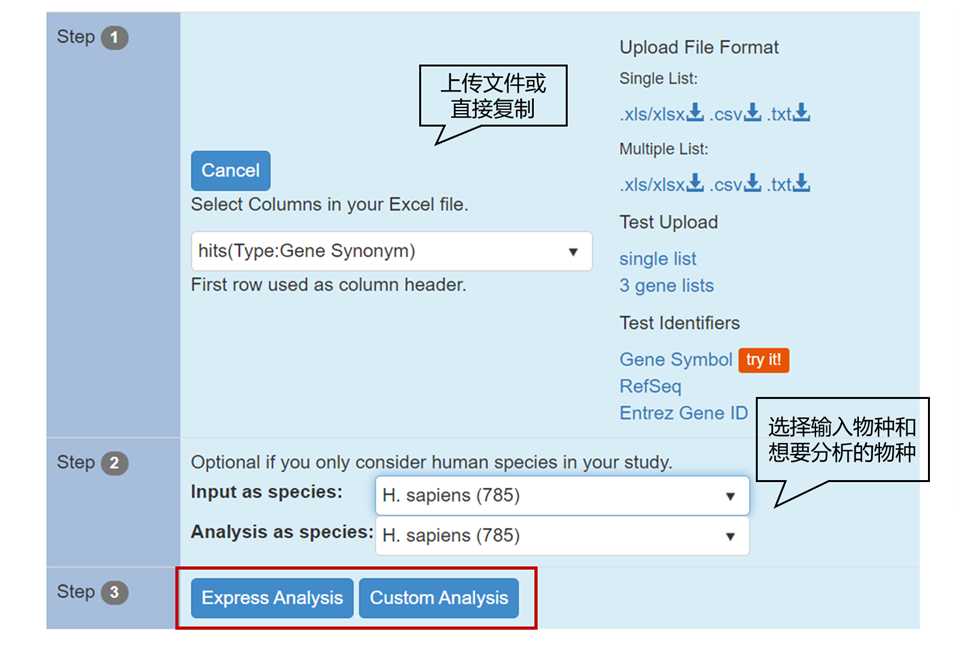
默认的Express Analysis会把许多个数据库的信号通路混一起,出现各种冗余。比如说默认把Reactome、KEGG、Hallmark和GO数据库全部一起展示,但是一般我们科研绘图时会分别展示GO一张图,KEGG一张图。
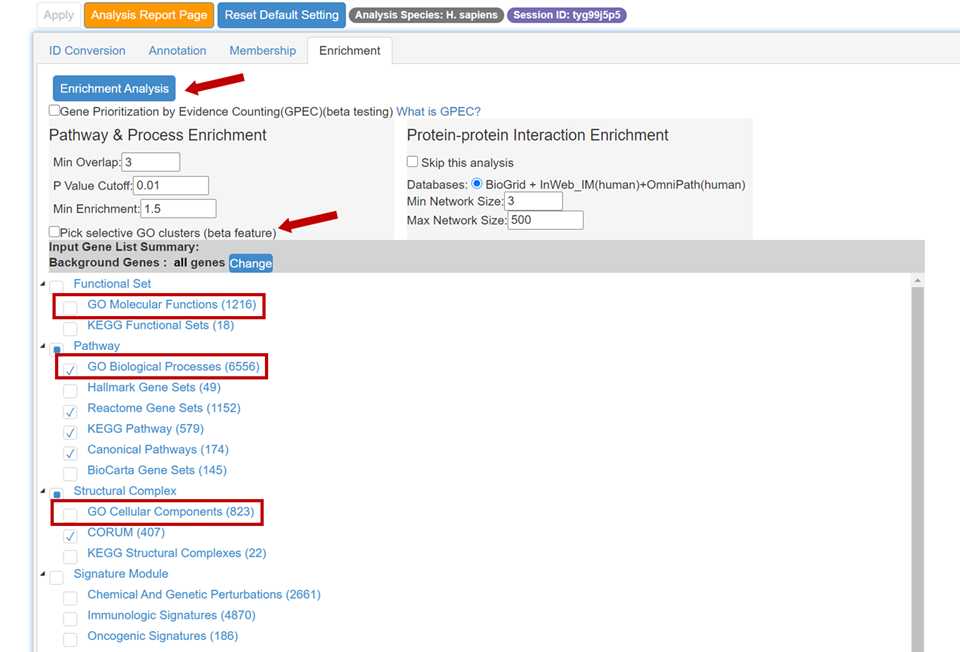
因此我们会在custome Analysis里面的enrichment选项中,从下至上,选择GO相关的数据库,然后勾选pick selective,然后点击enrichment analysis。如下图:
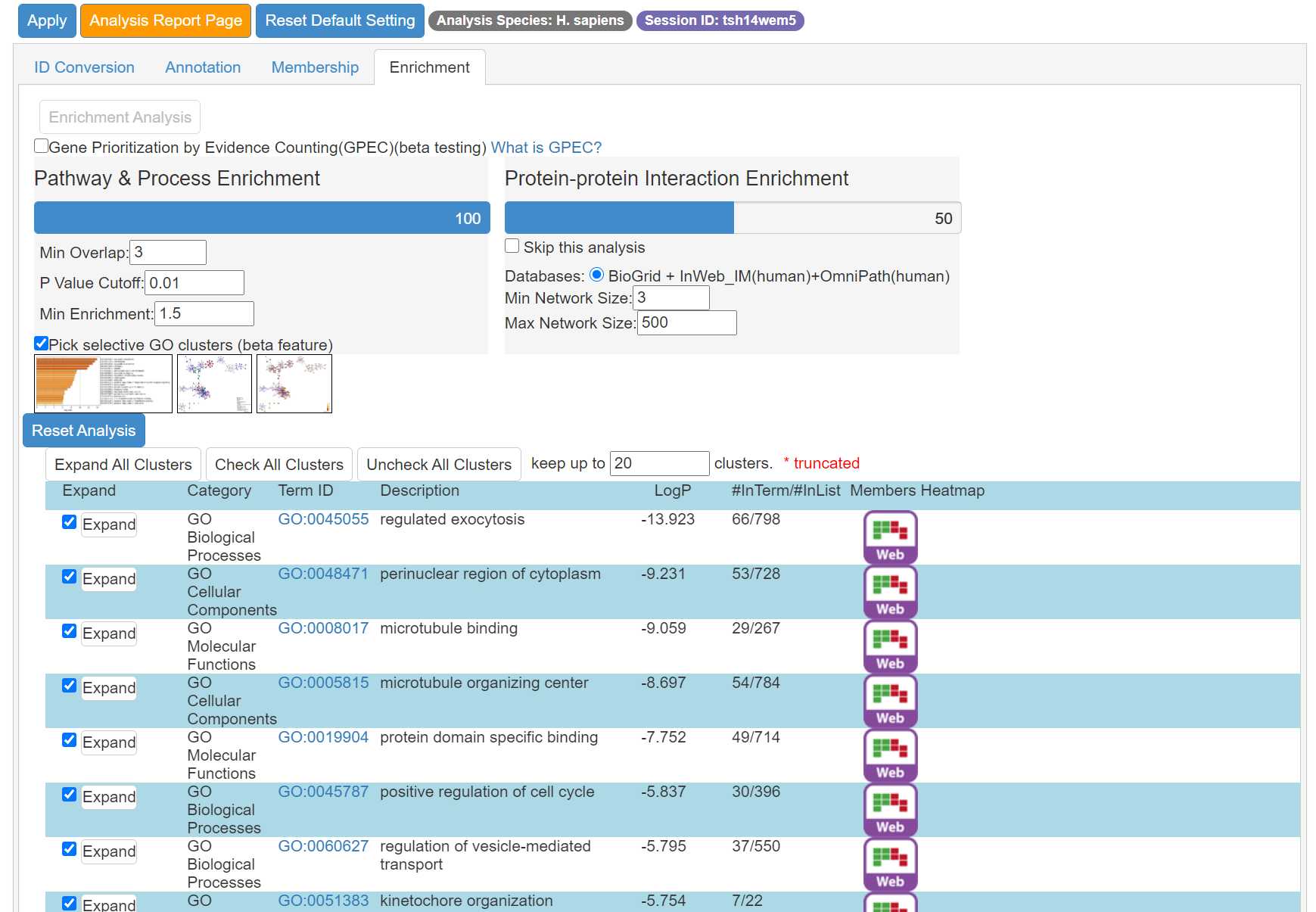
最后结果如下:
注释到的GO:
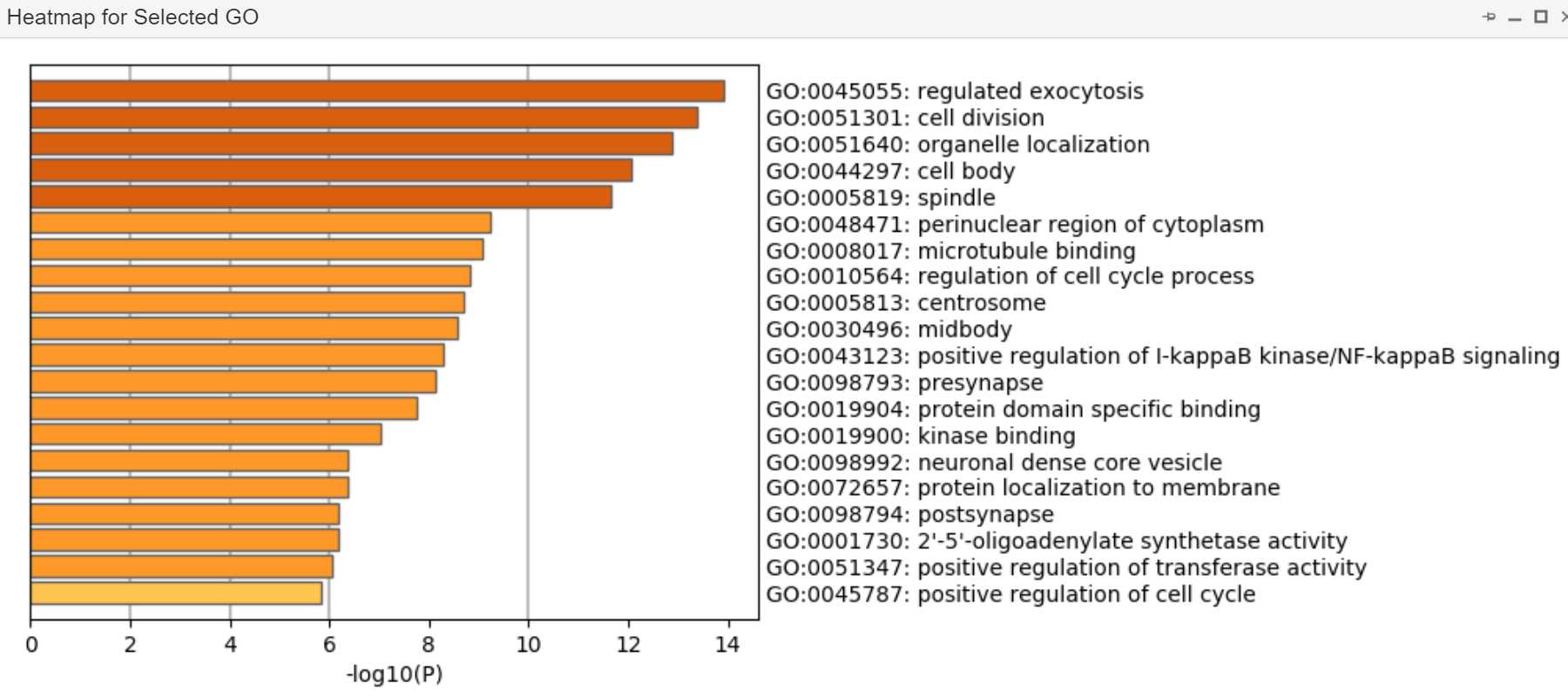
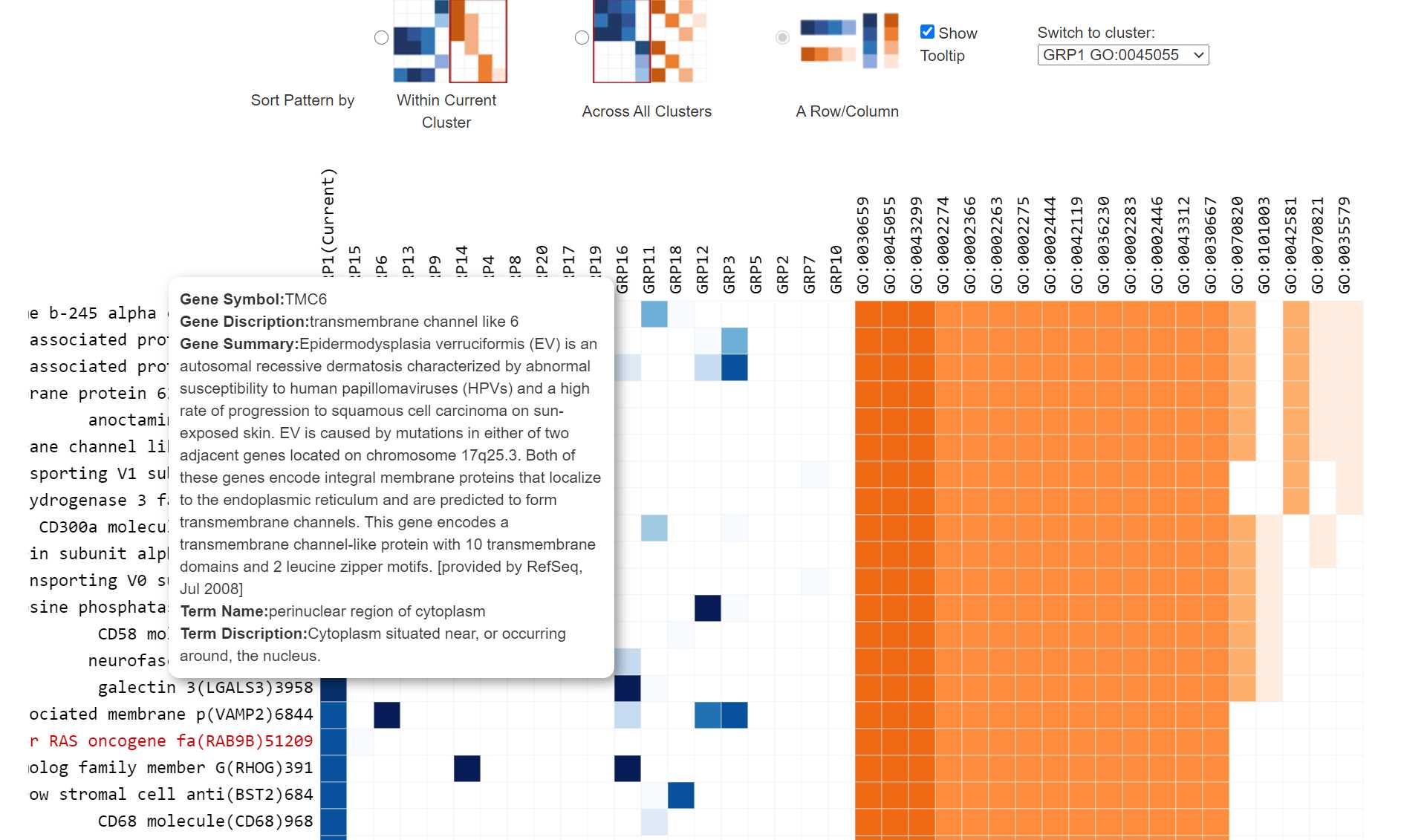
Members Heatmap:
3.3 clusterProfiler
clusterProfiler是大神Y叔写的R包,据说看过了它的绘图就会使人沉迷于其颜值无法自拔。
clusterProfiler中GO的注释信息来自Bioconductor,提供了19个物种的org类型的GO注释信息。(Bioconductor中更多的注释包可参考:bioconductior annotation)
KEGG的注释信息clusterProfiler通过KEGG 数据库的API来获取,https://www.kegg.jp/kegg/rest/keggapi.html
因此对于以上已有pathway注释的物种,只需要将物种简写输入给clusterProfiler, 它会通过联网自动获取该物种的pathway注释信息。
clusterProfiler Reference Manual
3.3.1 ID 转换
bitr():ID转换,ENTREZID是进行GO分析最好的ID类型
bitr(geneID, fromType, toType, OrgDb, drop = TRUE)
geneID:一个含有gene_name的矢量
orgDb:人类的注释包是 org.Hs.eg.db(org.Hs.eg.db包含多种gene_name的类型)
fromType:输入的gene_name的类型
toType:需要转换成的gene_name的类型,可以是多种类型,用大写character类型向量表示
#get the ENTREZID for the next analysis
sig.gene= diff_gene_deseq2 ## 读入分析得到的显著的差异表达基因
head(sig.gene)
gene<-rownames(sig.gene)
head(gene)
## 输入为ENSEMBL号,输出成基因名和ENTREZID
gene.df<-bitr(gene, fromType = "ENSEMBL",
toType = c("SYMBOL","ENTREZID"),
OrgDb = org.Hs.eg.db)
head(gene.df)
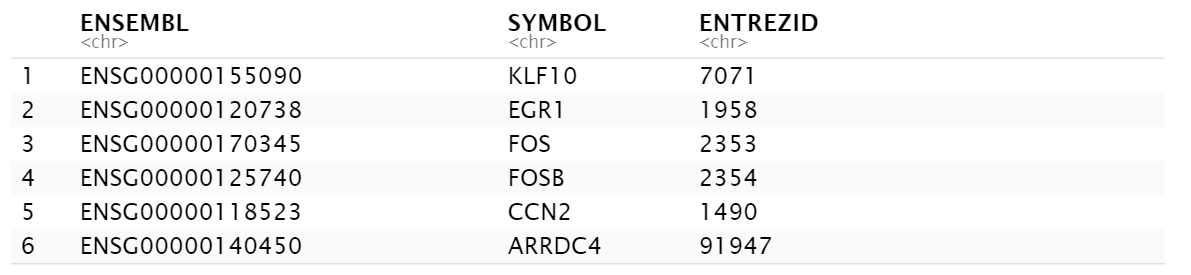
生成的gene.df:
3.3.2 GO enrichment
GO注释分为三大类,分别是:
- 分子生物学功能(Molecular Function,MF)
- 生物学过程(Biological Process,BP)
- 细胞学组分(Cellular Components,CC)
通过这三个功能大类,对一个基因的功能进行多方面的限定和描述。
MF:
#Go enrichment
ego_mf<-enrichGO(gene = gene.df$SYMBOL,
OrgDb = org.Hs.eg.db,
keyType = 'SYMBOL',
ont = "MF",
pAdjustMethod = "BH",
pvalueCutoff = 0.05,
qvalueCutoff = 0.05)
head(ego_mf)
## 绘图
barplot(ego_mf,showCategory = 18,title="The GO_MF enrichment analysis of all DEGs ")+
scale_size(range=c(2, 12))+
scale_x_discrete(labels=function(ego_bp) str_wrap(ego_bp,width = 25))
CC:
#Go enrichment
ego_cc<-enrichGO(gene = gene.df$ENSEMBL,
OrgDb = org.Hs.eg.db,
keyType = 'ENSEMBL',
ont = "CC",
pAdjustMethod = "BH",
pvalueCutoff = 0.01,
qvalueCutoff = 0.05)
head(ego_cc)
## 绘图
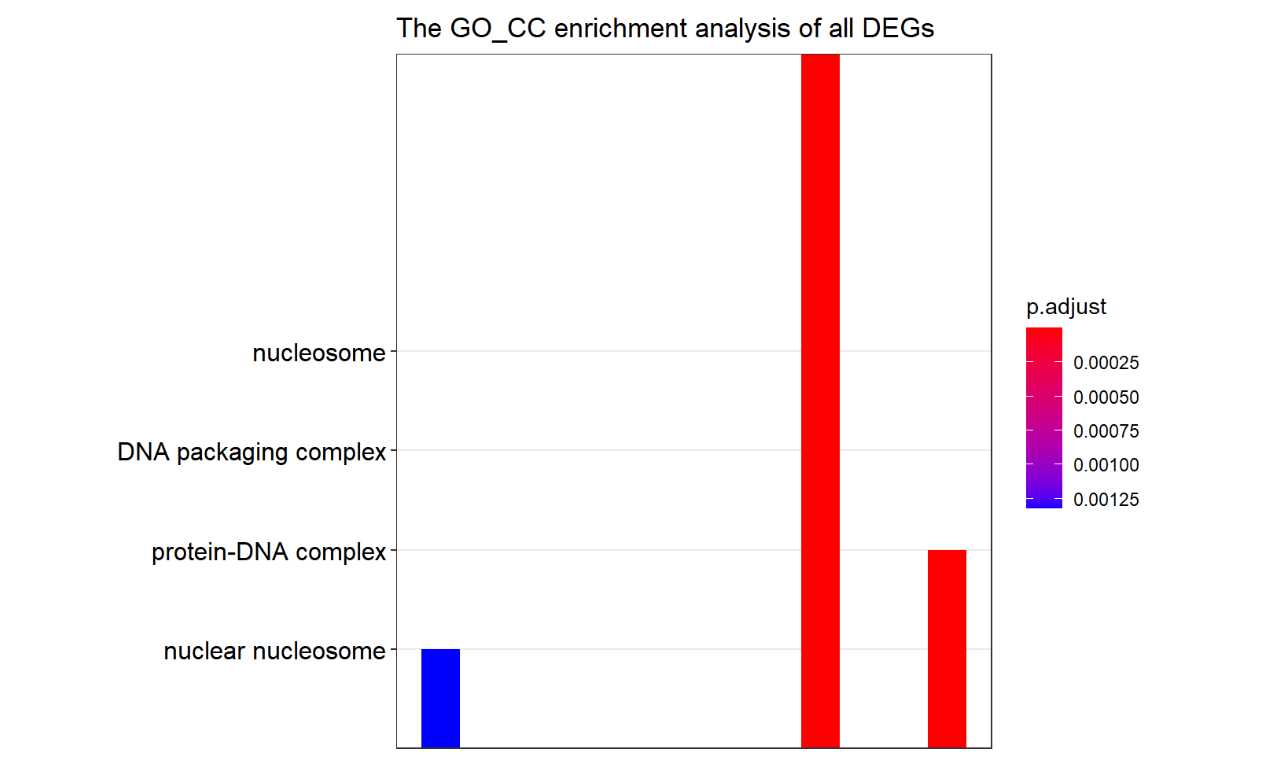
barplot(ego_cc,showCategory = 18,title="The GO_CC enrichment analysis of all DEGs ")+
scale_size(range=c(2, 12))+
scale_x_discrete(labels=function(ego_bp) str_wrap(ego_bp,width = 25))
BP(这里用SYMBOL表示)
ego_bp<-enrichGO(gene = gene.df$SYMBOL,
OrgDb = org.Hs.eg.db,
keyType = 'SYMBOL',
ont = "BP",
pAdjustMethod = "BH",
pvalueCutoff = 0.01,
qvalueCutoff = 0.05)
head(ego_bp)
## 绘图
barplot(ego_bp,showCategory = 18,title="The GO_BP enrichment analysis of all DEGs ")+
scale_size(range=c(2, 12))+
scale_x_discrete(labels=function(ego_bp) str_wrap(ego_bp,width = 25))
3.3.3 KEGG enrichment
# kk<-enrichKEGG(gene =gene.df$ENTREZID,
# organism = 'hsa',
# pvalueCutoff = 0.05)
# 不知道为啥在Rmd里调用会出错,脚本里明明好好的
# 错误说:species = ”hsa“不在官方给出的物种列表里,太奇怪了!!
# 这里直接调用RData
load("enrichKEGG-kk.RData")
kk[1:30]
#绘图
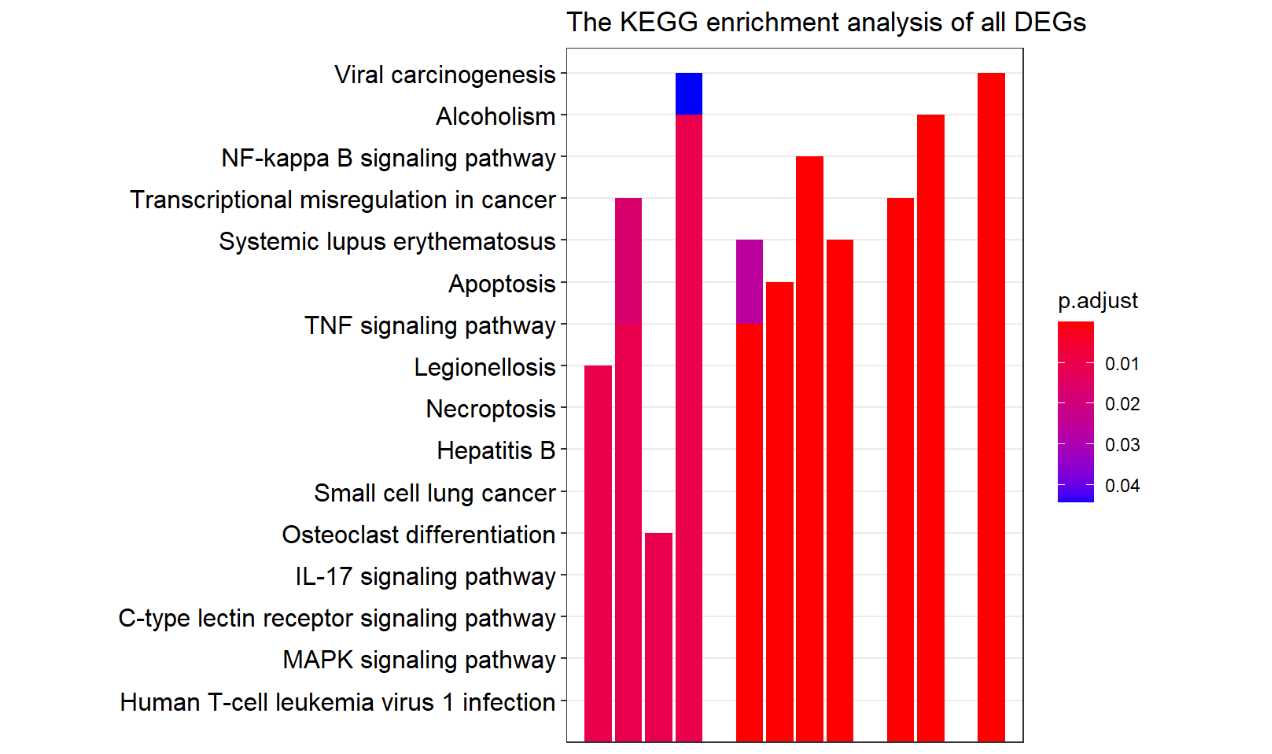
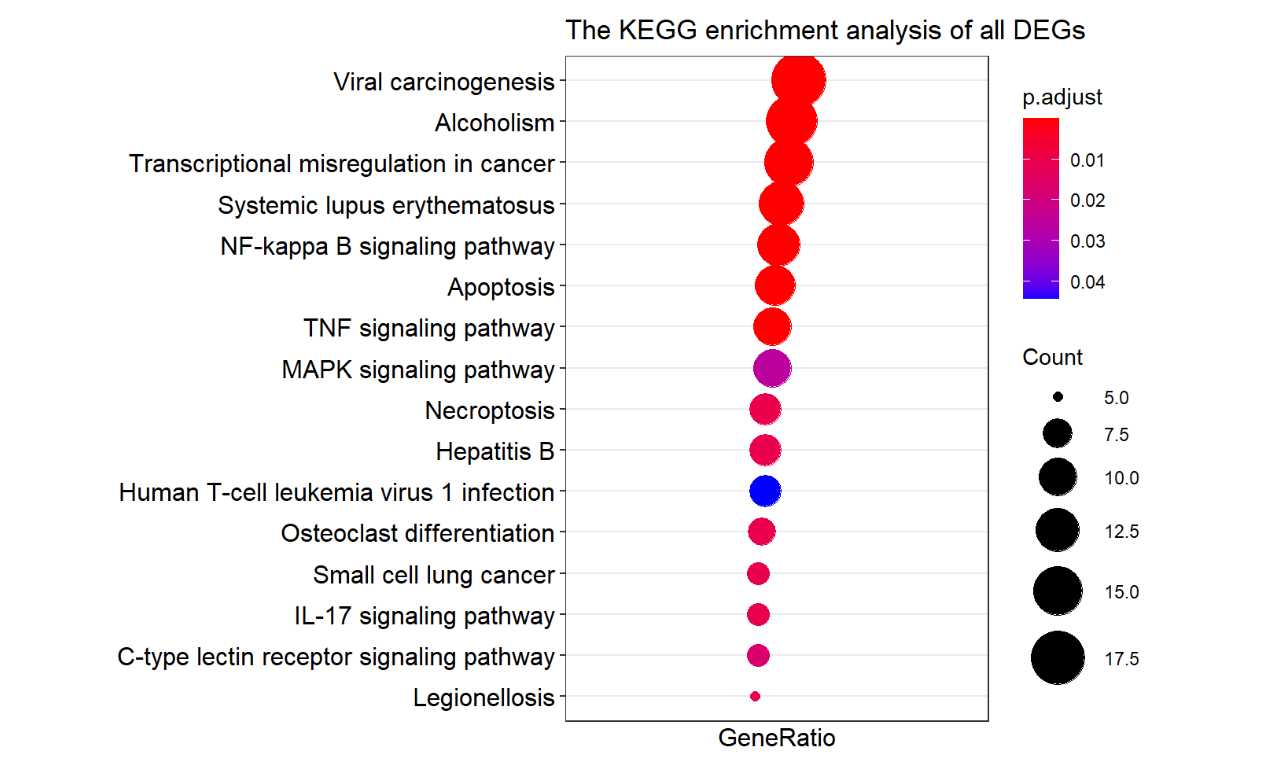
mykegg<-barplot(kk,showCategory = 20, title="The KEGG enrichment analysis of all DEGs")+
scale_size(range=c(2, 12))+
scale_x_discrete(labels=function(kk) str_wrap(kk,width = 25))
mykegg
dotplot(kk,showCategory = 20, title="The KEGG enrichment analysis of all DEGs")+
scale_size(range=c(2, 12))+
scale_x_discrete(labels=function(kk) str_wrap(kk,width = 25))
3.4 KOBAS
这个是我在公众号上看到的一个在线工具,由北大魏文丽课题组开发,主要功能是用于基因/蛋白质功能注释与功能富集,注释功能相当强大,包括疾病的查询、IncRNA的鉴定、注释、富集,GSEA,GSA,PADOG等功能。
KOBAS(KEGG Orthology Based Annotation System)
网址:http://kobas.cbi.pku.edu.cn/kobas3/?t=1
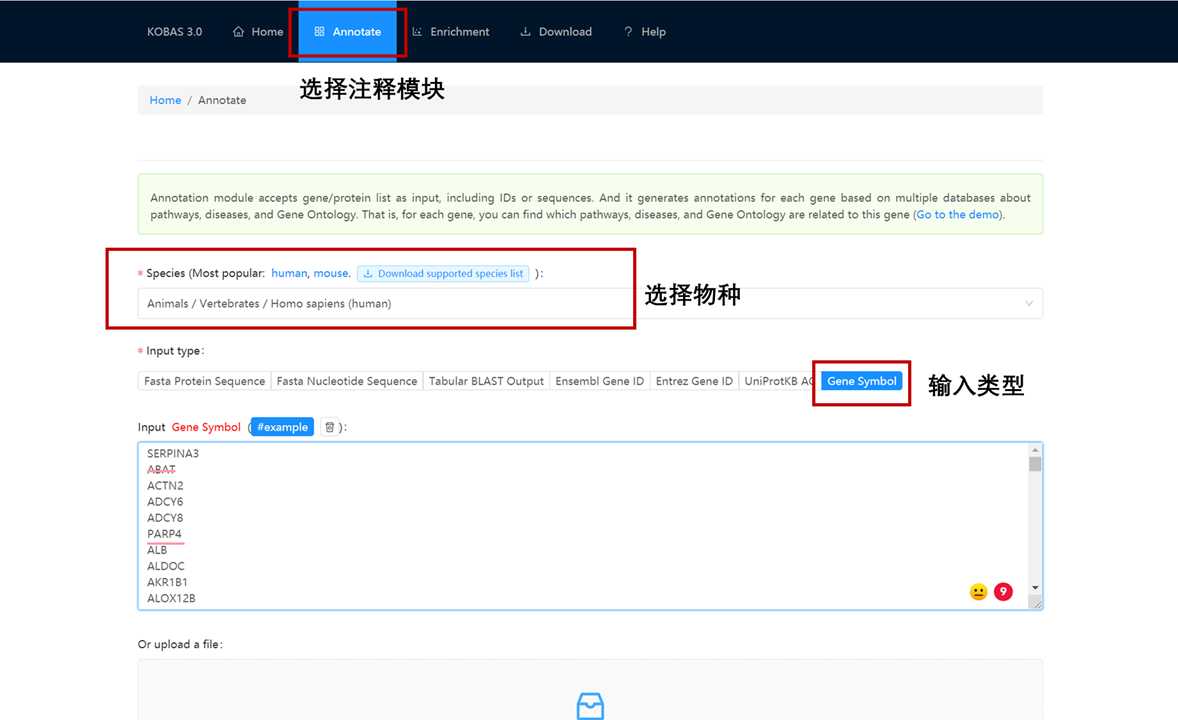
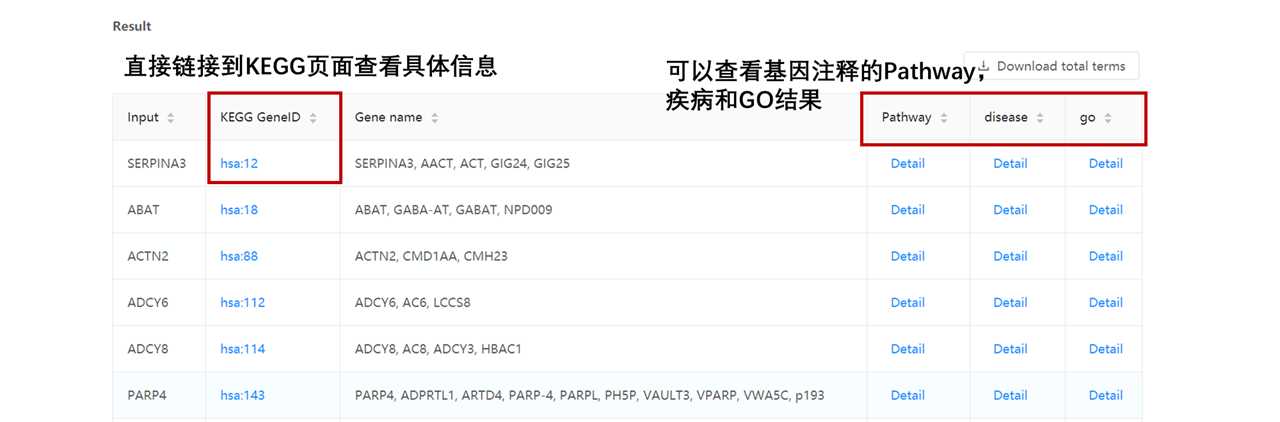
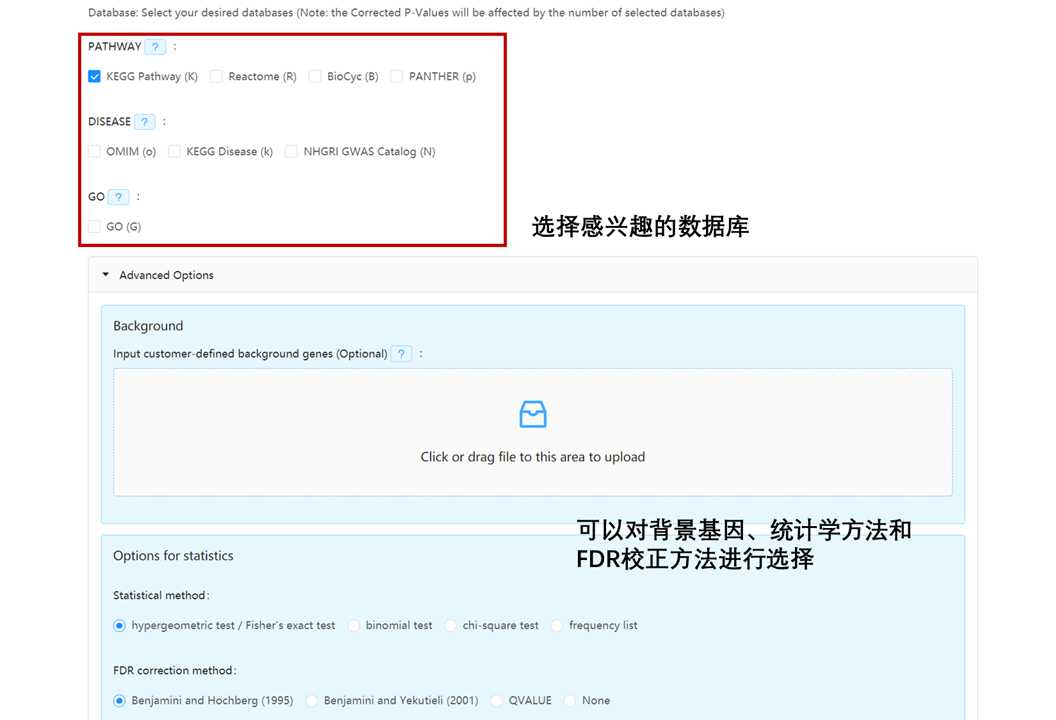
基因注释:
富集分析:
可视化的配色好看!!!我太爱了!!
3.5 GOEAST
中科院遗传所王秀杰老师课题组2008年发表的在线工具GOEAST
访问网站:http://omicslab.genetics.ac.cn/GOEAST/index.php
GOEAST自发表以来一直在更新维护,并且每周与Gene Ontology网站同步GO注释数据库,保证分析结果的时效性和准确性。文章从发表至今,被引用504次,总访问量近140万次,总使用量近87万次。可以输入邮箱上传后等着去邮箱查收结果就好了。
这款工具最初是为芯片研究开发的,有一些芯片分析特有的功能。同时GOEAST还支持多个GO富集分析结果的比较和自定义背景数据集。
官方教程:GOEAST-Tutorial
四、参考
生信富集分析
别搜啦!关于富集分析你想知道的这里都有!
零代码功能富集分析(Metascape数据库数据库使用教程)
使用clusterProfiler进行富集分析
工具篇丨GO和KEGG富集不到通路?快试试这个超赞的功能分析工具吧
今天的文章【Bioinfo Blog 011】【R Code 008】——功能富集分析分享到此就结束了,感谢您的阅读,如果确实帮到您,您可以动动手指转发给其他人。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/24764.html