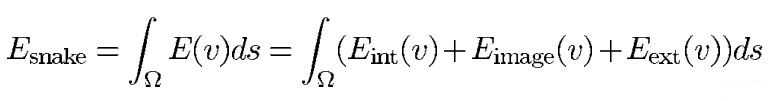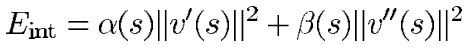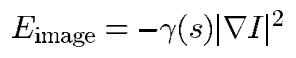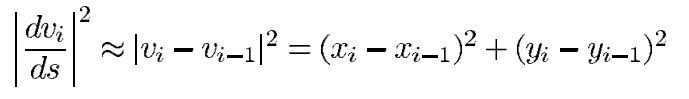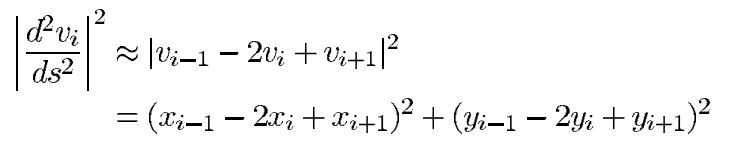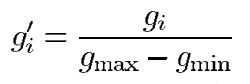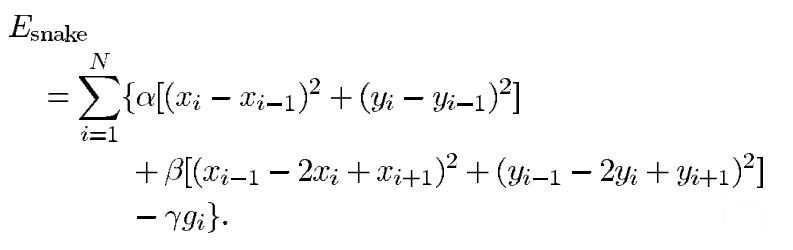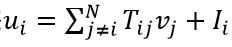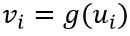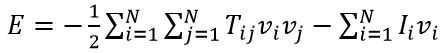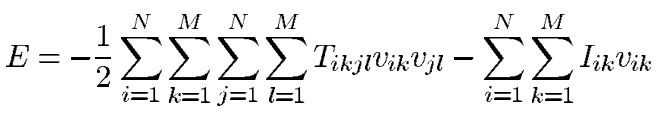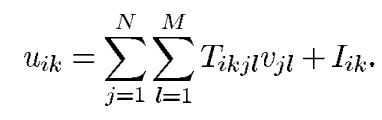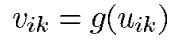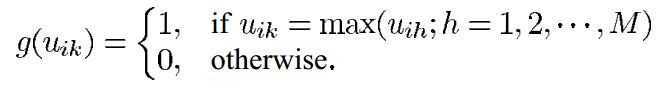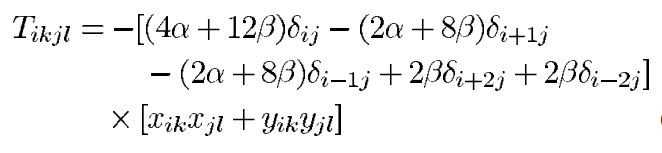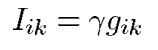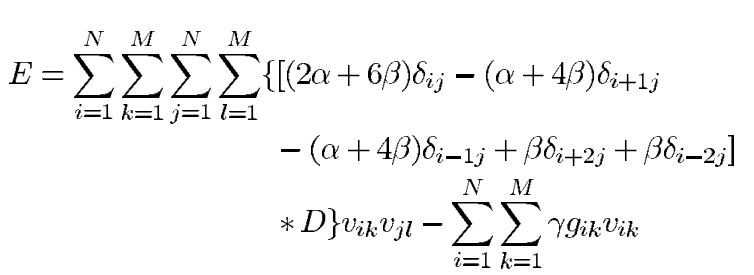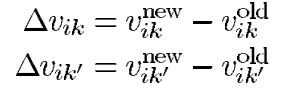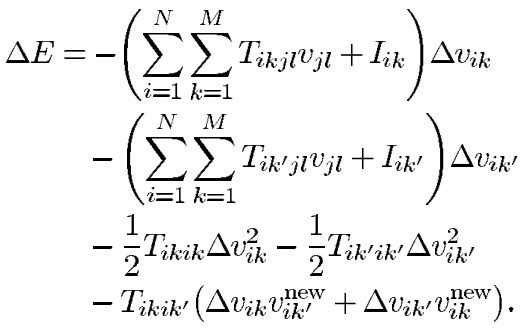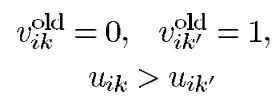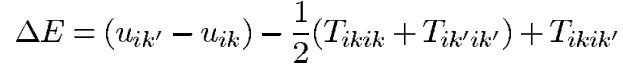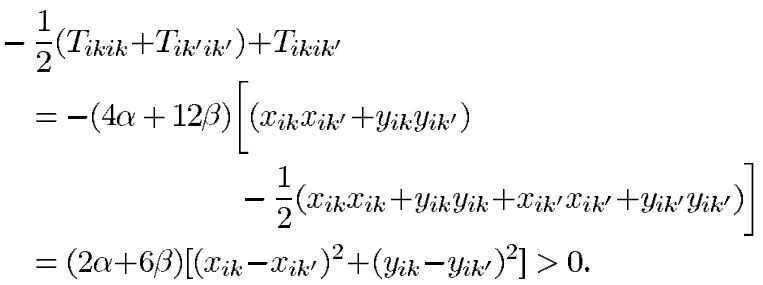Computerized Tumor Boundary Detection Using a Hopfield Neural Network这篇论文给出了结合的思想
此方法在论文中被用于提取人脑多层MRI图像上每个切片中的脑肿瘤的边界。首先使用低通线性滤波器对每个原始MRI数据切片进行预处理以增强图像。然后根据形态学过程得到的轮廓为初始边界,设置初始搜索网格。最后一步是使用Hopfield神经网络检测肿瘤边界。
1、 预处理
在使用Hopfield神经网络提取轮廓之前,必须估计初始轮廓。由于整个大脑是一个连接实体,因此我们假设一个切片中的肿瘤形状与其邻近切片中的肿瘤形状非常相似。基于这个假设,当前切片中检测到的轮廓被选为下一切片的初始轮廓估计。 然后,初始化问题就是在第一个分析切片中找到初始轮廓。
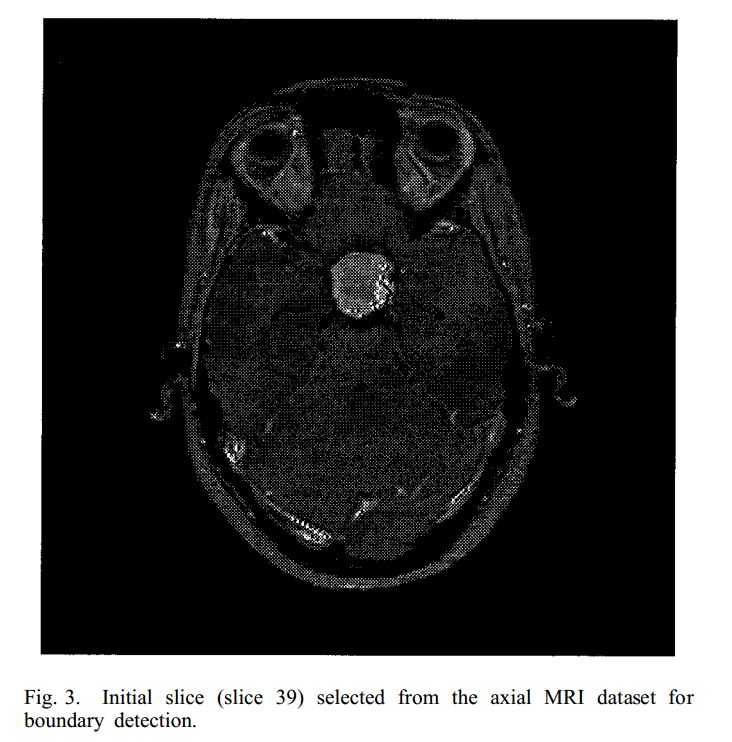
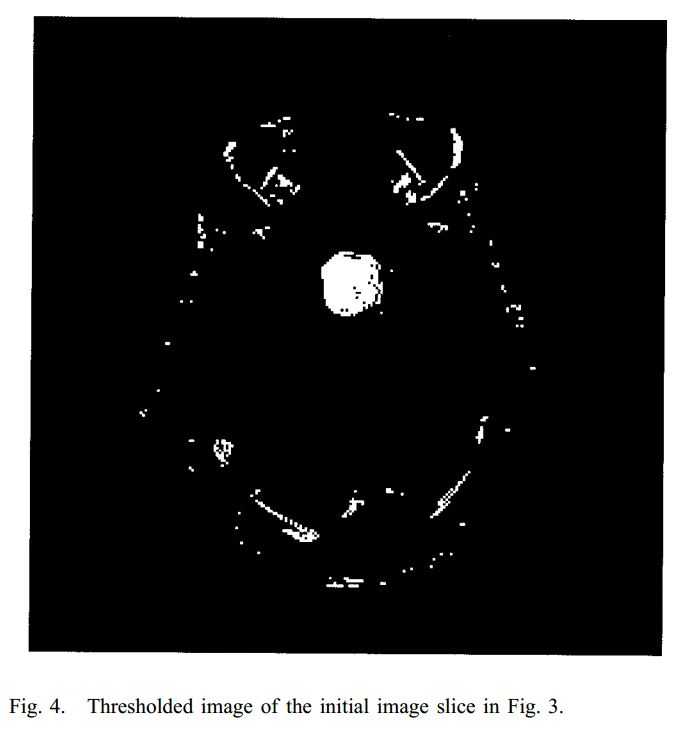
为了实现快速和准确的计算,我们应该选择具有最佳强度对比度的切片作为第一个切片。我们首先对灰度图像进行阈值提取肿瘤。假设脑瘤比其周围组织具有更高的灰度值,阈值被设置为高于白质的水平。 实际上,阈值对于边界检测并不重要,因为在这个阶段只需要一个粗略的边界。
预处理包括复试和膨胀算法,以消除图像噪声同时保留脑肿瘤区域。
1、 轮廓初始化:
(1)给初始轮廓放置N个采样点;
(2)在每个采样点上沿着垂直于轮廓方向设置一条搜索线;
(3)在每条搜索线上放置M个采样点。
3、将snake模型的能量函数离散化:
(1)原始snake模型能量函数形式:
其中
int、image、ext分别代表了内部约束力、图像力和外部约束力。内部里代表了由于弯曲和不连续而产生的内部力。因为我们想让轮廓被吸引在边缘点上,所以图像力依赖于梯度。
将snake模型能量函数离散化:
(2)能量函数离散化:
上式中的
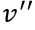
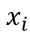
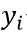
其中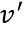
由于MR图是灰度图,所以对采样点进行灰度值均一化。
其中,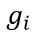
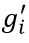
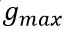
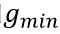
最终,离散化后的能量函数是如下形式:
4、 离散型Hopfield神经网络能量函数:
神经元状态
神经元输出
能量函数:
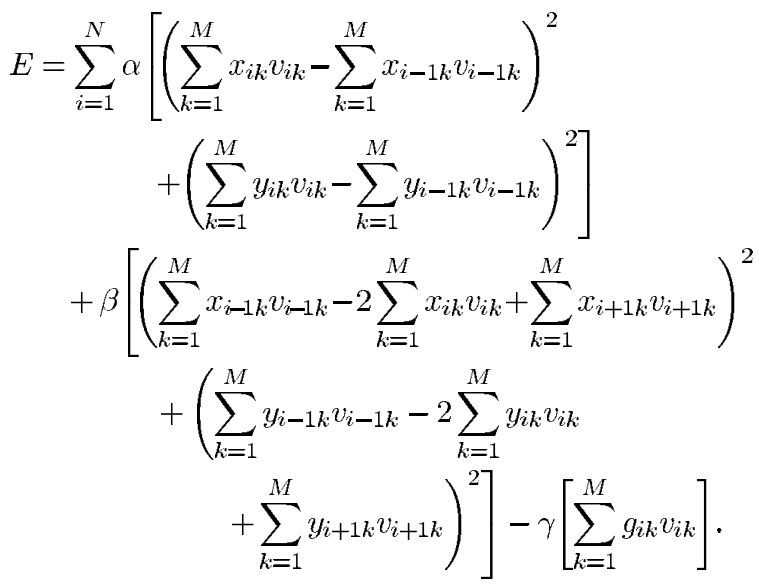
4、 构建snake_hopfield能量函数:
(1) 将图像上的采样点和Hopfield神经网络的结构对应起来:
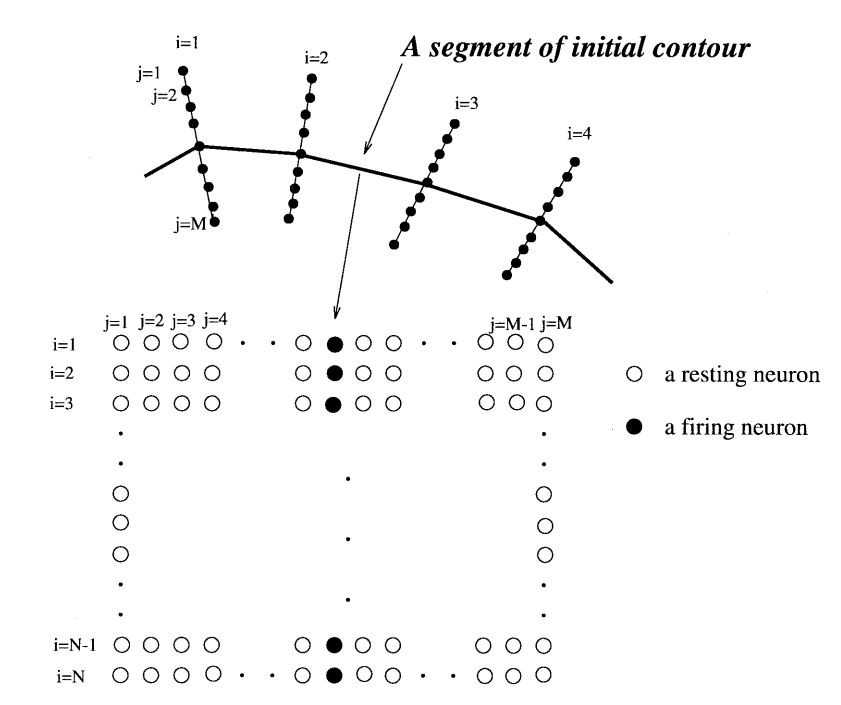
如图所示,上图表示的是真实MR图中的轮廓,i代表的是轮廓上的采样点,j代表的是搜索线上的采样点,初始轮廓便是一条i=1,2,…,N同时j都相同的一系列采样点。我们发现这样的结构正好可以对应在离散Hopfield神经网络上的神经元。下图表示的便是神经网络的结构,让每个采样点对应到神经网络中的神经元。初始轮廓便是列中激活的神经元,搜索线上的其他采样点对应的就是处于抑制状态的神经元。通过能量函数最小化使得每行中神经元的状态发生改变,最终神经元的状态就是轮廓的演化结果。
(2) 将snake模型的能量函数与Hopfield神经网络能量函数对应:
用上式表示对应后的Hopfield神经网络的能量函数。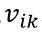
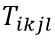
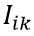
i行k列神经元的状态更新用下式表示:
相对应的输出为:
其中
最终得到的snake_hopfield能量函数便是:
将上式与Hopfield神经网络做对比,通过附录给出的方法,我们可以得到神经元之间权重和神经元偏置的表达式:
最终通过异步或同步更新方式(但是一般异步方式更好)来更新神经元状态。通过权重表达式可以看出权重矩阵对角线上的元素不为0,所以神经元存在自反馈,自反馈可能会造成能量函数有一个过渡,用确定性规则可以确保神经网络收敛,有效方法在论文附录中。
神经网络能量下降是通过迭代过程中的最大值确定性决策规则决定的:由于网络中每次更新,每行都只能有一个神经元被激活,所以每行至多有两个神经元的状态会发生翻转,因此将被激活和被抑制的神经元分别用k和k’表示,所以神经元的状态和能量函数变化由下式表示:
根据最大值确定性决策规则可得,当:
可得:
所以能量函数的变化可以简化为下式:
我们可以发现上式第一项总为负,所以第二项:
大于第一项时,能量函数变化量大于0。
为确保能量收敛,只有当流量函数变化小于0时,才会接受神经元的新状态。
实验结果:
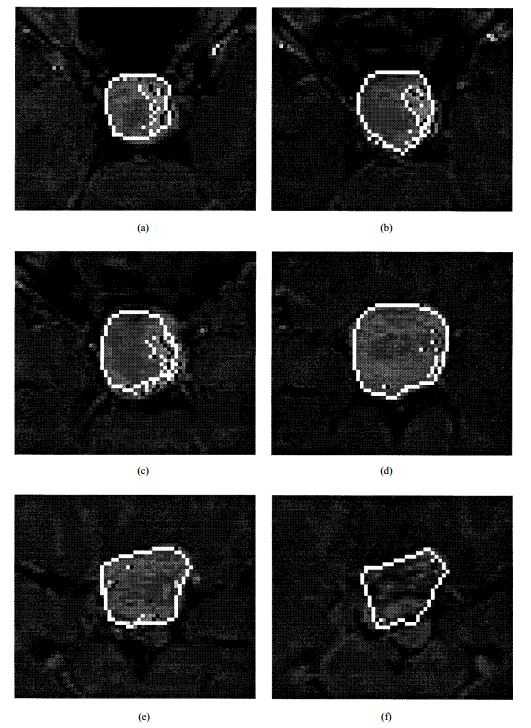
今天的文章如何将snake模型与Hopfield神经网络结合用于提取边缘分享到此就结束了,感谢您的阅读,如果确实帮到您,您可以动动手指转发给其他人。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/27711.html