工具:R语言biomaRt包
生物学背景:同源,最基本的意义就是具有共同祖先。当然这里不是说人和鼠有相同的祖先,我们这里的分子水平的同源性。从分子水平讲则是指两个核酸分子的核苷酸序列或两个蛋白质分子的氨基酸序列间的相似程度,那我们就说二者同源,就是说可以相互转化的基因具有一定的序列相似性,那么二者的基因便可以成为同源基因。许多不同的物种间都具有同源性。现代分子生物学中的同源性描述的是基因与基因之间相似关系,它表明的是两个相比较的序列之间的匹配程度。一般来说,如果两条基因序列相似性达80% ,就可以把它们称为“同源基因(homologousgene)”。
- 安装biomaRt包
install.packages("BiocManager")
BiocManager::install("biomaRt")
- 选择目标数据库和数据集(人和小鼠)
library(biomaRt)
human=useMart("ensembl",dataset="hsapiens_gene_ensembl")
class(human)
mouse=useMart("ensembl",dataset="mmusculus_gene_ensembl")
class(mouse)
#useMart一般跟两个参数,第一个借助ensemble数据库,第二个参数是选择的物种数据集
- 构造测试基因集
genes=c("Tmx2","Trp53","Zfp286")
- 小鼠基因同源映射到人
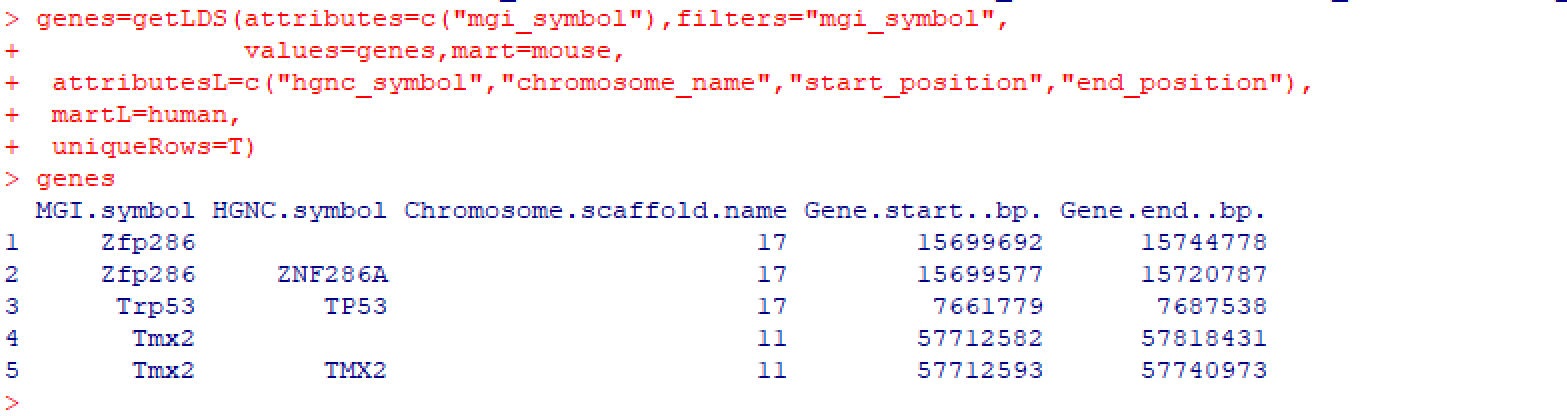
genes=getLDS(attributes=c("mgi_symbol"),filters="mgi_symbol",
values=genes,mart=mouse,
attributesL=c("hgnc_symbol","chromosome_name","start_position","end_position"),
martL=human,
uniqueRows=T)
#attributes指所要检索数据集的属性参数,可用listAttributes函数检索属性列表
#values指构造的数据集(即输入的数据集)
#mart指输入数据的对象(如mouse)
#attributesL指需要转化的目标数据集
#martL指输出的对象(如human)
结果如下
- 写一个简单的封装函数并测试
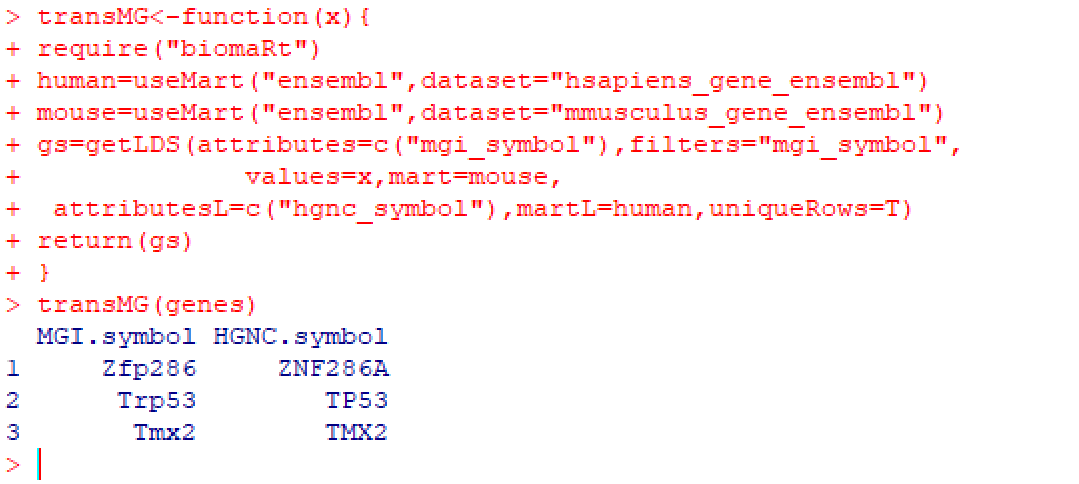
transMG<-function(x){
require("biomaRt")
human=useMart("ensembl",dataset="hsapiens_gene_ensembl")
mouse=useMart("ensembl",dataset="mmusculus_gene_ensembl")
gs=getLDS(attributes=c("mgi_symbol"),filters="mgi_symbol",
values=x,mart=mouse,
attributesL=c("hgnc_symbol"),martL=human,uniqueRows=T)
return(gs)
}
transMG(genes)
结果如下
- 人转小鼠基因同理
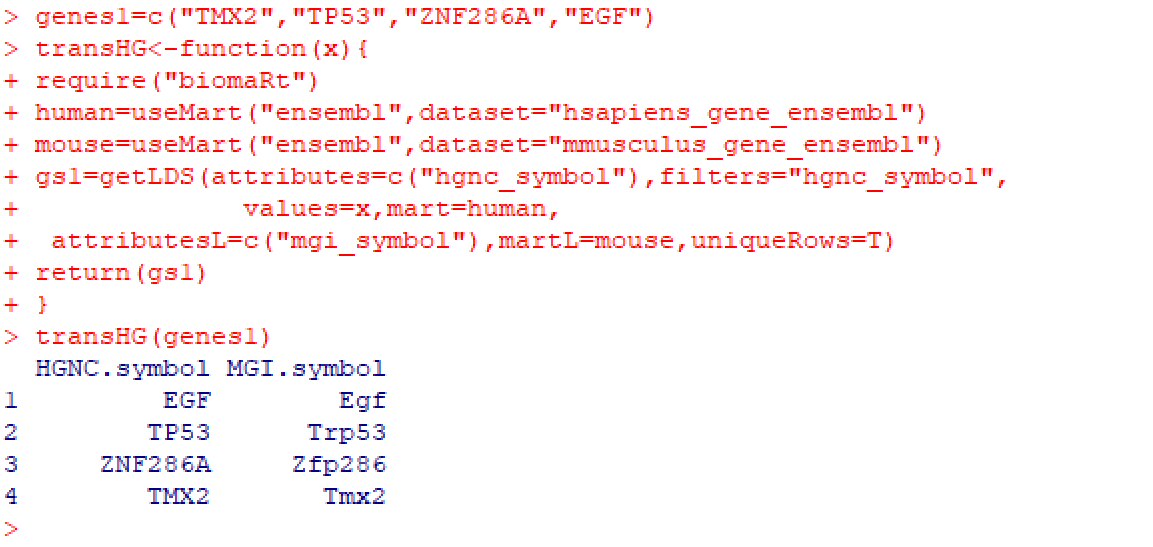
genes1=c("TMX2","TP53","ZNF286A","EGF")
transHG<-function(x){
require("biomaRt")
human=useMart("ensembl",dataset="hsapiens_gene_ensembl")
mouse=useMart("ensembl",dataset="mmusculus_gene_ensembl")
gs1=getLDS(attributes=c("hgnc_symbol"),filters="hgnc_symbol",
values=x,mart=human,
attributesL=c("mgi_symbol"),martL=mouse,uniqueRows=T)
return(gs1)
}
transHG(genes1)
结果如下
以上,就是跨物种基因同源转换的方法之一,biomaRt包用起来真的很方便,不过在转换的时候可能会出现一对多的现象。
参考:https://www.jianshu.com/p/96039fc8f4eb
今天的文章人鼠基因同源转换分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/61238.html