目录
1. 基因表达式编程 GEP Python 代码实现【★★★★★】
今天将用python实现GEP算法中,染色体选择,复制,变异等的内容
【PS:注意结合前边内容,今天的内容比较多,包括染色体选择、复制、变异等内容,需要对GEP的基本知识内容认识。】
基因表达式编程(GEP)自学 第【1】天 Python 实现
基因表达式编程(GEP)自学 第【2】天 Python 实现
基因表达式编程(GEP)自学 第【3】天 Python 实现
基因表达式编程(GEP)自学 第【4】天 Python 实现(代码)
基因表达式编程(GEP)自学 第【5】天 Python 实现(代码)
2. 轮盘赌算法 实现染色体的选择
2.1. 基本思想:
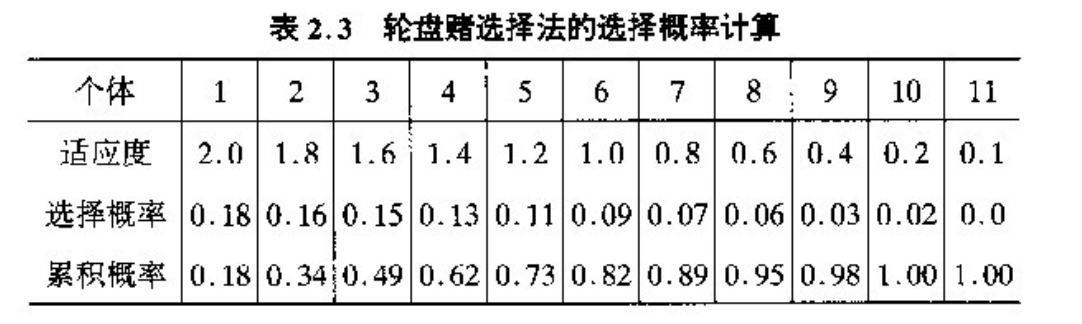
个体被选中的概率与其适应度函数值成正比
设群体大小为n,个体i的适应度为Fi,则个体i被选中遗传到下一代群体的概率为
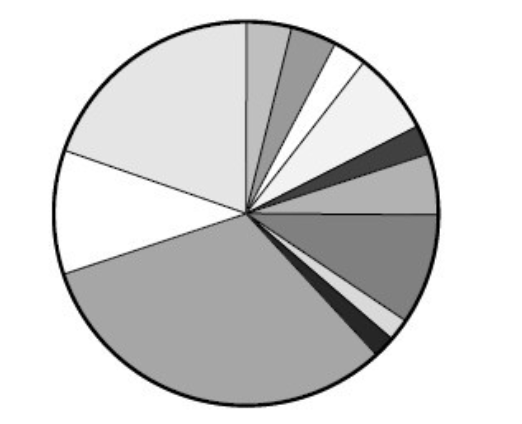
设想群体全部个体的适当性分数由一张饼图来代表 ,如下图:
群体中每一染色体指定饼图中一个小块。块的大小与染色体的适应性分数成比例,适应性分数愈高,它在饼图中对应的小块所占面积也愈大。为了选取一个染色体,要做的就是旋转这个轮子,直到轮盘停止时,看指针停止在哪一块上,就选中与它对应的那个染色体。
2.2. 代码实现:
Environment 类:
def select(self, sum_fitness,NowBestChromosome): ''' :param sum_fitness: 总的适应度 :param NowBestChromosome: 用于保存当代最佳染色体 :return: selected 用于保存赌盘选择出的染色体 ''' accumulator = 0 # 设置累加器 roulette=[] # 设置轮盘 selected=[] #制作赌盘,如果percentage 区间为0的 for i in range(len(self.population)): percentage = self.population[i][1] / sum_fitness * 10 if percentage != 0: roulette.append(percentage + accumulator) accumulator = accumulator + percentage else: roulette.append(0) #一共选择 len(self.population)-1 条染色体体,最后一条保存最佳染色体。 for i in range(len(self.population)-1): #产生随机数 ranNumber=random.uniform(0,10) #进行选择染色体 for i_pos in range(len(self.population)): if ranNumber <= roulette[i_pos]: selected.append(self.population[i_pos][0]) break #最后一条保存最佳染色体 selected.append(NowBestChromosome) return selected 3. 染色体 复制更新
PS: 将赌盘后选择种群染色体,进行染色体复制,保证更新在 self.population
Environment 类:
def replicate(self, chromosomes): ''' 进行染色体复制,保证更新在 self.population :param chromosomes: 赌盘后选择种群染色体 :return: ''' self.population = [] for chromosome in chromosomes: self.population.append(chromosome.replicate()) Chromosome 类:
def replicate(self): ''' 基因复制 newGenes 保存新的基因 newHomeotics 保存新的连接基因 newChromosome 创建新的染色体,并赋值 :return: newChromosome 返回新的染色体 ''' newGenes = [] newHomeotics = [] # 基因复制 for gene in self.genes: newGenes.append(gene.replicate()) # 连接基因复制 for homeotic in self.homeotics: newHomeotics.append(homeotic.replicate()) # 创建新的染色体 newChromosome = Chromosome() newChromosome.genes = newGenes newChromosome.homeotics = newHomeotics return newChromosome Gene 类:
def replicate(self): ''' 创建新的基因,并对基因内的元素进行复制 :return: newGene 返回新的基因 ''' newGene = Gene(self.genome, self.homeotic_flag) newGene.head = self.head[:] newGene.tail = self.tail[:] return newGene 4. 染色体修饰
包括:
- mutation: 突变
- ISTransposition IS转座
- RISTransposition RIS转座
- geneTransposition 基因转座
- onePointRecombination 单点重组
- twoPointRecombination 两点重组
- geneRecombination 基因重组
4.1 Environment 类:
def modify(self): ''' 染色体修饰 mutation: 突变 ISTransposition IS转座 RISTransposition RIS转座 geneTransposition 基因转座 onePointRecombination 单点重组 twoPointRecombination 两点重组 geneRecombination 基因重组 :return: ''' for i in range(len(self.population)): chromosome = self.population[i] chromosome.mutation(self.mutationRate, self.homeoticRate) chromosome.ISTransposition(self.ISTranspositionRate, self.homeoticRate) chromosome.RISTransposition(self.RISTranspositionRate, self.homeoticRate) chromosome.geneTransposition(self.geneTranspositionRate, self.homeoticRate) #选择于i不同的染色体的,otherIndex otherIndex = i while otherIndex == i: otherIndex = random.randint(0, len(self.population) - 1) chromosome.onePointRecombination(self.onePointRecombinationRate, self.homeoticRate, self.population[otherIndex]) otherIndex = i while otherIndex == i: otherIndex = random.randint(0, len(self.population) - 1) chromosome.twoPointRecombination(self.twoPointRecombinationRate, self.homeoticRate, self.population[otherIndex]) otherIndex = i while otherIndex == i: otherIndex = random.randint(0, len(self.population) - 1) chromosome.geneRecombination(self.geneRecombinationRate, self.homeoticRate, self.population[otherIndex]) 4.2. Chromosome类:
4.2.1. 突变 mutation
变异(突变)可以发生在染色体内的任何位置。然而,染色体的结构组织必须保持完整。所以在头部中,任何符号都可以变成符号或者终点;在尾部中,终点只能够变成终点。通过这种方法,染色体的结构组织得以保持,而且由变异产生的新个体是结构上正确的程序。
def mutation(self, rate, homeoticRate): ''' :param rate: 基因突变的概率 :param homeoticRate: 连接基因突变的概率 :return: head: 保存基因头部 tail 保存基因尾部 functions 保存操作符如:【“+”,“-”,“*”,“/”】 ''' for gene in self.genes: head = gene.head tail = gene.tail functions = list(gene.genome.functions.keys()) functions.pop(functions.index("max_arity")) # 基因头部突变 for i in range(len(head)): if random.random() < rate: head[i] = random.choice(functions + gene.genome.terminals) # 基因尾部突变 for i in range(len(tail)): if random.random() < rate: tail[i] = random.choice(gene.genome.terminals) #连接基因的突变为 rate = homeoticRate*rate rate *= homeoticRate for homeotic in self.homeotics: head = homeotic.head tail = homeotic.tail functions = list(homeotic.genome.functions.keys()) functions.pop(functions.index("max_arity")) # 连接基因头部突变,突变的选择有些不同:functions + list(range(len(self.genes))) for i in range(len(head)): if random.random() < rate: head[i] = random.choice(functions + list(range(len(self.genes)))) # 连接基因尾部部突变 for i in range(len(tail)): if random.random() < rate: tail[i] = random.randint(0, len(self.genes) - 1) 4.2.2 IS转座 ISTransposition
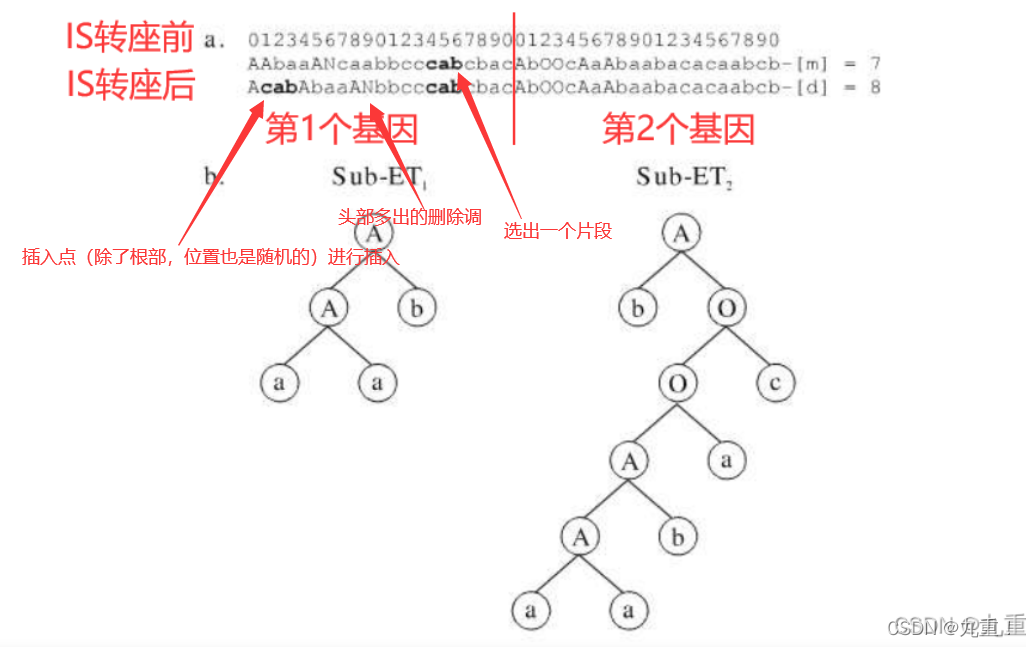
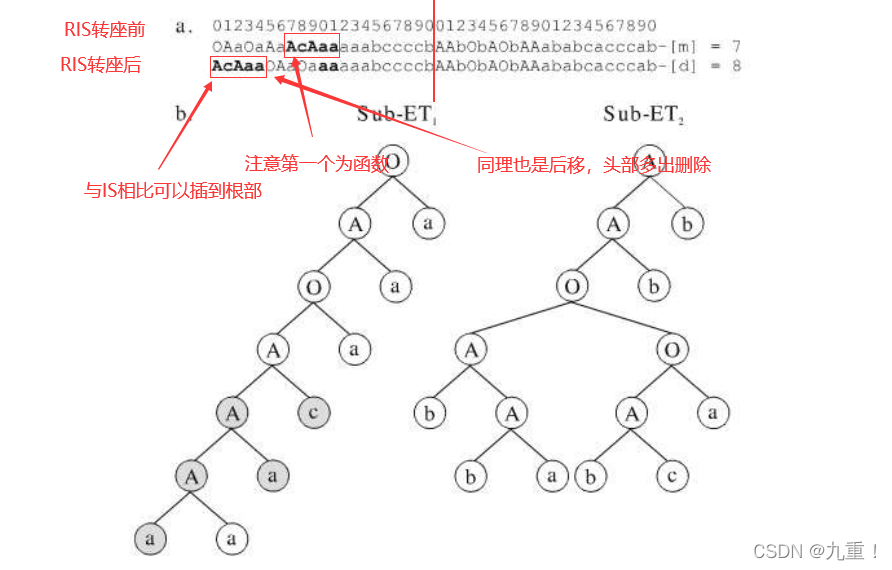
【PS:IS转座是从种群中随机选择一个染色体,然后随机从IS转座长度中选择IS长度,然后在染色体中选择IS长度的基因片段,并随机选择基因,插入到除基因首元素之外的头部部分中】
def ISTransposition(self, rate, homeoticRate): ''' IS转座是从种群中随机选择一个染色体,然后随机从IS转座长度中选择IS长度, 然后在染色体中选择IS长度的基因片段,并随机选择基因,插入到除基因首元素之外的头部部分中。 :param rate: 基因IS概率 :param homeoticRate: 连接基因IS概率 :return: seq 将所有基因进行连接保存 start 选择一小段的起点 stop 选择一小段的终点 ISSeq 保存选出的小段 insertPoint 插入点 ''' # 出现IS转座 if random.random() < rate: seq = [] for gene in self.genes: seq = seq + gene.head + gene.tail # 随机选择一个基因 gene = random.choice(self.genes) headLength = len(gene.head) # 选择截取一小断保存在 ISSeq 中 start = random.randint(0, len(seq) - 1) stop = random.randint(start, start + headLength) ISSeq = seq[start:stop + 1] # 选择一个插入点,进行插入,并且去掉超过长度的部分 insertPoint = random.randint(1, headLength - 1) for i in range(len(ISSeq)): gene.head.insert(insertPoint + i, ISSeq[i]) gene.head = gene.head[:headLength] #连接基因的IS转座为 rate = homeoticRate*rate rate *= homeoticRate if random.random() < rate: seq = [] for homeotic in self.homeotics: seq = seq + homeotic.head + homeotic.tail # 随机选择一个基因 homeotic = random.choice(self.homeotics) headLength = len(homeotic.head) # 选择截取一小断保存在 ISSeq 中 start = random.randint(0, len(seq) - 1) stop = random.randint(start, start + headLength) ISSeq = seq[start:stop + 1] insertionPoint = random.randint(1, headLength - 1) # 选择一个插入点,进行插入,并且去掉超过长度的部分 for i in range(len(ISSeq)): homeotic.head.insert(insertionPoint + i, ISSeq[i]) homeotic.head = homeotic.head[:headLength] 4.2.2 RIS转座 RISTransposition
【PS:所有的RIS元素都是从一个函数开始,因此是选自头部分中的序列。因此在头中任选一点,沿基因向后查找,直到发现一个函数为止。该函数成为RIS元素的起始位置。如果找不到任何函数,则变换不执行任何操作。该算子随机选取染色体,需要修饰的基因,RIS元素以及其长度。】
def RISTransposition(self, rate, homeoticRate): ''' :param rate: 基因RIS转座概率 :param homeoticRate: 连接基因RIS转座概率 :return: seq 将所有基因进行连接保存 functions 保存操作符如:【“+”,“-”,“*”,“/”】 start 选择一小段的起点 end 选择一小段的终点 RISSeq 保存选出的小段 insertPoint 插入点 ''' # 进行RIS转座 if random.random() < rate: seq = [] for gene in self.genes: seq = seq + gene.head + gene.tail # 随机选择一个基因 gene = random.choice(self.genes) headLength = len(gene.head) functions = list(gene.genome.functions.keys()) functions.pop(functions.index("max_arity")) # 选择截取一小断保存在 RISSeq 中 start = random.randint(0, len(seq) - 1) #直到发现一个函数为止 while seq[start] not in functions: if start == len(seq) - 1: break start += 1 stop = random.randint(start, start + headLength) RISSeq = seq[start:stop + 1] #进行插入,并且去掉超过长度的部分 for i in range(len(RISSeq)): gene.head.insert(i, RISSeq[i]) gene.head = gene.head[:headLength] #连接基因的RIS转座为 rate = homeoticRate*rate rate *= homeoticRate if random.random() < rate: seq = [] for homeotic in self.homeotics: seq = seq + homeotic.head + homeotic.tail # 随机选择一个基因 homeotic = random.choice(self.homeotics) headLength = len(homeotic.head) functions = list(homeotic.genome.functions.keys()) functions.pop(functions.index("max_arity")) # 选择截取一小断保存在 RISSeq 中 start = random.randint(0, len(seq) - 1) while seq[start] not in functions: if start == len(seq) - 1: break start += 1 stop = random.randint(start, start + headLength) #进行插入,并且去掉超过长度的部分 RISSeq = seq[start:stop + 1] for i in range(len(RISSeq)): homeotic.head.insert(i, RISSeq[i]) homeotic.head = homeotic.head[:headLength] 4.2.3 基因转座 geneTransposition
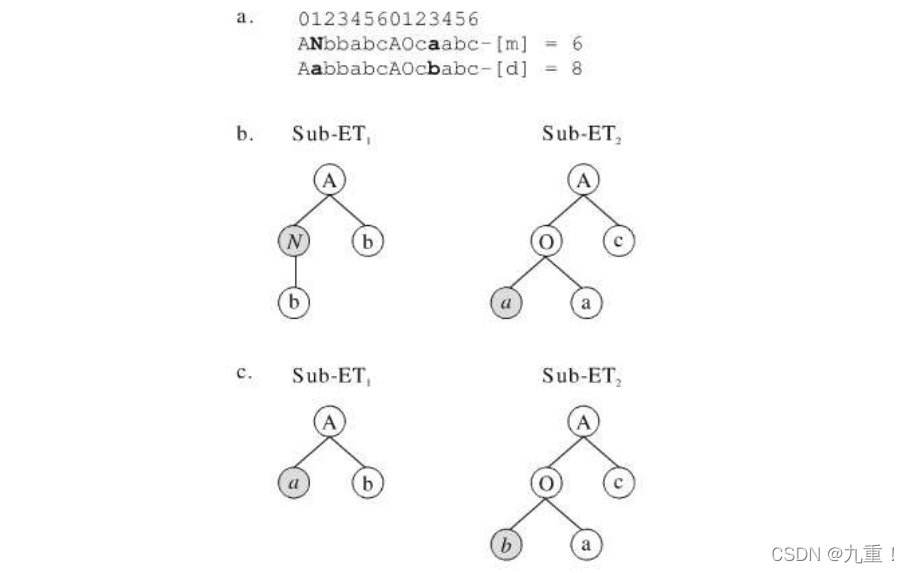
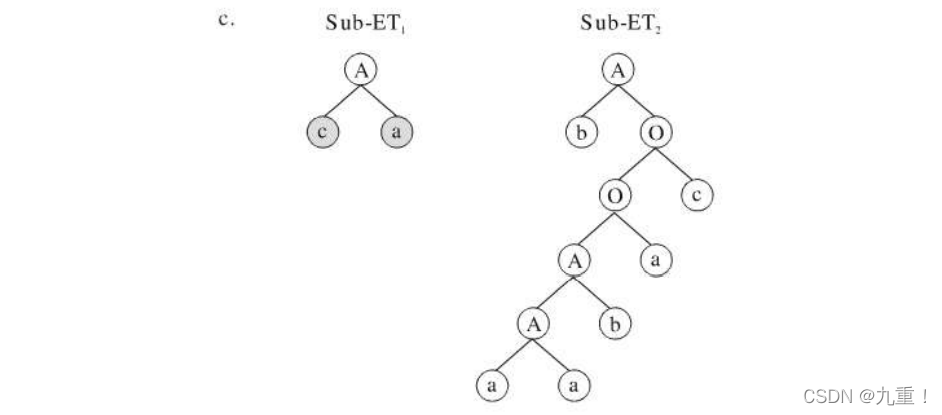
基因转座仅仅改变基因在同一染色体中的位置,如图所示:
def geneTransposition(self, rate, homeoticRate): ''' 基因转座仅仅改变基因在同一染色体中的位置 :param rate: 基因转座概率 :param homeoticRate: 连接基因转座概率 :return: ''' if random.random() < rate: self.genes[random.randint(0,len(self.genes)-1)] = random.choice(self.genes).replicate() rate *= homeoticRate if random.random() < rate: self.homeotics[random.randint(0,len(self.homeotics)-1)] = random.choice(self.homeotics).replicate() 4.2.4 单点重组 onePointRecombination
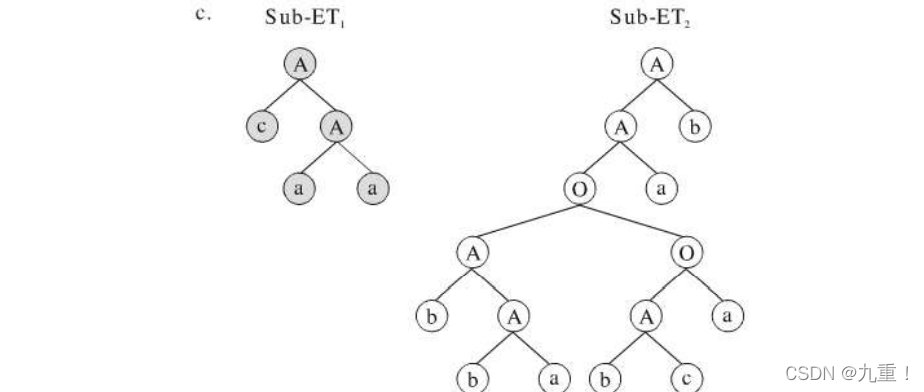
进行单点重组的时候,父代染色体相互配对并在相同的位置切断,两个染色体相互交换重组点之后的部分。
def onePointRecombination(self, rate, homeoticRate, otherChromosome): ''' 进行单点重组的时候,父代染色体相互配对并在相同的位置切断,两个染色体相互交换重组点之后的部分 :param rate: 单点重组概率 :param homeoticRate: 连接基因单点重组概率 :param otherChromosome: 染色体 :return: seq 当前染色体将所有基因进行连接保存 otherSeq 不同于当前染色体将所有基因进行连接保存 recombinationPoint 重组结点 ''' # 进行单点重组 if random.random() < rate: seq = [] otherSeq = [] for gene in self.genes: seq = seq + gene.head + gene.tail for otherGene in otherChromosome.genes: otherSeq = otherSeq + otherGene.head + otherGene.tail if len(seq) != len(otherSeq): return # 进行单点重组(交换) recombinationPoint = random.randint(0, len(seq) - 1) seq[recombinationPoint:], otherSeq[recombinationPoint:] = otherSeq[recombinationPoint:], seq[recombinationPoint:] # 单点重组后,当前染色体进行保存 for gene in self.genes: gene.head = seq[:len(gene.head)] del seq[0:len(gene.head)] gene.tail = seq[:len(gene.tail)] del seq[0:len(gene.tail)] # 单点重组后,不同于当前染色体进行保存 for otherGene in otherChromosome.genes: otherGene.head = otherSeq[:len(otherGene.head)] del otherSeq[0:len(otherGene.head)] otherGene.tail = otherSeq[:len(otherGene.tail)] del otherSeq[0:len(otherGene.tail)] #连接基因的单点重组为 rate = homeoticRate*rate rate *= homeoticRate if random.random() < rate: seq = [] otherSeq = [] for homeotic in self.homeotics: seq = seq + homeotic.head + homeotic.tail for otherHomeotic in otherChromosome.homeotics: otherSeq = otherSeq + otherHomeotic.head + otherHomeotic.tail if len(seq) != len(otherSeq): return # 进行单点重组(交换) recombinationPoint = random.randint(0, len(seq) - 1) seq[recombinationPoint:], otherSeq[recombinationPoint:] = otherSeq[recombinationPoint:], seq[recombinationPoint:] # 单点重组后,当前染色体进行保存 for homeotic in self.homeotics: homeotic.head = seq[:len(homeotic.head)] del seq[0:len(homeotic.head)] homeotic.tail = seq[:len(homeotic.tail)] del seq[0:len(homeotic.tail)] # 单点重组后,不同于当前染色体进行保存 for otherHomeotic in otherChromosome.homeotics: otherHomeotic.head = otherSeq[:len(otherHomeotic.head)] del otherSeq[0:len(otherHomeotic.head)] otherHomeotic.tail = otherSeq[:len(otherHomeotic.tail)] del otherSeq[0:len(otherHomeotic.tail)] 4.2.5 两点重组 twoPointRecombination
进行两点重组的时候,父代染色体相互配对,在染色体中随机选择两个点,将染色体切断。两个染色体相互交换重组点之间的部分,形成两个新的子代染色体。
def twoPointRecombination(self, rate, homeoticRate, otherChromosome): ''' 进行两点重组的时候,父代染色体相互配对,在染色体中随机选择两个点,将染色体切断。 两个染色体相互交换重组点之间的部分,形成两个新的子代染色体 :param rate: 两点重组概率 :param homeoticRate: 连接基因两点重组概率 :param otherChromosome: 不同于当前的染色体 :return: seq 当前染色体将所有基因进行连接保存 otherSeq 不同于当前染色体将所有基因进行连接保存 recombinationPoint 重组结点 otherPoint 另一个重组结点 ''' # 进行两点重组 if random.random() < rate: seq = [] otherSeq = [] for gene in self.genes: seq = seq + gene.head + gene.tail for otherGene in otherChromosome.genes: otherSeq = otherSeq + otherGene.head + otherGene.tail if len(seq) != len(otherSeq): return # 进行两点重组(交换) recombinationPoint = random.randint(0, len(seq) - 1) otherPoint = random.randint(recombinationPoint, len(seq) - 1) seq[recombinationPoint:], otherSeq[recombinationPoint:] = otherSeq[recombinationPoint:], seq[recombinationPoint:] seq[:otherPoint], otherSeq[:otherPoint] = otherSeq[:otherPoint], seq[:otherPoint] # 两点重组后,当前染色体进行保存 for gene in self.genes: gene.head = seq[:len(gene.head)] del seq[0:len(gene.head)] gene.tail = seq[:len(gene.tail)] del seq[0:len(gene.tail)] # 两点重组后,不同于当前染色体进行保存 for otherGene in otherChromosome.genes: otherGene.head = otherSeq[:len(otherGene.head)] del otherSeq[0:len(otherGene.head)] otherGene.tail = otherSeq[:len(otherGene.tail)] del otherSeq[0:len(otherGene.tail)] #连接基因的两点重组为 rate = homeoticRate*rate rate *= homeoticRate # 进行两点重组 if random.random() < rate: seq = [] otherSeq = [] for homeotic in self.homeotics: seq = seq + homeotic.head + homeotic.tail for otherHomeotic in otherChromosome.homeotics: otherSeq = otherSeq + otherHomeotic.head + otherHomeotic.tail if len(seq) != len(otherSeq): return # 进行两点重组(交换) recombinationPoint = random.randint(0, len(seq) - 1) otherPoint = random.randint(recombinationPoint, len(seq) - 1) seq[recombinationPoint:], otherSeq[recombinationPoint:] = otherSeq[recombinationPoint:], seq[recombinationPoint:] seq[:otherPoint], otherSeq[:otherPoint] = otherSeq[:otherPoint], seq[:otherPoint] # 两点重组后,当前染色体进行保存 for homeotic in self.homeotics: homeotic.head = seq[:len(homeotic.head)] del seq[0:len(homeotic.head)] homeotic.tail = seq[:len(homeotic.tail)] del seq[0:len(homeotic.tail)] # 两点重组后,不同于当前染色体进行保存 for otherHomeotic in otherChromosome.homeotics: otherHomeotic.head = otherSeq[:len(otherHomeotic.head)] del otherSeq[0:len(otherHomeotic.head)] otherHomeotic.tail = otherSeq[:len(otherHomeotic.tail)] del otherSeq[0:len(otherHomeotic.tail)] 4.2.5 基因重组 geneRecombination
在 GEP 的第三种重组中,两个染色体中的整个基因相互交换,形成的两个子代染色体含有来自两个父体的基因。
def geneRecombination(self, rate, homeoticRate, otherChromosome): ''' 在 GEP 的第三种重组中,两个染色体中的整个基因相互交换, 形成的两个子代染色体含有来自两个父体的基因。 :param rate: 基因重组概率 :param homeoticRate: 连接基因重组概率 :param otherChromosome: 不同于当前的染色体 :return: ''' # 进行基因重组 if random.random() < rate: if len(self.genes) != len(otherChromosome.genes): return recombinationPoint = random.randint(0, len(self.genes) - 1) self.genes[recombinationPoint:recombinationPoint+1], otherChromosome.genes[recombinationPoint:recombinationPoint+1] = otherChromosome.genes[recombinationPoint:recombinationPoint+1], self.genes[recombinationPoint:recombinationPoint+1] #连接基因的基因重组为 rate = homeoticRate*rate rate *= homeoticRate if random.random() < rate: if len(self.homeotics) != len(otherChromosome.homeotics): return recombinationPoint = random.randint(0, len(self.homeotics) - 1) self.homeotics[recombinationPoint:recombinationPoint+1], otherChromosome.homeotics[recombinationPoint:recombinationPoint+1] = otherChromosome.homeotics[recombinationPoint:recombinationPoint+1], self.homeotics[recombinationPoint:recombinationPoint+1] 5. 打印染色体
Chromosome 类
def printChromosome(self): for gene in self.genes: print(gene.head + gene.tail, end=':\n') for homeotic in self.homeotics: print(homeotic.head + homeotic.tail, end=';\n') print() 6. 将以上功能更新到run上
Environment类:
def run(self, inputsOutputs, evalFunction,generationCount=200,M=100,DataCount=10): ''' :param inputsOutputs: 保存输入与输出集合 :param evalFunction: 适应度函数 :param generationCount: 执行代数 :return: generation: 表示第几代种群 i_chromosome 用于保存当代种群第i条染色体 C_valueList 表示染色体,样本输出值 fitness 保存当前染色体的适应度 maxFitness_flag=True 为True,返回的maxFitness:M*DataCount ''' ResultBestFitness=float("-inf") ResultBestChromosome=[] generation = 0 while True: sum_fitness=0 NowBestFitness=float("-inf") NowBestChromosome=[] #进行种群打印 print("generation: ",generation) # 可以打印每一染色出来看。。。 # self.printChromosomes(generation) generation += 1 for i in range(len(self.population)): C_valueList = [] i_chromosome = self.population[i] #计算每个染色体对应的 C_value # print(inputsOutputs[0]) for inputs in inputsOutputs[0]: C_valueList.append(i_chromosome.eval(inputs)) #evalFunction 进行适应度评估 fitness = evalFunction(C_valueList, inputsOutputs[1], M=M, DataCount=DataCount) sum_fitness=sum_fitness+fitness #保存当代种群最佳适应度的值,与对应的染色体 if fitness > NowBestFitness: NowBestFitness = fitness NowBestChromosome=i_chromosome # 完美解停止执行 if fitness == evalFunction([], [], maxFitness_flag=True,M=M,DataCount=DataCount): print("*" * 46, " 完美解 ", "*" * 46) i_chromosome.printChromosome() return i_chromosome ##-----输出测试-----## # print("[",i,"] "," Fitness=",fitness) ####----------输出测试----------#### self.population[i] = (i_chromosome, fitness) # 打印当代最佳染色体 print("NowBestFitness=",NowBestFitness) NowBestChromosome.printChromosome() print("*"*46) # 保存最终最佳解,与对应的染色体 if ResultBestFitness<NowBestFitness: ResultBestFitness=NowBestFitness ResultBestChromosome=NowBestChromosome # 繁衍代数大于generationCount退出, 并输出最佳解 if generation >= generationCount: print("*"*46," ",generationCount,"代后最佳解 ","*"*46) print("result_fitness= ", ResultBestFitness) ResultBestChromosome.printChromosome() return ResultBestChromosome selected=self.select(sum_fitness,NowBestChromosome) self.replicate(selected) self.modify() self.population[0]=NowBestChromosome 6. 输出测试
if __name__=='__main__': #---------------操作数设置 Begin--------------# genome = Genome() genome.functions.update(Genome.linker_set) genome.terminals = ['a','b'] genome.symbols() my_link_genome=Genome() my_link_genome.functions.update(Genome.linker_set) #----------------操作数设置 end---------------# #---------------种群初始化 Begin--------------# environment = Environment() environment.init(populationSize=20,numGenes=3,numHomeotics=1,headLength=4,homeoticHeadLength=5,genome=genome,link_genome=my_link_genome) environment.setRates(homeoticRate=0.5, mutationRate=0.044,ISTranspositionRate=0.1, RISTranspositionRate=0.1, geneTranspositionRate=0.1,onePointRecombinationRate=0.3, twoPointRecombinationRate=0.3, geneRecombinationRate=0.1) # ##----------输出测试----------## #----------------种群初始化 end---------------# #---------------Run Begin--------------# M=100 DataCount=10 generationCount=1000 inputsOutputs=GenerateData(DataCount=DataCount) result=environment.run(inputsOutputs,evalFunction,M=M,generationCount=generationCount) #----------------Run end---------------# 输出结果:
部分结果
********************************************** generation: 999 0 :['b', 'b', 'a', 'a', 'a', 'b', 'b', 'b', 'b']:['+', '+', '+', 'b', 'b', 'b', 'a', 'a', 'b']:['+', '+', 'b', '+', 'a', 'a', 'a', 'a', 'a']:['+', 1, '+', '+', 1, 1, 0, 0, 0, 1, 2]: 1 :['*', '+', '+', '+', 'a', 'b', 'b', 'b', 'b']:['+', 'a', 'b', 'b', 'b', 'a', 'a', 'b', 'a']:['+', '+', '+', 'a', 'a', 'a', 'a', 'a', 'a']:[1, 0, '+', 1, '+', 1, 2, 0, 0, 1, 2]: 2 :['+', '+', '+', '/', 'a', 'b', 'a', 'b', 'b']:['+', '+', 'a', 'b', 'b', 'b', 'a', 'a', 'b']:['+', '+', '+', 'a', 'a', 'a', 'a', 'a', 'a']:[0, '+', 0, '-', 0, 1, 2, 2, 0, 0, 0]: 3 :['+', 'a', '/', 'a', 'a', 'b', 'b', 'b', 'b']:['b', 'b', '/', 'a', 'b', 'a', 'a', 'a', 'a']:['+', 'a', 'b', 'a', 'a', 'b', 'b', 'b', 'a']:['+', 0, '/', 0, 1, 1, 2, 2, 0, 0, 0]: 4 :['*', '+', '+', '+', 'a', 'b', 'a', 'b', 'b']:['+', '+', 'a', '+', 'b', 'a', 'a', 'a', 'b']:['+', 'b', '+', '/', 'a', 'a', 'a', 'a', 'b']:[0, '+', 0, '-', 0, 1, 2, 2, 0, 0, 0]: 5 :['/', '*', 'a', 'a', 'a', 'b', 'b', 'b', 'b']:['/', 'a', 'b', 'b', 'a', 'b', 'b', 'b', 'b']:['a', 'a', 'b', '+', 'b', 'b', 'a', 'a', 'a']:['+', 0, '+', 0, 2, 1, 2, 2, 0, 0, 0]: 6 :['*', 'a', 'a', '/', 'a', 'b', 'b', 'b', 'b']:['*', 'a', 'a', 'a', 'a', 'b', 'b', 'a', 'b']:['+', 'b', '+', '+', 'b', 'b', 'b', 'a', 'a']:[2, 1, '+', 0, 0, 1, 2, 1, 0, 1, 0]: 7 :['+', '-', 'a', 'b', 'a', 'a', 'a', 'b', 'b']:['b', 'b', '/', '/', 'b', 'a', 'a', 'a', 'b']:['+', '+', '+', '+', 'a', 'a', 'a', 'a', 'a']:[0, '+', 2, '+', 0, 1, 2, 2, 2, 0, 0]: 8 :['+', '-', 'a', 'b', 'a', 'a', 'a', 'b', 'b']:['b', 'b', '/', 'a', 'b', 'a', 'a', 'a', 'a']:['+', '+', '+', '/', 'a', 'a', 'b', 'b', 'b']:['+', 0, '+', 0, 2, 1, 2, 2, 0, 0, 0]: 9 :['+', '+', 'a', 'b', 'a', 'a', 'a', 'b', 'a']:['+', 'a', 'a', 'a', 'b', 'a', 'a', 'b', 'b']:['+', '+', '+', '+', 'a', 'a', 'b', 'a', 'a']:['+', 1, '+', 1, 0, 1, 0, 0, 0, 1, 0]: 10 :['+', 'a', 'a', '-', 'a', 'b', 'a', 'b', 'b']:['+', '+', 'a', 'b', 'b', 'b', 'a', 'a', 'b']:['+', '+', '+', '/', 'a', 'a', 'b', 'b', 'b']:['+', 1, 2, '+', 0, 1, 2, 2, 0, 0, 0]: 11 :['+', 'a', 'a', '-', 'a', 'b', 'a', 'b', 'b']:['+', '+', 'a', 'b', 'b', 'a', 'a', 'a', 'b']:['+', '+', '+', '/', 'a', 'a', 'b', 'b', 'b']:['+', 1, 2, '+', 0, 1, 2, 0, 0, 0, 0]: 12 :['+', 'a', 'a', '-', 'a', 'b', 'b', 'b', 'b']:['+', 'a', 'b', 'a', 'b', 'b', 'a', 'b', 'a']:['+', '+', '+', '/', 'a', 'a', 'b', 'b', 'b']:['+', 1, 2, '+', 0, 1, 2, 2, 0, 0, 0]: 13 :['-', '+', 'b', 'b', 'b', 'a', 'a', 'b', 'a']:['a', '+', '/', 'a', 'b', 'b', 'a', 'a', 'a']:['+', '+', '+', '/', 'a', 'a', 'b', 'b', 'b']:['+', 0, '+', 0, '+', 1, 2, 2, 0, 1, 0]: 14 :['+', 'a', 'a', 'a', 'a', 'b', 'a', 'b', 'b']:['a', '+', 'b', 'a', 'a', 'b', 'a', 'b', 'b']:['+', '+', 'b', '+', 'b', 'a', 'b', 'a', 'a']:[2, 1, '+', 0, 0, 1, 2, 1, 0, 1, 2]: 15 :['+', '+', '/', 'a', 'a', 'b', 'b', 'b', 'b']:['b', 'b', 'b', 'b', 'b', 'b', 'a', 'a', 'b']:['a', '+', '+', '+', 'b', 'a', 'b', 'b', 'b']:[0, '+', 0, '+', 2, 1, 0, 0, 0, 2, 0]: 16 :['+', '+', '+', '/', 'b', 'b', 'a', 'b', 'b']:['+', '+', 'a', '+', 'b', 'a', 'a', 'b', 'b']:['a', '+', 'b', '+', 'a', 'a', 'a', 'a', 'b']:[0, '+', 1, '+', 0, 1, 0, 0, 2, 1, 0]: 17 :['+', 'a', 'a', '-', 'a', 'b', 'a', 'b', 'b']:['+', '+', 'a', 'b', 'b', 'b', 'a', 'b', 'b']:['+', '+', '+', '/', 'a', 'a', 'b', 'b', 'b']:['+', 1, 2, '+', 0, 1, 2, 2, 0, 0, 0]: 18 :['b', '+', 'a', 'b', 'a', 'a', 'b', 'b', 'b']:['b', 'b', 'b', 'a', 'a', 'a', 'a', 'a', 'b']:['*', 'a', '/', 'a', 'a', 'a', 'a', 'a', 'b']:[0, 0, '+', 0, '+', 1, 0, 0, 0, 1, 2]: 19 :['+', '*', '+', 'a', 'a', 'a', 'a', 'b', 'b']:['+', 'a', 'a', 'a', 'b', 'a', 'a', 'b', 'b']:['+', '+', '+', 'a', 'a', 'a', 'b', 'b', 'b']:['+', 1, '+', 1, 0, 1, 0, 2, 0, 0, 0]: NowBestFitness= 906 ['*', '+', '+', '+', 'a', 'b', 'a', 'b', 'b']: ['+', '+', 'a', '+', 'b', 'a', 'a', 'a', 'b']: ['+', 'b', '+', '/', 'a', 'a', 'a', 'a', 'b']: [0, '+', 0, '-', 0, 1, 2, 2, 0, 0, 0]; ********************************************** 1000 代后最佳解 ********************************************** result_fitness= 988.0 ['+', '+', 'b', 'a', 'a', 'a', 'b', 'b', 'b']: ['a', '+', '/', 'a', 'b', 'a', 'b', 'a', 'b']: ['+', '+', '*', '/', 'b', 'a', 'b', 'b', 'b']: ['+', '+', '+', '+', '+', 0, 0, 0, 1, 2, 2]; 7. 代码实现整合
为了方便演示,我将每个类整合到一个py文件上,GEP-6 复制修饰实现(GEP基本实现了)
本文作者:九重!
本文链接:https://blog.csdn.net/weixin_/article/details/
关于博主:评论和私信会在第一时间回复。或者直接私信我。
声援博主:如果您觉得文章对您有帮助,可以点击文章右下角【点赞】【收藏】一下。您的鼓励是博主的最大动力!
今天的文章
基因表达的重编程_中文编程分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/81257.html