目录
前言
随着三代纳米孔测序技术的不断发展,Oxford Nanopore Technologies(ONT)公司在持续更新其测序试剂耗材(比如发布R10版本的Flow cell和Kit14系列测序试剂)的同时,也在同步更新其配套分析软件,如碱基识别软件Guppy/Dorado/Bonito、碱基修饰识别软件Remora、测序控制软件MinKNOW、一致性序列生成软件Medaka等。纳米孔测序技术虽然有着便捷、实时、读长长等特点,但其本身存在一定的测序错误。ONT官方已经发布了多个版本的Medaka,该工具基于修正序列(sequence correction)来生成一致性序列和鉴别突变。
一、Medaka是什么?
medakais a tool to create consensus sequences and variant calls from nanopore sequencing data. This task is performed using neural networks applied a pileup of individual sequencing reads against a draft assembly. It provides state-of-the-art results outperforming sequence-graph based methods and signal-based methods, whilst also being faster.
这是ONT官方对Medaka的基本定义,目前已更新至1.7.3版本。该软件的更新几乎与ONT测序芯片和测序试剂的更新迭代同步。比如,ONT发布了最新的R10.4.1芯片和E8.2(Kit14系列)测序试剂不久,Medaka就随之更新,以支持上述最新测序试剂耗材产生的测序数据的分析。
该软件最大的特点就是分析速度快且无需读入原始测序电信号fast5文件。仅需fasta或fastq文件即可开始分析。与ONT早期突变识别工具Nanopolish相比,速度提升极大。
Features
- Requires only basecalled data. (
.fastaor.fastq)- Improved accuracy over graph-based methods (e.g. Racon).
- 50X faster than Nanopolish (and can run on GPUs).
- Includes extras for implementing and training bespoke correction networks.
- Works on Linux and MacOS.
- Open source (Mozilla Public License 2.0).
二、使用步骤
1.安装
可参考其Github网页进行安装,地址为https://github.com/nanoporetech/medaka.
(1)conda安装
在Terminal中输入如下命令即可安装:
conda create -n medaka -c conda-forge -c bioconda medaka(2)pip安装
另一种便捷的安装方式是使用pip。但要注意的是首先提前安装依赖的其他软件,包括:
- samtools
- minimap2
- tabix
- bgzip
然后再运行如下命令:
pip install medaka其他安装方式可参考Github网页。
2.快速使用
(1)核心参数”-m”
Medaka基于序列修正来生成一致性序列和鉴别突变。前面提到,ONT的测序准确度虽然一直在提升,但其长读长序列仍存在一定错误,因此仍需进行校正操作以提升下游分析的准确性。不论是生成一致性序列还是鉴别突变,都需要向软件提供”-m”参数及具体模型。该参数与测序使用的碱基识别软件版本(一般是Guppy)、碱基识别模型(fast/hac/sup)、测序芯片(R9或R10)、测序试剂(Kit9、Kit10、Kit12或Kit14)和测序仪(MinION/GridION/PromethION)等密切相关。其基本模式为:
{pore}_{device}_{caller variant}_{caller version}比如r941_min_fast_g303可以处理的数据应满足如下标准:在MinION或GridION测序仪上使用R9.4.1芯片上测序且用Guppy v3.0.3 快速碱基识别模型(fast)产生的测序数据。 再比如,r941_prom_hac_g507可以处理的数据应满足如下标准:在PromethION测序仪上使用R9.4.1芯片上测序且用Guppy v5.0.7高准确度识别模型(high accurate)产生的测序数据。
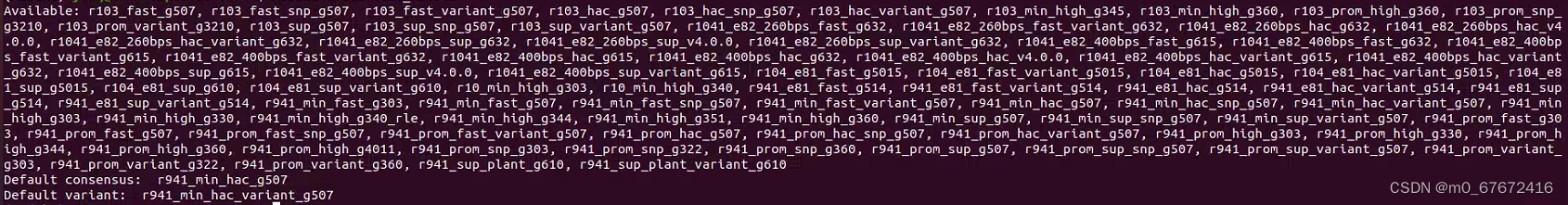
可使用如下命令来查看Medaka支持的模型。当实际测序使用的Guppy版本与Medaka模型不一致时,选择的Medaka模型中的Guppy版本不得高于测序使用的Guppy版本。
medaka tools list_models运行完上述命令以后可以得到如下模型列表,还会看到诸如r941_min_sup_variant_g507等包含variant关键词的模型,这些模型用于鉴别基因组突变。
(2)生成一致性序列
可使用medaka_consensus来生成一致性序列。基本输入包括拼接后的基因组草图,拼接时使用的fastq或fasta文件以及匹配的model文件,基本使用方法如下:
NPROC=$(nproc)
BASECALLS=basecalls.fa
DRAFT=draft_assm/assm_final.fa
OUTDIR=medaka_consensus
medaka_consensus -i ${BASECALLS} -d ${DRAFT} -o ${OUTDIR} -t ${NPROC} -m r941_min_high_g303生成的一致性序列在-o指定的目录里。这里需要注意的是,基因组草图应使用拼接软件Flye(https://github.com/fenderglass/Flye)生成。
(3)鉴别单倍体突变
可使用medaka_haploid_variant来鉴别细菌或病毒基因组突变。基本输入包括参考序列、测序fastq或fasta、匹配的model文件等,基本命令行如下:
medaka_haploid_variant -r ${ref} -i basecalls.fq -m r941_min_sup_variant_g507 -t 12 -o ${name}_medaka
会在指定目录里产生medaka.annotated.vcf文件。需要注意的是,Medaka一般只能鉴别consensus水平的突变,如果想要鉴别低频的宿主内变异,需要使用其他软件如LoFreq、VarScan2或Bcftools来进行。
3.高阶应用
(1)其他用法
Medaka除上述使用方法外,还支持其他命令行调用方式:
比如medaka consensus和medaka snp或medaka variant连用可鉴别基因组突变,注意调用命令行时medaka与其他命令中间要加空格。
(2)ARTIC分析流程
新冠病毒肆虐全球时,ONT和一些科学家联合发布了针对新冠病毒扩增子测序的分析流程ARTIC,用户可选择使用该流程内置的Medaka软件(主要调用medaka consensus、medaka variant和medaka tools annotate )完成相关分析。
总结
本文简单介绍了Medaka的使用方法,对Medaka的GPU加速和并行计算并未提及,有机会会在以后的文章中继续交流。
今天的文章oxford imagine_maestro制谱软件分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/88479.html