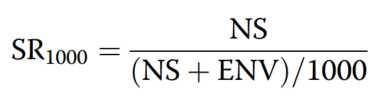文章目录
- 环境样品中微生物群落丰度的绝对定量
-
- 写在前面
- 摘要
- 背景
- 方法
- 结果
-
- 嵌合spiking可准确估算微生物的丰度
- 合成的spikes可以测量原位土壤DNA的丰度和不同土壤中的微生物群的组成
- 图 3 合成spike添加的模型
- 表 2 spike水平对rRNA基因估计拷贝数的影响
- 图 4 微生物群落结构的MDS图
- 测定两种对比土壤中的域丰度
- 图 5 利用合成的spikes对Bawburgh (B)和Wytham (W)土壤中原位微生物基因的丰度进行了估计
- 绝对定量改变了微生物群落的系统发育结构
- 图 6 Bawburgh和Wytham土壤中的原核和真核生物的群落结构
- 图 7 Bawburgh和Wytham土壤中的真菌群落结构
- 深入了解引物特异性
- 跨域比较
- 土壤中绝对和相对微生物群落丰度的比较
- 讨论
- 结论
- Reference
- 相关文章
- 猜你喜欢
- 写在后面

环境样品中微生物群落丰度的绝对定量
Absolute quantitation of microbiota abundance in environmental samples
Microbiome
Impact Factor 10.465
https://doi.org/10.1186/s40168-018-0491-7
发表日期:2018-06-19
第一作者:Andrzej Tkacz
通讯作者:Philip S. Poole(philip.poole@plants.ox.ac.uk)
合作作者:Marion Hortala
主要单位:
英国牛津大学植物科学系(Department of Plant Sciences, University of Oxford, South Parks Road, Oxford
OX1 3RB, UK)
写在前面
分享标题:Microbiome:绝对定量菌群丰度的新方法
关键字:spike,微生物群落,绝对定量,菌群分析方法,qPCR,相对丰度
点评:菌群相对丰度削弱了不同样品间的可比性,也难以揭示生物域水平的菌群结构变化。本研究采用人工合成的嵌合体DNA作为内参,可以计算土壤这样的复杂环境样品中的细菌绝对丰度,有助于不同样品间的比较,并且强调了绝对丰度与相对丰度在描述菌群变化中的差异。该成果对优化菌群分析手段、解读菌群相关的研究成果都具有参考价值,值得专业人士关注。国内中科院白洋团队和王二涛团队分别报导了植物样品中的绝对定量方法,详见(https://www.mr-gut.cn/papers/read/1082713111)和(https://www.mr-gut.cn/papers/read/1067164721)
摘要
背景
微生物组(microbiota)影响人类和动物的疾病和免疫、地球化学养分循环和植物生产力。我们通过PCR扩增特定的组,包括细菌,古细菌,真核生物或真菌,以评估亚组(例如属)的相对丰度。然而,既没有揭示亚组的绝对丰度,也没有比较不同的扩增子家族(即衍生自一对特定的PCR引物,例如细菌16S,真核生物18S或真菌ITS的OTUs)。这就阻碍了对特定群体绝对丰度的测定,而且微生物群落丰度的域水平的变化可能无法被检测到。
结果
我们已经开发出了使用合成的嵌合DNA 内参(spikes)对扩增子家族进行绝对定量的方法。将合成的内参直接添加到环境样品中,共提取并进行PCR扩增,从而可以计算扩增子家族的绝对丰度。(例如每单位质量样品的原核生物的16S,真核生物的18S和真菌ITS)。
结论
内参可适应任何扩增子特异性群体,包括土壤中的根瘤菌(rhizobia),人肠道的厚壁菌门(Firmicutes)和双歧枝菌(Bifidobacteria)或食品样品中的肠杆菌(Enterobacteriaceae)。重要的是,我们研究表明,通过使用高度复杂的土壤样本,即使特定群体的相对丰度降低,其绝对丰度也可以保持稳定或增加。因此,如果没有绝对定量,微生物群的潜在病理学、生理学和生态学可能会被它们的相对丰度所掩盖。
背景
在过去的十年里,从人们可以负担得起的扩增子DNA测序显示,这种微生物群影响着人类的免疫、消化、心理健康以及植物的生长发育。这些在不同领域的研究揭示了不同微生物群之间的微妙关系,就植物而言,突出了土壤微生物群落对植物健康的重要性。环境微生物群落具有丰富的多种组成成分,通常通过标记基因的PCR扩增进行研究。然而,在大多数研究中,只研究了一个微生物组中某一类别(域),如细菌。此外,大多数微生物组研究受到使用域特异性引物产生的PCR扩增子(来自单个环境样品的扩增基因)的限制,进而导致任何两组或两组以上PCR扩增子之间的定量比较丢失。然而,为了揭示肠道、土壤和其他环境的真正复杂性,必须确定主要微生物群之间的数量关系。
相互连接的细菌,古细菌,真菌,病毒和单细胞真核生物之间存在复杂的关系,所有这些生物均通过不同组的PCR引物进行不同程度的PCR扩增。目前的DNA技术允许对多个样本进行平行测序,产生数百万个短读长。分析原核生物群落最常见的方法是扩增16S rRNA基因,而真核生物的分析通常是通过18S rRNA的扩增。然而,从使用不同引物对的PCR实验来看,微生物群的任何跨域比较都是不可能的。当考虑到针对真菌或特定细菌群(如Bifidobacteria,其是肠道菌群的关键组成部分)设计的引物时,定量比较的问题变得更加棘手。有一些策略试图克服这个问题。基因编码蛋白,如Cpn60,普遍存在于原核生物、真核线粒体和叶绿体中。因此,它们可用于描述原核生物群落,并比较其相对丰度与真核宿主细胞器的丰度。此外,基于基因编码Cpn60的比较被证明在预测细菌基因组相似性方面是有用的。但是,该方法目前的局限性是参考数据库相对较小。人们基于原核和真核核糖体基因序列之间的相似性,开发了一套具有生命普遍性的引物。这是该领域的一个有希望的进展,但该引物是为海绵共生体开发的,在更复杂的肠道或土壤环境中可能会失去它们的系统发育相容性。还可以使用管家基因如rpoB、amoA、pmoA、nirS、nirK、nosZ和pufM进行微生物分析。在这些基因中,rpoB被证明在区分亲缘关系很近的物种方面非常有效。此外,这些基于编码Cpn60或通用16S/18S rRNA基因的基因的方法,尽管能够比较域内的相对丰度,但无法提供样品中总DNA丰度的估计。qPCR可用于估计微生物的丰度,因为它可以计算每个DNA总量中特定基因的拷贝数。然而,它没有提供关于环境原位基因丰度的信息。此外,在初始宏基因组测序之后,需要对每一群组进行单独和复杂的qPCR分析。其它方法包括基于三磷酸丰度(ATP),流式细胞术(FCM),磷脂脂肪酸(PLFA)和微生物生物量碳(MBC)的测量。
在一项突破性的研究中,我们使用了一种复杂的流式细胞术方法,确定了微生物组的丰富度是Crohn’s病的关键驱动因素。对生物绝对丰度的测量表明,Bacteroides与Prevotella(被认为是肠道健康的重要制造者)的比例,是相对定量的人工产物。虽然这种复杂的方法不容易转移到其他生物体上,但通过添加某一物种(在给定环境中通常找不到这种物种)已知数量的细菌细胞,有可能‘spike’ 样品。我们在哺乳动物肠道样品中掺入了肠道中不存在的极端土壤嗜盐菌,并且通过比较嗜盐菌的16S rRNA读长数量相对于其输入的丰度来确定所有群组的绝对丰度。这受限于对环境中缺少哪些细菌种类的先验知识的要求以及将培养物制备至高度受控的细胞密度的必要性。
在基于RNA序列的研究中,通常在RNA分离之前将合成的RNA标准品添加到环境样品中。然后对环境和合成的RNA进行共纯化,转化为cDNA,并进行测序。根据这些在测序输出中的标准化比率,可以比较样品之间的RNA相对丰度。
在这里,我们显示了RNA-seq方法的基于DNA的PCR扩增子适应的结果。我们设计了短的嵌合合成DNA片段,其中包含对三个主要微生物区域(原核生物,真核生物和真菌)具有特异性的通用引物结合位点。在PCR期间,由于存在合成填充区,这些合成DNA分子会产生各自的预期扩增子大小。将已知数量的合成DNA spikes直接添加到环境样本中,并计算它们在测序输出中的相对丰度,可以在扩增子类别之内和之间确定特定生物群的绝对丰度。在这里,我们展示了这种方法在纯细菌培养和复杂土壤样品中的优势。我们首先在一定数量的细菌细胞上测试我们生物spiking方法。之后,我们将展示如何将此方法应用于具有未知微生物结构的样品。根据不同的估计,土壤微生物群至少比肠道微生物群的多样性高一个数量级。因此,该方法可适用于分析较简单的环境,例如食物样品或人类/动物肠道的环境。令人惊讶的是,我们发现,当比较样品时,即使样品的绝对丰度较低,微生物类群的相对丰度也可能较高。
方法
P、E、F合成spikes的设计
Design of P, E and F synthetic spikes
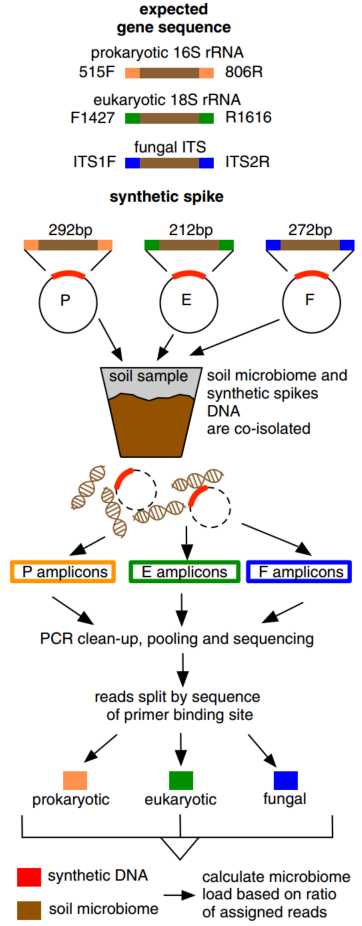
合成spikes的设计包含三个关键要素:(i)分别用于鉴定原核生物§,真核生物(E)和真菌(F)的常见基因的引物结合位点(PBSs);(ii)与体内靶标相同长度和GC含量的优化合成填充序列及(iii)易于获得,易于处理的合成spikes的DNA来源(图. 1)。
图 1 合成spike设计
Synthetic spike design
利用PBSs序列以及分别来自原核生物§,真核生物(E)和真菌(F)的扩增子的长度和GC含量共同分别设计了P、E、F合成spikes。对于P合成spikes,引物结合位点(PBS)以橙色显示,对于E为绿色,对于F为蓝色。
PBS基于选择用于微生物群扩增的三组PCR引物。对于原核生物§,使用原核生物的16S rRNA V4区引物515F和806R,因为它们是土壤研究中常用的引物。对于真核生物,我们使用了18S引物对F1427和R1616,其针对范围广泛的真核生物类群,包括藻类,硅藻,动物,挖掘物(原生生物,鞭毛),真菌和霉菌。第三对引物专门针对真菌(F),ITS1F和ITS2R,广泛用于土壤微生物群研究。这些引物针对森林土壤中常见的门Ascomycota和Basidiomycota中可变大小的ITS片段。扩增子长度的变异性会影响PCR扩增效率,因此P (16S rRNA)和E (18S rRNA)的填充序列的长度应与天然PCR产物的长度相匹配。真菌ITS扩增子在长度上表现出更大的可变性,但根据我们先前测序的结果,最常见的大小为272bp;因此,采用272bp的填充序列设计F的合成spike (图. 1)。在每种情况下,GC含量均设计为与其环境基因对应物相似,其序列使用随机DNA生成器设计(https://www.faculty.ucr.edu)。通过Geneart(Invitrogen)合成了P,E和F的合成spikes,并将其克隆到质粒pMA-T中,分别形成pSpike-P,pSpike-E和pSpike-F。将质粒转化到大肠杆菌中,并以质粒#101172,#101173和#101174的形式存储在https://www.addgene.com上。
土壤样品的特征
Characterisation of soil samples
使用的两种土壤都来自至少20年没有耕种的休耕土地。Bawburgh土含NO3– 3.49 mg kg -1, p-3120.5 mg kg -1, K+ 168.2 mg kg -1, Mg2+ 33.55 mg kg -1,有机质含量相对较低为2.92%,以及先前测量的pH7.5。Wytham土壤来自牛津大学Wytham森林,采集自一个位于北纬51°46’14’‘和西经1°20’18’’的森林开放地。其化学特征表明,其含有p-3122.9 mg kg -1, K+ 483.6 mg kg -1, Mg2+ 304.9 mg kg -1,有机质含量为16.78%,pH为7.22。Wytham 潜育土的矿物和有机质含量明显高于Bawburgh的淋溶土;然而,它们的pH值相似。在分析之前,将两种土壤风干,以使其重量和体积之间的差异最小。
微生物学技术
Microbiological techniques
Rhizobium leguminosarum bv. viciae 3841培养物(100 ml)在TY(一种丰富的生长培养基)中生长直至OD600为0.54,然后离心分离并重悬于50 ml的最小限度的培养基中以阻止细菌生长。采用连续稀释法进行细菌计数,以评估细菌数量。
通过光学密度(OD600)和平板计数法测定Rhizobium细胞的丰度。在每个实验中,每1 ml细菌悬浮液使用1.11* 109个菌落形成单位(cfu)。在添加合成spikes之前先提取DNA时,可对基于色谱柱分离过程中DNA丢失的数据进行校正(预测总DNA量除以混合了合成spikes 的DNA量,用于PCR)。
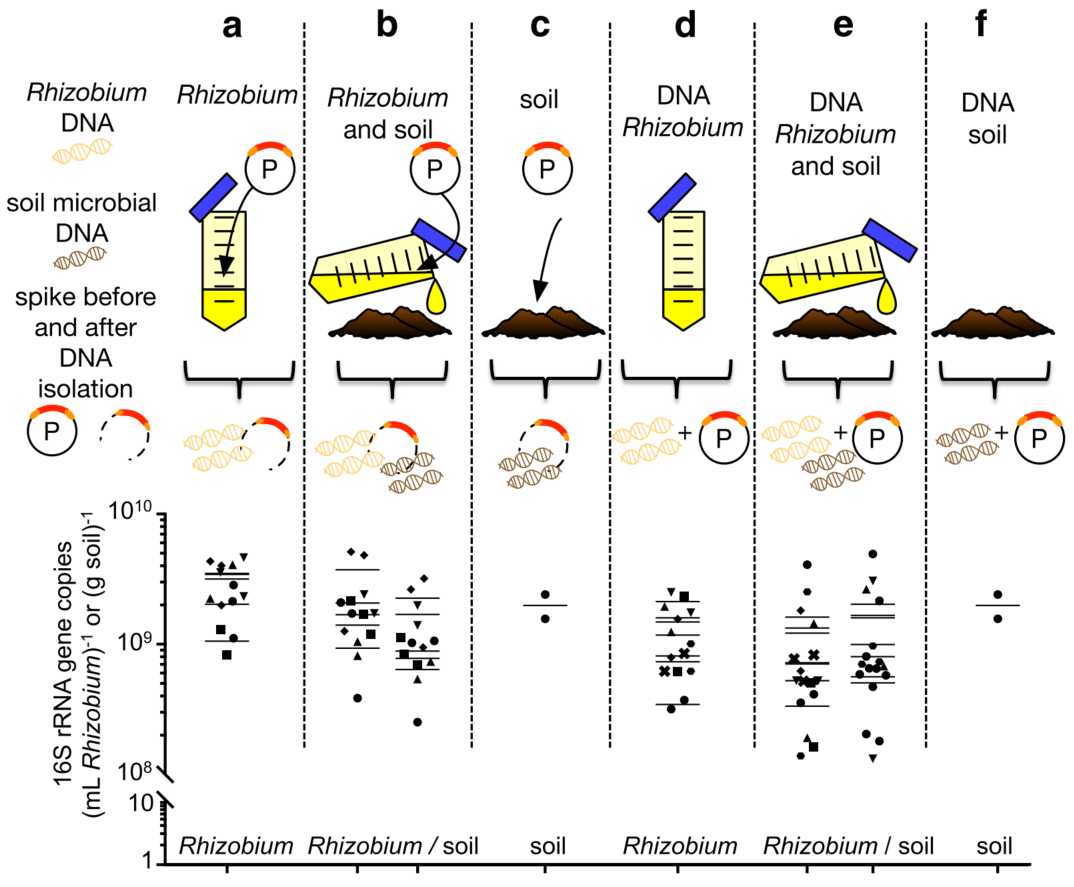
对于将Rhizobium细胞添加到土壤中的实验,在提取DNA之前(图. 2a–c),每1 mlRhizobium或1 g土壤中使用14至340 pg 的spikes的浓度梯度添加P合成的spike。对于将从Rhizobium中提取的DNA添加到Bawburgh土壤中的实验(图.2d–f),我们将P合成的spike以七个不同的浓度添加到先前提取的DNA中:以每1μg微生物的DNA 2、4、8、20、40、80和200 pg的量。
图 2 DNA提取前后添加的spikes对微生物定量的影响
Effect of added spikes before and after DNA isolation on microbial quantitation
在分离DNA(a-c)之前,将P合成的spike DNA添加到样品中,以如下浓度梯度(a-b):14(圆形),34(正方形),70(向上三角形),140(向下三角形)和340(菱形)pg 每(1毫升)Rhizobium培养物或(1克)土壤,和(c)39(正方形)pg每(1克)土壤,然后与微生物DNA共提取。还以2(圆形),4(正方形),8(向上三角形),20(向下三角形),40(菱形),80(六角形)和200(差号)pg每(μg)分离微生物DNA的浓度梯度将spikes添加到纯化的微生物DNA(d-f)中。a–c 合成的spike质粒DNA在DNA纯化过程中可能发生裂解。水平线代表每组样品的16S rRNA平均数量。整个过程中都使用了Bawburgh土壤。
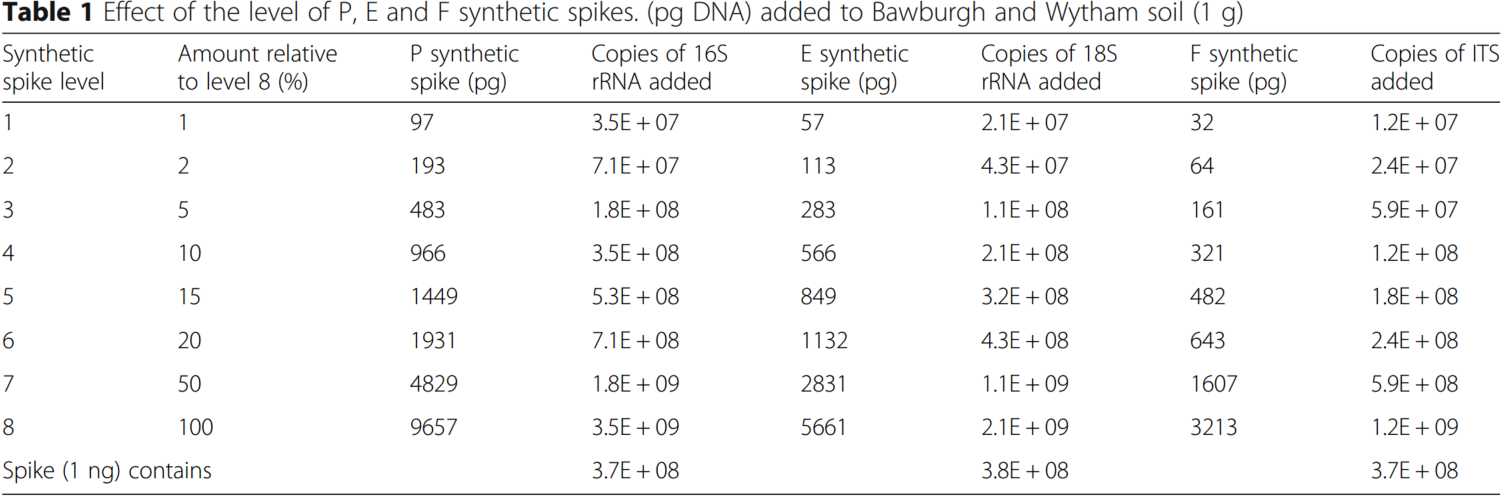
在比较Bawburgh和Wytham土壤的实验中,常规使用300 mg土壤。即使土壤充分混合,微生物组成和丰度也会有所变化。对于每种土壤,准备了24个样品,一式三份地使用了8种水平的PEF合成spikes(表1)。这些水平包含每个P,E和F合成spikes的确定的数量。根据我们以前的工作,我们预计与原核生物16S rRNA相比,真核生物18S rRNA和真菌ITS的丰度会降低,因此,我们使用了不同量的PEF spikes。
表 1 P、E、F合成spikes水平的影响
Effect of the level of P, E and F synthetic spikes
(pg DNA)被添加到Bawburgh和Wytham土壤中(1g)
根据制造商的说明,使用土壤DNA提取试剂盒(D6001,Zymo research,Irvine,US)对样品进行处理。共提取微生物和合成spike的DNA,通过PCR共扩增并共测序。所有的DNA浓度测量均使用dsDNA BR Qubit试剂盒进行(Invitrogen)。所有读长均使用基因特异性数据库进行注释,并添加P,E和F合成spike的DNA序列。在原核生物门,真核生物域/门或分区水平对读长进行注释,在属和分区水平对真菌ITS读长进行注释。将微生物来源的读长合并后的数量与归因于每个合成spike的读长数量进行了比较。为了消除由不同测序深度引起的样品间差异,我们计算了每1000个总读长中合成spike读长的相对丰度。
PCR,测序及qPCR
PCR, sequencing and qPCR
所有引物都列在附加文件1中。
为了进行域特异性PCR,我们将样品分成三等分并分别用特定的引物对扩增:515F/806R(用于原核生物),F1427/R1616(用于真核生物)或ITSF1/ITSF2(用于真菌)。如前所述,使用了两步PCR系统(DI,双标记)。初始引物没有条形码,但在其5’端含有12个bp的已知序列扩增‘pads’。第一个PCR扩增靶基因,并在扩增子的两侧添加12bp的pads。PCR条件如下:高保真Phusion(0.2μl),HF缓冲液(4μl)(Thermo Scientific F520 L),dNTPs(0.4μl),引物(为每一个1μl 10μM),模板DNA(1.5 μl 5 ng/μl)和水(11.9μl)构成反应体系(20μl)。PCR条件如下:98°C持续1分钟,35个循环98 °C持续15 s,57°C持续15 s和72°C持续30 s,最终延伸步骤为72°C持续7 min。PCR产物经PCR净化试剂盒(D4014,
Zymo research)纯化,作为DNA模板进行第二轮PCR。在这一步,使用靶向DNA模板两侧的12bp pads的双条形码引物进行扩增。PCR条件相同,不同之处在于将循环数减少至25,并将退火温度提高至61°C。这些参数是根据退火温度梯度PCR试验确定的。汇集最终的PCR产物,并用PCR净化试剂盒进行纯化。将样品送至MR DNA实验室(Molecular Research LP, Texas, UK) ,通过Illumina Miseq 300PE进行测序。测序数据使用定制的Linux和Python脚本进行处理,并辅以FASTX-toolkit的脚本(http://www.hannonlab.cshl.edu)。
使用iQ SYBR Green Supermix(BioRad),模板DNA(5 ng)和先前使用的细菌16S rRNA和真核18S rRNA基因引物进行定量PCR。将反应体系在thermocycler (CFX96, BioRad)中于95°C孵育5分钟,然后以94°C 15 s,57°C(16S)或64°C(18S)30s 72°C 30s的条件下进行40个循环。三个技术重复显示出高重复性(平均SEM <平均值的0.6%),因此仅进行了生物学重复(n = 10)。根据细菌和真核生物的Ct值计算它们的比例。
Data analysis
数据分析
对于每个实验,根据PBS和条形码序列,将约1 M的双端读长进行比对和分箱。根据Usearch10 流程使用unoise3算法,通过zero-radius OTUs对Reads进行binned,使用挑选的 SILVA、PR2和ITSone数据库分别对原核生物16S rRNA、真核生物18S rRNA和真菌ITS进行注释。将PEF合成spikes的DNA序列添加到数据库中。
根据注释,将读长分配到合成物和微生物(域/门水平)中,剩下的未分配的作为singleton OTUs或大小不正确的读长,包括嵌合体和测序错误。仅对16S rRNA的252-254 bp的读长(读长的99.4%)、18S rRNA的210 -212 bp的读长(读长的93.4%)和ITS扩增子的250-300 bp的读长(读长的98.4%)进行了分析。我们剔除了测序深度较低的样本(每个样本群组的平均读数不到40%),以及结果与其他生物重复的数量级不同的样本。由于这些原因,5个原核生物样本和4个真核生物样本被移除了。我们假设这可能是由于移液或PCR错误引起的,尤其是在这个过程的第一个循环中。此外,由于实验室E. coli污染严重,还去除了八个原核样品。为了清楚起见,我们在附加文件3和附加文件4:图S1和附加文件5:图S2中展示了使用完整数据集获得的结果。
在培养对照实验中,共从78个样品中移除15个样品,因为它们显示出的合成物和微生物的读长数之比至少比平均值高出2.5倍,(基于三次生物重复的计算)。
多维标度图(MDS)
Multidimensional scaling plots (MDS)
使用zero-radius OTU(无PEF合成spikes和singletons)的系统发育数据被标准化,通过平方根进行转换,并使用PRIMER 6软件(PRIMER-E, Plymouth)基于Bray-Curtis差异矩阵在MDS图上进行可视化分析。这是为了研究添加PEF合成spike DNA是否对微生物的系统发育结构产生任何影响,并直观地表示两种不同土壤微生物群之间的差异。
结果
嵌合spiking可准确估算微生物的丰度
Chimeric spiking gives an accurate estimation of microbial abundance
我们采用一系列已知和未知数量的微生物DNA的采样策略,对定量宏基因组学的准确性进行了测试(图. 2)。
将P合成的spikes以不同比例与1.11 × 109个Rhizobium细胞混合,并提取和共扩增其DNA(图. 2a)。由于该Rhizobium的基因组具有三个拷贝的16S rRNA基因,因此预期结果是每1 ml培养物产生3.33 × 109个16S rRNA的拷贝。在三个最高水平的P spikes下,估计的Rhizobium基因含量(每1 ml培养物3.11 × 109, 3.44 × 109 and 3.38 × 109个 16S拷贝)的平均值为实际数量的99.3%(图. 2a)。较低的spike水平似乎不足,因为它们低估了Rhizobium的丰度(每1 ml培养物2.00 × 109,
1.04 × 109个16S拷贝)。
此外,为了检查是否可能因土壤的存在而改变,在已经与土壤混合的Rhizobium培养物中添加了P spikes(图. 2a)。可以看出,对Rhizobium rRNA拷贝数的估计更具可变性:每1 ml培养物1.38 × 109, 1.66 × 109, 9.19 × 108,
2.04 × 109 and 3.67 × 109个16S rRNA 拷贝图. 2b)。然而,它们与从纯培养物中得到的结果相似(图2a),这表明添加土壤并不会强烈干扰P合成spike土壤DNA的共提取和PCR共扩增。土壤颗粒可能会吸收一些Rhizobium,其中的腐殖酸可能会干扰PCR中的聚合酶,并引入多样化的微生物群落,从而使测序更加困难。可能有一些根瘤菌细胞附着在土壤颗粒上,然后它们的DNA没有完全提取出来。在本实验中,土壤微生物群估计包含:每克土壤7.6 × 108, 8.7 × 108, 6.3 × 108, 1.7 × 109 及 2.2 × 109 个16S
rRNA 拷贝。通过单独的实验(仅向土壤中添加P spikes),得出类似的1.1×109 个16S拷贝的估计值 (图. 2c)。因此,spiking方法允许在添加或不添加大量Rhizobium细胞的情况下,重复估算土壤微生物群的丰度。
接下来,将spikes添加到已经提取出的细菌和/或土壤DNA中(图. 2d–f)。在已经提取出的微生物DNA中添加P合成spike会导致我们对16S rRNA基因拷贝数的估计降低。来自培养的Rhizobium的DNA平均估计为每1毫升培养物有1.16 × 109个16S rRNA拷贝(图. 2d) ,来自培养的Rhizobium的DNA被添加到土壤中实现每1毫升培养物可以得到9.2 × 108个Rhizobium16S拷贝(图. 2e)。与Rhizobium 培养物混合测定时,土壤DNA的平均16S rRNA基因拷贝数估计为每1 ml培养物1.17 × 109个,单独测定时,每1 ml培养物中含有平均估计为1.96 × 109个16S拷贝(图. 2f)。为了获得这些值,我们测量了提取的DNA的浓度,以便计算Rhizobium和/或土壤微生物群16S rRNA拷贝的丰度。这可能会从DNA数量的估计中引入一个显著的偏差。此外,虽然16S rRNA的拷贝数在Rhizobium等已确定的物种中已知,但我们只能对总微生物群中未知的OTUs进行近似估算。为了比较起见,我们使用了Rhizobium的值,其基因组量为7.8 Mb,重40亿Da,每个基因组有3个rRNA操纵子拷贝,结果是每1 ng DNA有377,155个16S rRNA基因拷贝。根据这些结果,在DNA提取之前将spikes添加到原材料中可获得最准确的结果。
合成的spikes可以测量原位土壤DNA的丰度和不同土壤中的微生物群的组成
Synthetic spikes allow measurement of in situ soil DNA abundance and microbiota composition in different soils
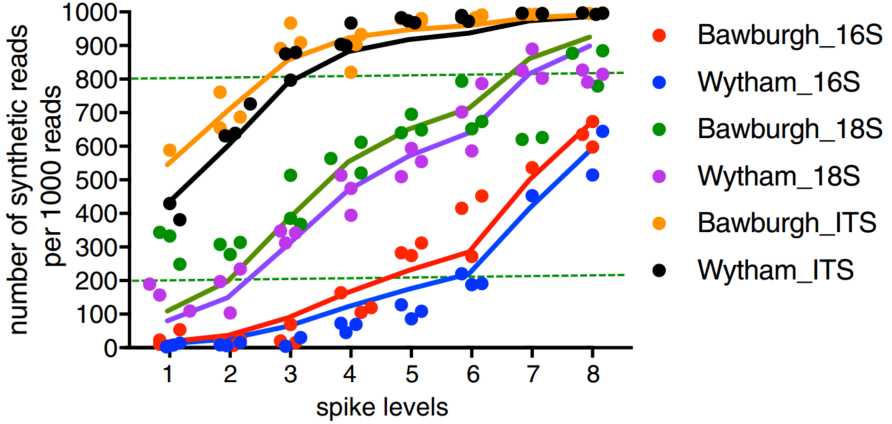
由于添加spikes可准确估计土壤微生物群的丰度,因此在两种对比土壤中测定了总原核生物、真核生物和真菌的丰度。为估算土壤微生物群落的丰度,将P(细菌16S rRNA)、E(真核生物18S rRNA)和F合成spikes(真菌ITS)分别添加到Bawburgh和Wytham土壤中(表 1)。使用P,E和F spikes的梯度来测试合成DNA和环境DNA量之间的DNA提取和/或PCR扩增的偏差。正如预期的那样,添加到土壤样品中的合成spike数量越多,它们在测序输出中的回收率就越高,尽管在最高的F合成spike水平上达到了稳定状态(图. 3)。为了确定最佳的spike水平,我们建立了一个简单的模型,显示了每1000个总序列的期望合成序列数。首先,对微生物基因丰度进行平均(图3),并使用以下方程式将该值用于建模。
其中SR1000是测序输出的每1000个读长的合成读长数。NS是每克土壤中增加的P、E或F的 spikes的数量。ENV是每克土壤中原核生物16S rRNA,真核生物18S rRNA或真菌ITS的基因拷贝数。
图 3 合成spike添加的模型
Model of synthetic spike addition
在添加不同水平的P,E和F合成的spikes到土壤中(x轴)后,16S rRNA,18S rRNA和ITS的每1000个总读长的合成spike的测序读长数(y轴)。实验结果用实线符号表示,而模型数据则用相应颜色的线条表示。该模型显示了使用特定土壤的平均基因丰度对每个spike水平在测序输出中预期的spike贡献(见表 2)。绿色虚线表示该区域每1000个读长有200-800个合成读长。x轴上的spike水平对应于表1中的合成spike水平。
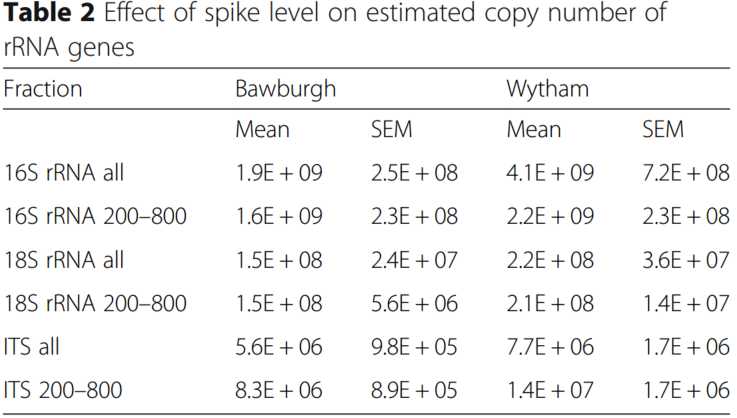
表 2 spike水平对rRNA基因估计拷贝数的影响
Effect of spike level on estimated copy number of rRNA genes
显示了从以添加水平为1-8的合成spikes的样本(表1)(所有)和对于那些符合每1000个总读长中含有> 200和<800个合成的spike读长的样本(200 800)计算出的目标基因的拷贝数。对于16S rRNA,基因200-800区域是7和8级,对于18S rRNA是3,4、5和6级,对于ITS是1和2级。
实验数据(图 3,符号)与模型集(图 3,线条)相似,表明尽管spike浓度相差100倍,但仍能准确估计微生物群丰度。在每个合成spikes的水平上,F合成spike(ITS)的每1000个总读长比E合成spike(18S rRNA)的读长多,而E合成spike(18S rRNA)的读长比P合成spike(16S rRNA)的读长多(图. 3)。不出所料,在土壤中原核16S rRNA比真核18S rRNA基因丰富,而真核18S rRNA基因比真菌ITS更丰富。此外,添加合成的spikes(水平 1–8,表1)并没有导致原核生物、真核生物和真菌群落的测量组成发生显著变化(图. 4a–c)。为了清晰起见,从图4中删除了两个显示Annelida DNA过表达的真核生物样本的数据点。这两个土壤样本可能被一条蚯蚓污染了。但是,组织的大小不足以改变检测到的16S rRNA和ITS的数量或其系统发育概况。
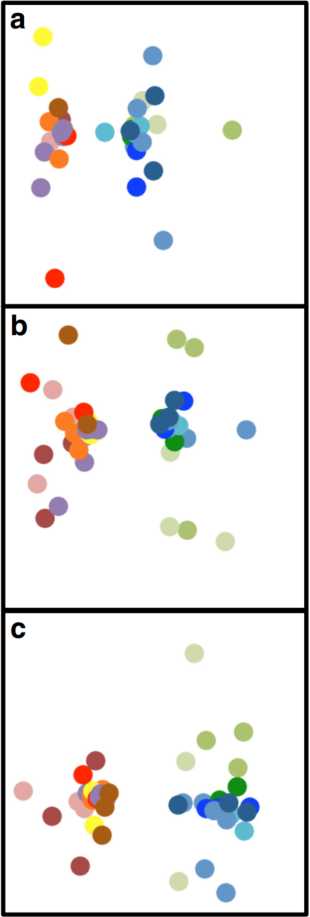
图 4 微生物群落结构的MDS图
MDS plots of microbial community structure
a原核生物b真核生物和c真菌的群落。Bawburgh(暖色)和Wytham(冷色)土壤的微生物群落。每种土壤使用八种不同的颜色,每种颜色代表不同的合成spike水平(1–8, 表 1)
测定两种对比土壤中的域丰度
Determining domain abundance in two contrasting soils
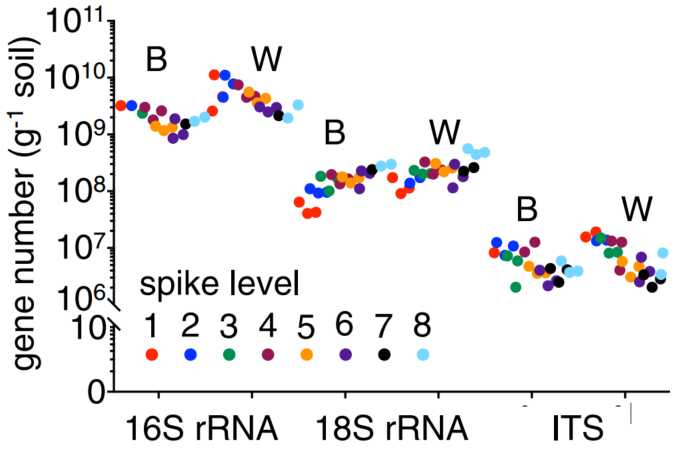
由于已知添加到土壤中的合成spike DNA的量,可以计算出各个基因(16S rRNA,18S rRNA或ITS)在土壤中的原位丰度(图. 5)。与Bawburgh土壤相比,在Wytham土壤中每个合成的spike水平都有相对更多的16S rRNA,18S rRNA和ITS基因。
图 5 利用合成的spikes对Bawburgh (B)和Wytham (W)土壤中原位微生物基因的丰度进行了估计
Estimated abundance of in situ microbial genes in Bawburgh (B) and Wytham (W) soils using synthetic spikes
土壤丰度计算方法如下:微生物rRNA丰度=(微生物数量-原始读长数/原始合成读长数量*对于带有P合成spike的16S rRNA,带有E合成spike的18S rRNA和带有F合成spike的ITS在DNA提取之前添加到样品中的合成spike的拷贝数。y轴上每克土壤的基因拷贝数被作图对应于x轴上的合成spike的等级(1–8, 表 1)。
在100倍的spikes浓度情况下,16S rRNA、18S rRNA和ITS的原位总丰度分别出现了4倍、6倍和7倍的变化(图. 5)。在每种情况下,省略每1000个总读长中少于200个和大于800个spike读长的样品(图3中的绿色虚线)(即非常低或非常高的合成spike的水平)可将变异降低到大约1.2到1.3倍(图. 5)。符合这些标准的样本为16S rRNA的7和8等级;18S rRNA的3、4、5、6等级;以及ITS的1、2等级(表 2)。
仅使用满足这些标准的样品(表 2),Bawburgh土壤的原核生物16S rRNA丰度估计值就从1.9 × 109降低到1.8 × 109,而Wytham土壤的估计值则从4.9 × 109降低到2.5 × 109。对于Bawburgh和Wytham土壤真核18S rRNA基因的丰度几乎保持不变,分别为1.5–1.7 × 107和2.5–2.3 × 108 ,而真菌ITS分别从5.8 × 106和8.0 × 106 增加到9.8 × 106和1.6 × 107。真菌spike等级3-8被合成的spikes饱和,并且原位微生物ITS的丰度不足(表 2)。根据扩增目标(即16S rRNA、18S rRNA或ITS)调整合成spike水平的需求与这些群组中每组的原位微生物DNA的总丰度相关。我们建议使用浓度大于(预期)环境微生物基因丰度20%的合成spike的浓度水平。合成spikes(> 80%)的过饱和会增加所需的测序深度,也可能会导致结果偏差。在每种情况下,Wytham土壤中的微生物群都比Bawburgh土壤中的微生物群丰富,分别在原核生物,真核生物和真菌中显示出多25%,29%和40%。这些结果与Wytham土壤中有机质和大量营养素含量高于Bawburgh土壤有关。
我们观察到基于P和F spike等级增加的微生物群落丰度估计中的梯度。一般情况下,添加的spikes越多,记录到的微生物区系丰度越低。对于20-80%比率区域内的18S rRNA以及16S rRNA和ITS数据集,我们未观察到这种关系(图. 5)。这表明低水平的spikes可能保留在土壤中,因此土壤类型可能会导致微生物群估计产生显著偏差,而非常高的水平会使DNA库过饱和。
绝对定量改变了微生物群落的系统发育结构
Phylogenetic structure of the microbiota is altered by absolute quantitation
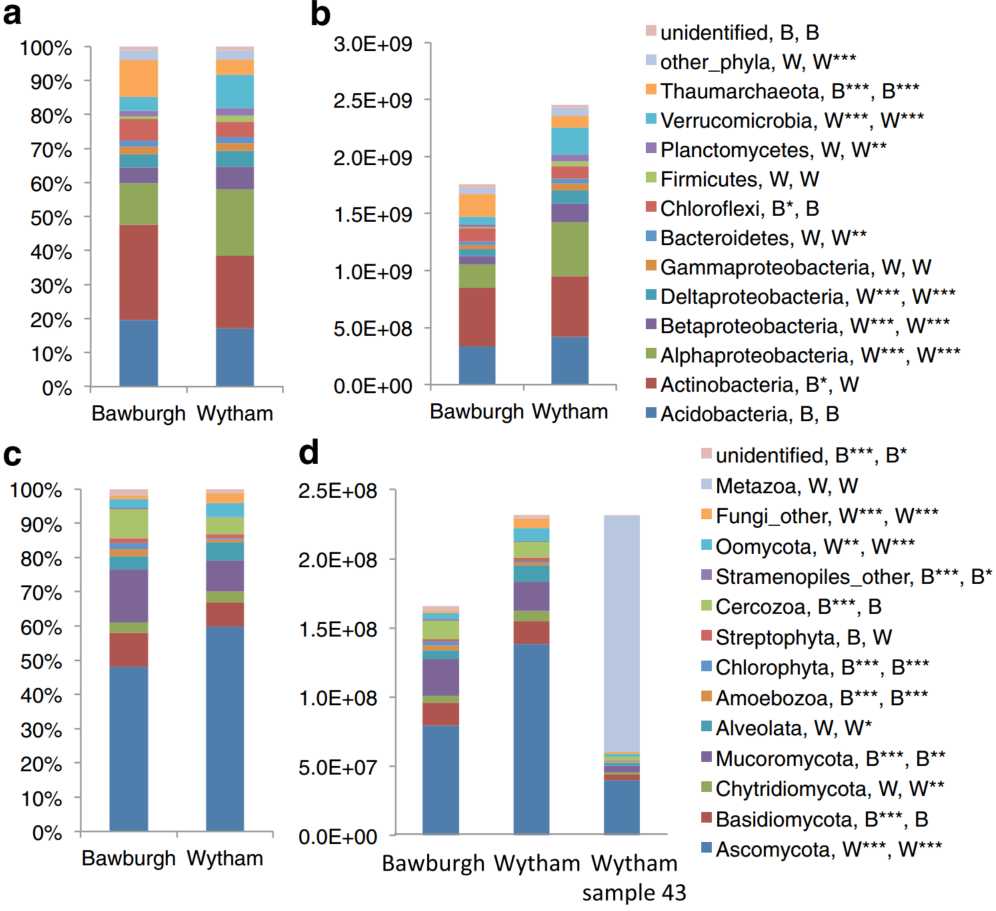
根据原核生物16S rRNA,真核生物18S rRNA和真菌ITS序列,为来自Bawburgh和Wytham的土壤样品构建微生物系统发育谱(图. 5)。与Bawburgh样本(大约2.5 × 109)相比,Wytham土壤中的原核生物更丰富(大约1.8 × 109)。我们比较了微生物群落结构的相对(图. 6a, c 和 图. 7a, c)结果和定量结果(图. 6b, d a和 图. 7b, d)。Proteobacteria,Actinobacteria和Acidobacteria是Bawburgh和Wytham土壤中的优势门(图. 6a, b)。这种原核生物的特征常见于中等pH值的土壤中。使用相对方法(图6a),这些土壤之间的主要差异是Bawburgh土壤中的Actinobacteria,Chloroflexi和Thaumarchaeota的丰度较高,而Wytham土壤中的Alpha-, Beta-,Delta-proteobacteria,Planctomycetes和Verrucomicrobia的丰度较高。然而,当进行定量校正时(图6b),除了Thaumarchaeota与Wytham土壤有关外,Bawburgh土壤中已不再富集任何门,而Wytham土壤则进一步富集了Bacteroidetes。这一致于Wytham土壤比大部分群落更丰富的Bawburgh土壤更肥沃。
图 6 Bawburgh和Wytham土壤中的原核和真核生物的群落结构
Prokaryotic and eukaryotic community structure in Bawburgh and Wytham soils
a 相对原核生物16S rRNA基因丰度。b绝对原核生物16S rRNA基因丰度。c相对真核生物18S rRNA基因丰度于d绝对的真核18S rRNA基因丰度,另外提供了Wytham土壤样品。’Wytham_43’中 存在高比例的大型有机体(Annelida)组织。Bit-score低于300的读长被归为low-blast组。’B’和’W’分别表示在Bawburgh或Wytham土壤中的类群更为丰富。* <0.05,** <0.01,*** <0.001,使用t检验和Bonferroni校正进行多重比较。
图 7 Bawburgh和Wytham土壤中的真菌群落结构
Fungal community structure in Bawburgh and Wytham soils
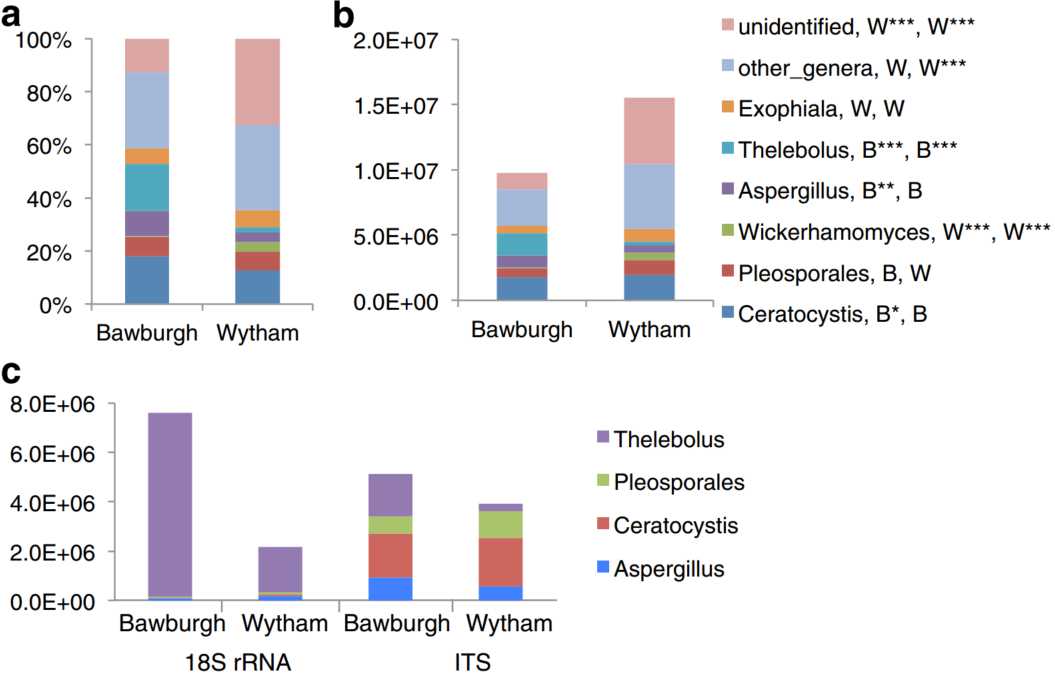
a相对真菌ITS丰度。b在属水平上显示的绝对真菌ITS丰度。c分区水平的相对真菌ITS丰度。d分区水平的真菌绝对ITS丰度。e基于18S rRNA或ITS的选定真菌属绝对丰度的比较分析。Bit-score低于300的读长被归为low-blast组。’B’和’W’分别表示在Bawburgh或Wytham土壤中的类群更为丰富。* <0.05,** <0.01,*** <0.001,使用t检验和Bonferroni校正进行多重比较。
真菌,尤其是Ascomycota, Basidiomycota 和
Mucoromycota,在Bawburgh和Wytham土壤中均占据着真核生物群落的主导地位。定量分析表明,在Wytham土壤中,Ascomycota和Chytridiomycota的优势种更加丰富,而Mucoromycota在Bawburgh土壤中占优势(图. 6c, d)。其中一个土壤样本(Wytham_43,图6d)包含一块Annelida组织,其中超过70%的测序读长属于该群组。可以预测,大量动物组织增加了18S rRNA拷贝的丰度,超过了Wytham土壤的平均值。
深入了解引物特异性
Insight into primers specificity
使用18S rRNA和ITS引物获得的系统发育谱和微生物群定量之间存在明显差异。真菌分别占Bawburgh和Wytham土壤中基于18S rRNA的真核生物群落总数的77%(相当于每1 g土壤中有1.3 × 108个基因拷贝)和79%(相当于每1克土壤中有1.8 × 108个基因拷贝)。但是,基于ITS的分析表明,真菌的丰度远低于18S rRNA分析所表明的结果,这些土壤的真菌丰度分别为8.3 × 106和1.4 × 107。18S rRNA引物过度代表了真菌种类的丰富程度,和/或ITS引物无法捕获完整的真菌分类,或者这两种方法均产生一定的偏差。并不奇怪,基于ITS和18S rRNA的分析表明常见真菌属的绝对和相对丰度有所不同(图. 7e)。已知18S rRNA引物无法揭示真菌分类学中的细节;但是,这两种引物对的结果在土壤与土壤的比较上是一致的,其中Wytham土壤中的Ceratocystis,Pleosporales和Aspergillus含量较高,而Bawburgh土壤中的Thelebolus含量较高。
跨域比较
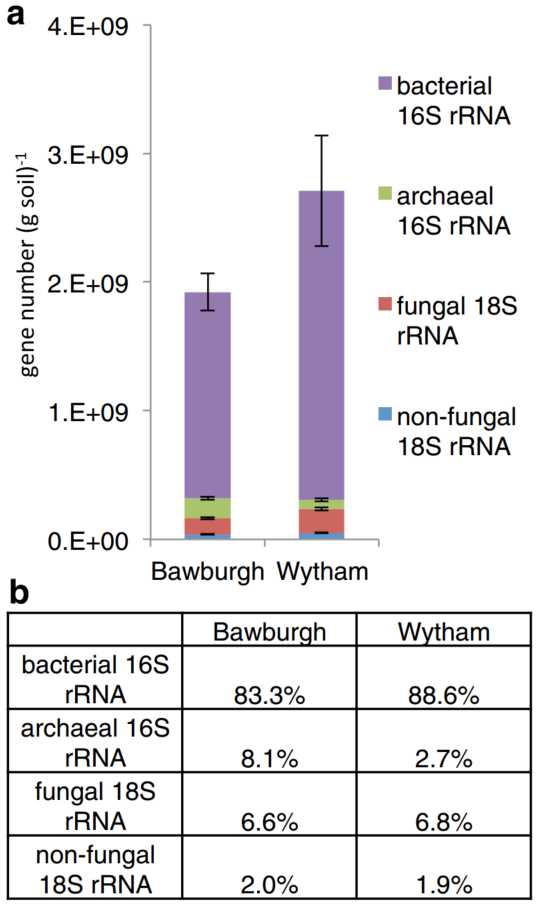
Cross-domain comparison
使用不同引物对的扩增反应可能不会针对所有潜在的微生物类群。使用的16S rRNA引物可能并不针对所有原核生物,而18S rRNA引物可能并非针对所有真核生物。但是,假设这些引物捕获了大多数群落,则可以确定原核生物与真核生物核糖体含量的比率(图. 8)。原核生物占土壤微生物群的91.4%,真核生物占8.6%。我们方法的局限性在于我们不能轻易地将结果与基于显微镜的计数相比较,因为原核细胞通常只有几个核糖体操纵子,而真核生物可能有数百个拷贝。与Bawburgh土壤相比,Wytham土壤的16S rRNA和18S rRNA含量更高。然而,这两种土壤的比率是相似的(图. 8)。这可能代表了一种控制(休耕)土壤中域间关系的生物学机制。
土壤中绝对和相对微生物群落丰度的比较
Comparison of absolute and relative microbiota abundance in soil
a绝对细菌和古菌16S rRNA和真菌和非真菌18S rRNA基因丰度及b其相对丰度
为了证实我们的发现,我们使用qPCR对从Bawburgh土壤中提取的DNA进行了检测。通过比较16S和18S rRNA基因靶标的相对扩增效率,我们计算出真核生物占土壤微生物群落的7.6%(最大 17%, 最小 4%),原核生物占92.4%(最大 96%, 最小 83%)。这些值与我们基于spike的方法的结果只有1%的差别。
我们的值也可以与一项转录组学研究结果进行比较,该结果表明原核16S rRNA和真核18S rRNA分别占土壤微生物群的91.5和2.8%(检测到的剩余6.1%的RNA无法分配)。一项比较土壤中真菌与细菌比例的研究发现,真菌对土壤的贡献率为0.6-4.2%,这取决于土壤和检测方法,如脂肪酸分析、RNA-seq和meta-蛋白组学。我们的研究中真菌与细菌的比例更高,在Bawburgh和Wytham土壤中分别为8.0%(真菌占总菌群的6.6%,细菌占总菌群的83.3%)和7.7%(真菌占总菌群的6.8%,细菌占总菌群的88.6%)(图. 8)。真菌与细菌的比例升高与高的土壤肥力、高的有机碳含量和缺乏耕作有关。这项研究中使用的休耕土壤确实具有很高的真菌丰度,因为它们的有机物含量很高,而且几十年来没有耕种过。
讨论
微生物菌群的绝对定量对于微生物生态学的各个方面都是至关重要的,我们的方法准确地估计了细菌培养和土壤微生物菌群的数量。在几个数量级上添加到土壤中的合成spikes数与获得的序列读长数成正比。尽管重复样本之间的差异通常很小,但有些样本的误差较大,这证实了对于非常复杂的样本(例如土壤),建议大量重复样本。例如,以前我们在调查植物-土壤微生物区系的工作中,我们针对每种条件使用了二十四个生物重复样品。在这项工作中,由于需要测试一个大的合成spike水平梯度,因此对于8个合成spike梯度的每一个水平,我们减少到3个重复的数量。但是,理想情况下,对于土壤微生物群的关键分析,重复数量应大于此数量。每克土壤中检出的原核生物16S rRNA约为109,真核生物18S rRNA约为108拷贝;然而,在对比土壤中可能存在一个数量级的差异。例如,分别对于肥沃的北京土壤和贫瘠的西藏土壤这两个中国土壤的广泛研究估算细菌载量其中基于ATP浓度其分别为 8 × 108和1.9 × 108,基于流式细胞仪其为1.7 × 109and 2.5 × 108 ,基于qPCR其为6.2 × 109 and 1.6 × 109,基于磷脂衍生的脂肪酸其为1.3 ×109 and 8.1 × 108 ,而基于最低杀菌浓度其为1.4 × 1010 and 1.0 × 1010。 利用van Bammelen因子转换(0.58×土壤有机质=土壤有机碳),我们可以比较中国和我国土壤的总有机碳含量:以mg / g土壤计;北京 21.5,Wytham 9.73,Tibetan 1.50, Bawburgh 1.69。我们分别从Bawburgh和Wytham的每克土壤获得的16S和18S基因拷贝数(16s+18s)为1.8×109和2.5×109,与上述针对中国土壤的微生物群落丰度值相似。
富含有机质的Wytham土壤比贫瘠的Bawburgh土壤显示出更高的微生物核糖体丰度。原核微生物和真核微生物都是如此(图. 5)。定量分析可以对土壤微生物的丰度进行统计校正。许多微生物种类在Wytham土壤中实际上更为丰富,尽管它们在Bawburgh土壤中的相对存在率更高(图. 6和7)。我们认为,这对于微生物群研究和生态学的各个方面都至关重要,它适用于从土壤到哺乳动物肠道等所有环境的微生物群。如果没有对类群进行绝对定量,则特定微生物类群作用的潜在生理学和生态学可能会被其相对丰度所掩盖。例如,关键共生体或病原体绝对丰度的变化可能被未改变的相对丰度所掩盖,或者相对丰度可能与绝对丰度相反。这可能是因为任何特定种群的相对丰度高度依赖于大多数生物的绝对丰度。
由于实验设置允许检测大多数微生物分类,即16S rRNA和18S rRNA引物针对大多数原核生物和真核生物的多样性,因此我们能够比较这些生命域的丰度(图. 8)。这些结果与以前基于RNA的土壤微生物群的估计相一致。添加合成spikes可以准确检测Rhizobium培养物对照样品中微生物16S rRNA的存在,将spikes直接添加到环境样品中而不是分离的DNA上更为有效(图. 2)。
DNA spiking可以与其他预处理步骤相结合,例如去除残留DNA(细胞外DNA)。叠氮丙啶染料与未被细胞膜保护的DNA反应并随后阻断其PCR扩增。可以将合成的spikes添加到初始样品的等分试样中,并去除残留的DNA。这种方法将揭示哪些类群是活着的,以及它们的绝对存在。
强烈建议使用初始校准曲线来确定给定环境条件下的最佳合成spike水平。尽管所使用的合成spike的水平并没有改变所测量的微生物群落结构,但与目标菌群相比,spike的添加量过高或过低都可能使定量结果产生偏差(图. 4,5,和6)。然而,一旦进行了初始校准,spike的水平可以变化超过三个数量级,并且具有很高的重现性。需要注意的是,要确保从不同的环境中有效地提取出DNA及确保其稳定性,因为这可能会使任何定量方法产生偏差。然而,鉴于这一警告,我们的方法很简单,只需要添加已知数量的合成spike的DNA并在测序后进行单个生物信息学步骤,即可定量微生物群研究中的原核生物,真核生物和真菌的绝对丰度。
结论
活跃的微生物组的量化将有助于更好地了解环境微生物学中的功能类群,并有助于产生更好的微生物组相互作用模型。这种定量方法已广泛应用于与病害或土壤生产力相关的微生物组/宏基因组的关联研究。
Reference
Andrzej Tkacz,Marion Hortala,Philip S. Poole. Absolute quantitation of microbiota abundance in environmental samples.Microbiome (2018) 6:110 doi:10.1186/s40168-018-0491-7
相关文章
- Plant Com:绝对定量检测宿主微生物组的HA-QAP技术简介 全文解读
- Science Bulletin:上海植生所王二涛组发表基于绝对定量丰度的植物根际微生物群落“扩增-选择”组装模型
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA
今天的文章菌群相对丰度_大肠菌群检测方法分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/89030.html