
- 代谢组数据分析零:从质谱样本制备到MaxQuant搜库
- 代谢组数据分析一:代谢组数据准备
- 代谢组数据分析二:数据预处理
- 代谢组数据分析三:降维分析
- 代谢组数据分析四:差异分析
- 代谢组数据分析五:功能分析
- 代谢组数据分析六:基于报告分数的功能分析
- 代谢组数据分析七:溯源分析
- 代谢组数据分析八:其他分析
- 代谢组数据分析九:相关性分析
- 代谢组数据分析十:偏相关分析
- 代谢组数据分析十一:典型相关分析
- 代谢组数据分析(十二):岭回归、Lasso回归、弹性网络回归构建预测模型
- 代谢组数据分析(十三):评估影响代谢物的重要临床指标
- 代谢组数据分析(十四):代谢物组间网络分析(spearman coefficient)
- 代谢组数据分析(十五):基于python语言构建PLS-DA算法构建分类模型
- 代谢组数据分析(十六):代谢组缺失值处理方法评估汇总
- 代谢组数据分析(十七):基于structToolbox代谢组分析流程讲解
- 代谢组数据分析(十八):随机森林构建代谢组诊断模型
- 代谢组数据分析(十九):随机森林构建代谢组预后模型
- 代谢组数据分析(二十):通过WGCNA识别核心代谢物
- 代谢组数据分析(二十一):通过MetaboAnalystR标准化构建sPLSDA预测模型
- 代谢组数据分析(二十二):Zscore标准化后主成分分析(PCA)及热图展示
文章目录
介绍
代谢组学和蛋白质组学是系统生物学中两个重要的分支,它们分别研究生物体内的小分子代谢物和蛋白质。它们的主要区别在于研究的对象和分析的层面:
- 研究对象:代谢组学关注的是生物体内所有代谢物,主要是分子量1000以下的小分子,如有机酸、氨基酸、糖类、脂类等 3。而蛋白质组学研究的是生物体中的蛋白质,包括它们的表达、修饰、功能等 3。
- 分析层面:代谢组学可以看作是生物学的终端信息,它反映的是生物体内已经发生的事件,如基因表达变化或环境变化后的代谢产物种类和数量 5。蛋白质组学则从分子水平上分析蛋白质的存在状态和活动规律 3。
- 研究手段:代谢组学的主要研究手段包括核磁共振技术(NMR)和质谱技术,特别是飞行时间质谱和高分辨质谱 35。蛋白质组学则常用凝胶技术和质谱技术,通过蛋白酶解后得到的肽段进行分析 3。
在质谱仪和鉴定荷质比的时候,代谢组和蛋白质组的区别主要体现在:
- 样品准备:代谢组学分析的样品通常是血浆、尿液等生物液体,而蛋白质组学分析的样品可能是细胞或组织的蛋白质提取物 5。
- 质谱技术:代谢组学中常用的质谱技术包括GC-MS、LC-MS、CE-MS等,它们适用于分析小分子代谢物 5。蛋白质组学则常用LC-MS和MALDI-TOF MS等技术,特别是MALDI-TOF MS可以测量高达100,000的分子量 4。
- 荷质比(m/z):在质谱分析中,代谢物和蛋白质的荷质比不同。代谢物由于分子量较小,其m/z值通常较低,而蛋白质由于分子量较大,其m/z值较高。在质谱图中,分子离子峰的m/z值可以帮助确定代谢物或蛋白质的分子量 24。
LC-MS/MS
Liquid Chromatography-Mass Spectrometry(LC-MS/MS ,液相色谱-质谱串联)可用于残留化合物检测、有机小分子检测、鉴定和定量污染物以及在医药和食品领域添加剂检测和生物小分子等检测。
LC-MS/MS一般包含五个步骤(Figure 1):
- 样本制备;
- 样本分离:使用液相色谱方法分离;
- 质谱上机:离子化、LUMOS原理、采集模式(DDA、DIA、SRM/PRM);
- 质谱鉴定:谱图格式(Raw、MzXML、MGF)、搜库、打分算法、FDR质控、蛋白推断;
- 生信分析:谱图校验和下游数据统计分析。

步骤详解
样本制备
提取样品中的蛋白质后,再使用蛋白酶对蛋白进行酶切处理,一般处理后的肽段在35AA以内即可上机(Figure2)。常用蛋白酶是Trypsin。

样本分离
液相色谱包括固定相和流动相的一类分离技术,以液体作为流动相,固定相可以是多种类型也可以是液体也可以是固体等。Figure3 固定相是3A中圆孔材料,流动相是两类液体,液体A可将肽段插入到固定相中。在不断增加液体B的浓度后,肽段可以及液体B的浓度大小呈现梯度整齐分布,最后计算肽段在液相过程的保留时间(Retention time, RT),该指标反应肽段的疏水作用,时间越长疏水效果越强。

质谱上机
质谱仪的构成简要图(Figure 4)

质谱仪件
离子源 [Ion Sources make ions from sample molecules.]
- 基质辅助激光解吸电离(MALDI)
- 电喷雾电离(ESI)
质量分析器[Mass analyzers separate ions based on their mass-to-charge ratio (m/z)]
- 傅立叶变换离子回旋加速器(FT-MS)
- 线性离子阱(Linear ion trap)
- 三维离子阱(3D ion trap)
- 轨道离子阱(Orbitrap)
- 四极杆(Quadrupole)
- 飞行时间(Time-of-flight, TOF)
信号检测器[Ion Detector registers the number of ions at each m/z value.]
- 微通道板(Microchannel Plate)
- 电子倍增器(Electron Multiplier)
- 光电倍增混合管(Hybrid with photomultiplier)
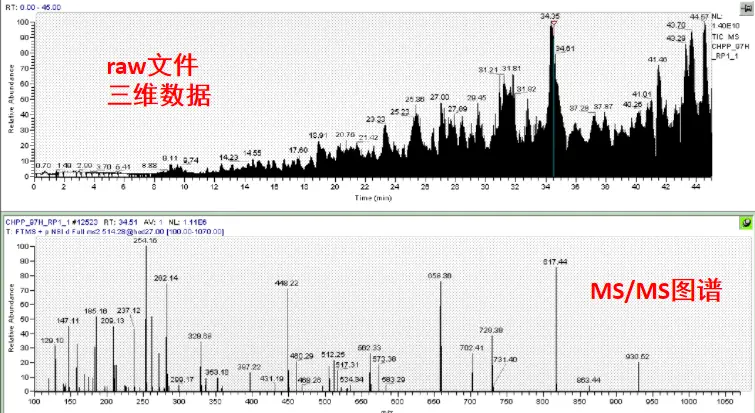
质谱图谱示意图(Figure5)图解:
- 正面主视图是液相色谱图(以Retention Time作为X轴,intensity可认为是relative abundance作为Y轴),每个峰表示RT时间相近也即是疏水作用强度相同的肽段离子;
- 侧面黑色X轴是m/Z是一级质谱图,x轴是肽段的质荷比,每一个峰表示该比值下的肽段集合;
- 侧面蓝色是上一步一级图谱同质荷比下的二级图谱,是分辨率更高的图谱结果;
- intensity可以理解为色谱和质谱的响应强度。

数据采集模式
非靶向质谱数据采集模式:Full-scan(全扫描)、DDA(数据依赖采集)和DIA(数据非依赖采集)。
- 全扫描模式是将肽段一级和二级图谱分开全部扫描,所需时间较长,但获取的峰最多。
- DDA数据依赖采集,根据全扫描的一级图谱中的一堆母离子的intensity以及动态时间去解析二级图谱,但该过程可能后漏掉携带有用信息的峰;优点:DDA采用了较窄m/z窗口,二级质谱图没有干扰离子影响,搜库鉴定容易;缺点:丰度依赖,谱图采样随机性大,重复性差。
- DIA数据非依赖采集,在一级图谱扫描过程不对碎裂母离子做筛选,而是设置窗口(窗口大小依据仪器和软件设定),落在该窗口内的母离子全部进入二级碎裂得到二级图谱。优点:全面获取所有离子的碎片信息。缺点:采集点多会导致谱图信息过度,难以解析。鉴定需要依赖于预先lable的DDA库。
由于DIA是一次性放了一堆母离子进来,同时碎裂,所以对于DIA来说,不是一张谱图对应一个母离子,而是一堆谱图对应一堆来自多个母离子的碎片离子混合物。因此,我们不可能通过对二级谱图的解析来得到一个一个的母离子。
那我们应该如何解析呢?我们用DIA二级谱图与平行实验中的DDA的二级谱图进行比对,从中抽取出相同的二级信号,拿这些数据来进行DIA数据的定性以及相应的定量,而这个定量就是依赖于MS2的信号强度。
Targeted是靶向质谱数据采集

质谱鉴定
质谱的原始图谱格式通常有:
- Thermo RAW(赛默飞二进制加密的图形文件,Thermo Xcalibur Browser软件查看);

- MGF(Mascot Generic Format,是常用格式但仅保留二级谱图信息,原始数据一般不能被肽段图谱比对打分软件识别,需要经过格式转换为打分软件可识别的格式);

- mzXML
质谱打分比对软件
图谱识别可通过De novo和Database Search两种方式实现.

搜库策略:软件根据设定理论参数对蛋白数据库模拟实验过程进行理论酶切,接着理论肽段又会生成理论图谱,最后将实验得到的图谱也就是质谱的图谱和数据库模拟出的理论图谱进行比较,最终鉴定和定量蛋白质。(Figure7)

搜库步骤
搜索引擎会对数据库里所有的蛋白序列进行理论酶切,得到肽段序列,再对肽段序列进行理论碎裂,形成理论谱图。
然后,用每一张导入搜索引擎的实验谱图与落入母离子质量误差窗口内的理论谱图进行匹配打分,并选择打分最好的理论谱图对应的肽段作为该实验谱图的鉴定结果。

搜库质控:实验图谱和理论图谱匹配不一定正确,一般需要设置p value等评估匹配结果的指标阈值
错误匹配原因:
- 蛋白质序列库不完整或者存在测序错误;
- 未知修饰,导致谱图难以被正确鉴定;
- 酶切实验的偏差,比如错切、漏切等;
- 母离子或子离子质量偏差;
- 搜索引擎的打分无法区分谱图对应的正确肽段和错误肽段,因此需要评价肽段鉴定的可靠性,搜索引擎才能根据鉴定到的可信肽段序列进行蛋白质推断。
定性和定量蛋白质
在获取质谱图谱数据后,可通过带有搜库引擎的软件进行定性和定量鉴定蛋白质。我们从仪器中心获得去质谱原始data是Thermo RAW的二进制加密文件,在一般的搜库软件是无法识别的,需要使用ThermoRawFileParser或MSconvert转换为软件可识别的文件。(FAIMS技术相当于在现有图谱分级基础上又再一次对肽段进行分级)
PS:在获取图谱过程中,常会用到2018年才推出的FAIMS(High-Field Asymmetric Waveform Ion Mobility Spectrometry)技术以用于加载不同电压(肽段在ESI离子化后,进入质谱之前实现快速气相分离,提高分离的峰容量),直接使用多电压下的raw data做MaxQuant定量分析是错误的,MaxQuant软件只能识别单电压的raw data,因此需要使用FAIMS MzXML Generator 软件将raw data转换成各自电压下的MzXML文件。
软件
格式转换软件
- ThermoRawFileParser
- MSconvert
- FAIMS MzXML Generator
搜库软件:
- 免费:MaxQuant,MSGFplus,Comet(前一个最好用win版本,后两个用linux版本,然后后两个是最好用主流的质控软件);
- 收费:Proteome Discoverer, Mascot;
- 也有基于X!Tandem算法的rTANDEM R包。
结果评估:
- PTXQC R包
- proteoQC R包
数据库
来自欧洲生物信息学中心的UniProt KB数据库是现在使用较多的蛋白质数据库。除了选择公共数据库外,还可以自行添加或者从头构建蛋白质数据库(一般会加入实验室常见污染物数据库,方便剔除)。另外,如果所研究的物种没有对应的蛋白质数据库,可选择最近物种的序列库。

实操过程
下面部分是原始质谱数据定性和定量蛋白质过程:
- 安装数据转换和搜库软件:本教程基于MaxQuant 1.6.17.0版本,该软件可从MaxQuant官网直接下载或从子目录Software直接解压获取。另外,需和仪器中心确定是否使用了FAIMS技术后,再从子目录Software中解压安装FAIMS MzXML Generator软件;

- 下载蛋白质数据库:Database子目录下已下载23/2/2021年的human的Uniprot蛋白质数据,或者直接上Uniprot官网下载(1.进入官网,选择Proteomes;2.以human作为关键字搜索后,选择Organism的Homo sapiens后进入;3.进入下图后选择Download按钮以FASTA格式下载数据即可);

- 转换原始数据格式:若使用FAIMS技术,则需要使用FAIMS MzXML Generator软件将RAW转换成40/60/80不同电压下的mzXML文件,否则跳过该步骤;

- 搜库鉴定蛋白质:我们使用免费开源的MaxQuant 1.6.17.0版本软件进行鉴定蛋白质,在运行前,需要配置好一下参数和注意以下事项:
- Raw data功能项导入图谱数据(mzXML),可通过最右侧功能栏设置样本的名称和分组等信息;

- Group-specific parameters:
- “Modifications”:选择样本相关的修饰(LabelFree增加Deamidation(NQ)、Gln->pyro-Glu);
- “Instrument”:MaxQuant会自动选择机器类型,也可以手动修改,参数选择软件默认的即可;
- “digestion”:选择默认的Trypsin/P;
- “Label-free quantification”:选择LFQ及其默认参数。

- Global parameters:(其他参数选择默认参数即可,也可根据实际情况自行设置)
- “Sequence”:选择"Add"导入蛋白质fasta文件,并点"Identifier",选择"Up to first space";
- “Identification”:选择Match between runs;
- “Label-free quantification”:选择iBAQ;
- “Folder locations”:可自行设置Combined结果的输出路径也可使用默认。

- 在设置完所有参数后,上面File选择保存parameters files,方便下次重复运行。

- 完成保存设置参数文件后,最左下方提供选择CPU数目,一般设置电脑total(CPU) -1的数目。设置完成后,可在电脑空闲时候选择Start,即可在Performance界面看到软件在运行的状态。
- Raw data功能项导入图谱数据(mzXML),可通过最右侧功能栏设置样本的名称和分组等信息;
结果
MaxQuant完成蛋白质定性和定量后,会生成如下combined目录,其子目录txt下的proteinGroups.txt文件即是蛋白质结果文件。

代谢组数据分析软件和平台

MZmine

MZmine是一个开源的、平台独立的质谱(MS)数据处理和可视化软件。它专为处理大规模质谱学研究而优化工作流程,支持多种原始数据格式,包括开放格式如mzML以及一些供应商特定的文件类型。MZmine能够处理来自不同仪器设置的数据,例如LC-MS、LC-IMS-MS、GC-MS,甚至是MALDI-(IMS-)-MS。
MZmine的主要特点包括:
- 数据处理和可视化:MZmine允许用户将原始的谱图数据转换成有意义的特征列表,并提供通过谱图库匹配、精确质量搜索、基于规则的脂质注释和公式预测等方法进行化合物注释。
- 支持多种数据格式:MZmine支持多种原始数据文件格式,如mzML、mzXML、mzData、NetCDF以及Thermo RAW、Waters RAW等。
- 批量处理能力:MZmine能够以批量模式运行多种数据处理方法,并且可以作为命令行应用程序运行,方便集成到自动化数据分析工作流程中。
- 峰值检测和列表处理:MZmine提供了多个模块用于处理峰值列表,包括填补间隙、同位素检测、过滤、对齐、标准化和鉴定。
- 统计分析:MZmine包含基本的统计分析方法,处理后的数据也可以轻松导出到第三方统计软件进行高级分析。
- 数据可视化:MZmine可以可视化原始数据以及峰值检测和鉴定结果,这对于评估不同的峰值检测方法和获取数据概览非常有用。
XCMS/R 和 XCMS Online

XCMS/R
XCMS/R 是一个基于 R 语言的开源软件包,专门用于非靶向代谢组学数据的预处理、对齐和定量分析。它提供了多个数据处理功能,包括峰识别、去噪、峰对齐等。XCMS/R 适合于需要进行大规模代谢组学数据预处理的用户,并且可以整合其他的 R 包进行多变量统计分析和绘图、聚类分析等。由于 XCMS/R 是命令行驱动的,因此需要用户具备一定的 R 语言基础。
XCMS Online
XCMS Online 是 XCMS 的网络版本,它是一个基于云的信息平台,旨在处理和可视化基于质谱的非目标代谢组数据。与 XCMS/R 相比,XCMS Online 不需要命令行环境,也不需要用户具备编程知识,使得非专业人员也能轻松使用。XCMS Online 支持多种实验方案数据的分析,包括相关(配对)两组比较、荟萃分析和多组比较,并且可以进行单变量分析和 PCA 分析。用户只需上传原始的质谱数据并选择参数,结果就会自动生成,非常便捷。XCMS Online 还整合了 METLIN 数据库,方便物质定性无缝连接,并且可以进行 pathway 分析,整合蛋白数据和基因数据
MetaboAnalyst

MetaboAnalyst 是一个基于网络的综合性工具套件,旨在帮助用户轻松地进行代谢组学数据分析、可视化和功能注释。它从最初的针对靶向代谢组学数据的统计和功能分析,发展到现在能够更流畅地处理非靶向LC-MS数据。MetaboAnalyst 提供了多种功能,包括:
- 统计分析:提供多种统计和机器学习方法,包括单变量方法(如fold change、t-test、volcano plot、ANOVA、相关分析)和多变量方法(如PCA、PLS-DA或OPLS-DA),以及聚类分析和有监督分类。
- 富集分析:执行代谢物集富集分析(MSEA),包含人类和哺乳动物代谢物集,以及化学类代谢物集。该模块接受化合物名称列表、化合物名称和浓度列表或浓度表。
- 通路分析:支持代谢通路分析,整合了通路富集分析和通路拓扑分析,提供超过120个物种的视觉探索,并允许用户进行联合通路分析。
- 生物标志物分析:提供基于接收器操作特征(ROC)曲线的方法,用于识别潜在的生物标志物并评估其性能。
- 网络分析:用户可以上传代谢物、基因或KEGG同源基因的列表,并在生物网络中视觉化探索这些分子。
- 统计分析:用户可以上传多个在相似条件下收集的注释代谢组学数据集,以识别跨多个研究的稳健生物标志物。
- LC-MS 光谱处理:用户可以上传LC-MS光谱并执行峰值提取、对齐和注释。
- MS/MS峰值注释:基于公共MS2数据库列表执行MS2峰值注释
metaX Pipeline

metaX Pipeline 是一个基于 R 语言开发的代谢组学数据分析软件包,它能够通过一组可互换的模块进行端到端的代谢组学数据分析。具体来说,metaX 提供了以下功能:
- 峰值拾取和注释:metaX 可以处理 mzXML 文件或峰值表文件作为输入,使用 R 包 XCMS 进行峰值检测,并使用 CAMERA 包进行峰值注释。
- 数据质量评估:metaX 提供了基于网络的界面(http://metax.genomics.cn)用于数据质量评估和归一化方法评估,并生成带有可视化界面的 HTML 报告。
- 缺失值插补:在预处理原始峰值强度数据时,metaX 实现了四种缺失值插补方法:k-最近邻(KNN)、贝叶斯主成分分析替换(BPCA)、svdImpute 和随机森林插补(missForest)。
- 数据归一化:metaX 提供了五种不同的数据缩放方法:Pareto 缩放、vast 缩放、范围缩放、自动缩放等。
- 单变量和多变量统计:metaX 能够进行单变量和多变量统计分析,包括功率分析和样本量估计。
- 接收器操作特征分析:用于生物标志物选择和性能评估。
- 路径注释:metaX 可以进行代谢物的路径注释分析。
- 相关网络分析:metaX 还提供了基于网络的相关性分析,以识别代谢物对之间关系的显著变化。
- 代谢物鉴定:metaX 支持代谢物的鉴定
Compound Discoverer 3.2
Compound Discoverer 3.2 (提供了试用版本) 是由Thermo Scientific提供的一款代谢组分析软件平台,专门用于小分子研究的定性数据处理。它集成了多种工具,能够处理和分析高分辨率质谱数据,尤其在代谢组学研究中应用广泛。以下是该软件平台的一些主要功能和特点:
- 化合物识别与结构分析:Compound Discoverer 3.2能够通过MS/MS数据库识别化合物,使用高分辨率MS全扫描同位素及MS/MS数据确定素组成,并实现化学数据库在线检索功能。
- 统计和差异化分析:软件提供了t-tests、ANOVA、PCA、PLS-DA、火山图、箱线图等统计和差异化分析工具,帮助用户识别样品集之间重要的统计学差异。
- 数据可视化:Compound Discoverer 3.2可以查看趋势图来可视化峰面积、各组平均峰面积或编入随时间变化分数标记的平均峰面积,以及查看峰面积的热图,CV值,调整后的p值等。
- 生物学通路分析:软件支持进行稳定性同位素标记实验,并使用Thermo Scientific Metabolika、KEGG及BioCyc数据库查看通路,匹配检测到的化合物、统计数据及编入通路中的分数标记。
- 广泛的应用支持:Compound Discoverer 3.2支持多种应用,包括杂质与降解产物研究、环境与食品安全应用研究、代谢物鉴定、法医毒理学研究和代谢组学研究。
- 工作流程定制:软件提供可定制的功能实现强大的数据处理工作流程,用户可以将工作流程节点从处理工具列表拖放到树形工作流程中,指定所需的数据流程。
- 新功能增强:Compound Discoverer 3.2版本中包含了新功能,如GC-Orbitrap数据处理能力、中性损失搜索、自定义标签、从SDF文件导入结构、从Excel电子表格导入研究信息、Apply Missing Value Imputation节点等。
- mzVault和mzCloud集成:软件允许使用mzCloud光谱库进行未知化合物的识别,并且可以与mzVault软件配合使用,创建、编辑和转换光谱库。
Transform your small molecule data, whether a small or large dataset, from liquid chromatography (LC), gas chromatography (GC), ion chromatography (IC), full-scan accurate mass, or MSn data, into actionable insights with Thermo Scientific Compound Discoverer Software. Compound Discoverer Software offers a fully integrated suite of advanced tools for both known-parent and unknown data processing and interpretation.
This powerful software streamlines compound identification, comparative analyses and provides extensive filtering and data visualization capabilities. With its easy-to-use workflows, integrated libraries, databases and robust statistical analysis tools, it enables rapid and precise insights from your valuable data.
Whether your research focuses on metabolomics to stable isotope labeling, environmental and food safety, pharma metabolite or impurity identification, extractables and leachables to forensic or clinical toxicology, Compound Discoverer Software offers an exceptional toolbox to drive precise and efficient data analysis in small molecule-research.
Compound Discoverer 3.2 tutorial

致谢
感谢张同学seminar分享的PPT,本文很多图片或知识点来自于他的PPT,再次感谢。
参考
- A Critical Review of Trypsin Digestion for LC-MS Based Proteomics
- Peptide retention time prediction
- 蛋白质组学数据分析基础3
- 2019-11-12p值、E值、FDR、q值…你晕菜了吗?
- MGF Files (MS/MS container files)
- ThermoRawFileParser
- proteoQC
- MaxQuant – Information and Tutorial
- Bioconductor的质谱蛋白组学数据分析









版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/bian-cheng-ji-chu/92001.html