微信公众号:生信小博士
单细胞——从降维聚类分群、细胞命名、到批量富集分析,一文打通GSE博来霉素小鼠模型单细胞数据
R语言|12. 森林图-1: 多因素Cox回归模型森林图 (基于forestplot包)
本期开始介绍Cox回归模型可视化。
之前学习过的的临床回归模型可视化方法主要是森林图和列线图 (Nomogram)。
计划是介绍一下单因素、多因素、亚组分析、其他类型森林图绘制;列线图部分介绍下基本的变量筛选方法、绘制过程、验证方法(ROC/DCA/校准图/分组比较等)、网页版列线图。
本期介绍forestplot包绘制多因素Cox回归模型森林图。
此外,本期在提取Cox回归等结果信息用到了之前介绍过的R语言|7. 快速制作多因素Cox回归三线表 和R语言|4. 轻松绘制临床基线表Table 1,需要的小伙伴点击链接阅读。
先看效果图
多因素Cox回归森林图
载入R包和数据
#1.载入包 library(tableone) library(survival) ##画森林图的包 library(forestplot) library(stringr) #2.清理工作环境 rm(list = ls()) #3.读入数据 aa<- read.csv('.csv') #4.查看数据前6行 head(aa) #5.查看数据数据性质 str(aa) #6.查看结局,0=复发,1=未复发 aa$status<-factor(aa$status) summary(aa$status) 一、构建森林图的基本表
#一-1.cox多因素回归分析 mul_cox<-coxph(Surv(time,status==0)~Age+T.stage+LNM+ER+HER2+G,data=aa) #一-2 multi1:提取:变量+HR+95%CI+95%CI mul_cox1 <- summary(mul_cox) colnames(mul_cox1$conf.int) multi1<-as.data.frame(round(mul_cox1$conf.int[, c(1, 3, 4)], 2)) #一-3、multi2:提取:HR(95%CI)和P multi2<-ShowRegTable(mul_cox, exp=TRUE, digits=2, pDigits =3, printToggle = TRUE, quote=FALSE, ciFun=confint) #一-4.将两次提取结果合并成表;取名result result <-cbind(multi1,multi2);result #一-5.行名转为表格第一列,并给予命名"Characteristics" result<-tibble::rownames_to_column(result, var = "Characteristics");result result表,6列表还缺人群数目列,参考变量,后面慢慢修改
二、森林图的基本图形fig1
fig1<- forestplot(result[,c(1,5,6)], #告诉函数, 合成的表格result的第1,5,6列还是显示数字 mean=result[,2], #告诉函数,表格第2列为HR,它要变成森林图的小方块 lower=result[,3], #告诉函数表格第3列为95%CI, upper=result[,4], #表格第5列为95%CI,它俩要化作线段,穿过方块 zero=1, #告诉函数,零线或参考线为HR=1即x轴的垂直线 boxsize=0.3, #设置小黑块的大小 graph.pos=2) #森林图应插在图形第2列 基本图形
三、森林图图形修饰
修饰项目
三-1、显示所有变量;
三-2、加入患者数目;
三-3、加水平线、垂直辅助线、x周标签、大标题、森林图占比;
三-4、字体大小增粗、线条粗细、线型(包括置信区间)、行间距、列间距;
三-5、颜色、HR方块形状等。
其中,1.2是对表的修改,改完表,图就改了;3-5步是通过改字体、线宽等优化的。这5项在我们掌握后可按照自己需要随意组合使用。
三-1、显示所有变量
#1.删除部分变量名,只保留亚变量即:"ER_Positive"变为"Positive" result$Characteristics<-str_remove(result$Characteristics,"T.stage|N|ER|HER2|G") #2. 给参考变量插入空行 #2-1.这步代码不用改 ins <- function(x) {c(x, rep(NA, ncol(result)-1))} ##2-2:插入空行,形成一个新表 for(i in 5:6) {result[, i] = as.character(result[, i])} result<-rbind(c("Characteristics", NA, NA, NA, "HR(95%CI)","p"), result[1, ], ins("T stage") ins("T1"), result[2, ], ins("N"), ins("0"), result[3:4, ], ins("ER"), ins("Positive"), result[5, ], ins("HER2"), ins("Positive"), result[6, ], ins("G"), ins("I"), result[7:nrow(result), ], c(NA, NA, NA, NA, NA,NA)# ) for(i in 2:4) {result[, i] = as.numeric(result[, i])} 显示所有变量的表
fig2 <- forestplot(result[,c(1,5,6)], mean=result[,2], lower=result[,3], upper=result[,4], zero=1, boxsize=0.5, graph.pos=2) 显示所有变量名的图fig2
三-2、显示所有亚组的患者数
#1-1 myVars <- c("Age","T.stage","LNM","ER","HER2","G") #1-2 catVars <- c("T.stage","LNM","ER","HER2","G") #1-3 table1<- print(CreateTableOne(vars=myVars, data = aa, factorVars = catVars), showAllLevels=TRUE) #2. 在基线表table1里插入空行,使它的行数和变量跟result一致 N<-rbind(c(NA,NA), table1[2, ], c(NA, NA), table1[3:4,], c(NA,NA), table1[5:7,], c(NA,NA), table1[8:9,], c(NA,NA), table1[10:11,], c(NA,NA), table1[12:14,], c(NA, NA)) N<-N[,-1] N<-data.frame(N) #3.把N表和result表合在一起 result1<-cbind(result,N) #调顺序。变为:变量-N-HR......顺序 result1<-result1[,c(1,7,2:6)] #4.优化第一行。第一行行名中加入"Number(%)" for(i in 2:7) {result1[, i] = as.character(result1[, i])} result1<-rbind(c("Characteristics","Number (%)",NA,NA,NA,"HR (95%CI)","P.value"), result1[2:nrow(result1),]) for(i in 3:5) {result1[, i] = as.numeric(result1[, i])} 显示人群数目列
#画图fig-3,注:因为多了一列,所以要注意改代码数字 fig3<-forestplot(result1[,c(1,2,6,7)], mean=result1[,3], lower=result1[,4], upper=result1[,5], zero=1, boxsize=0.4, graph.pos=3) 11.png
三-3、图形细节优化
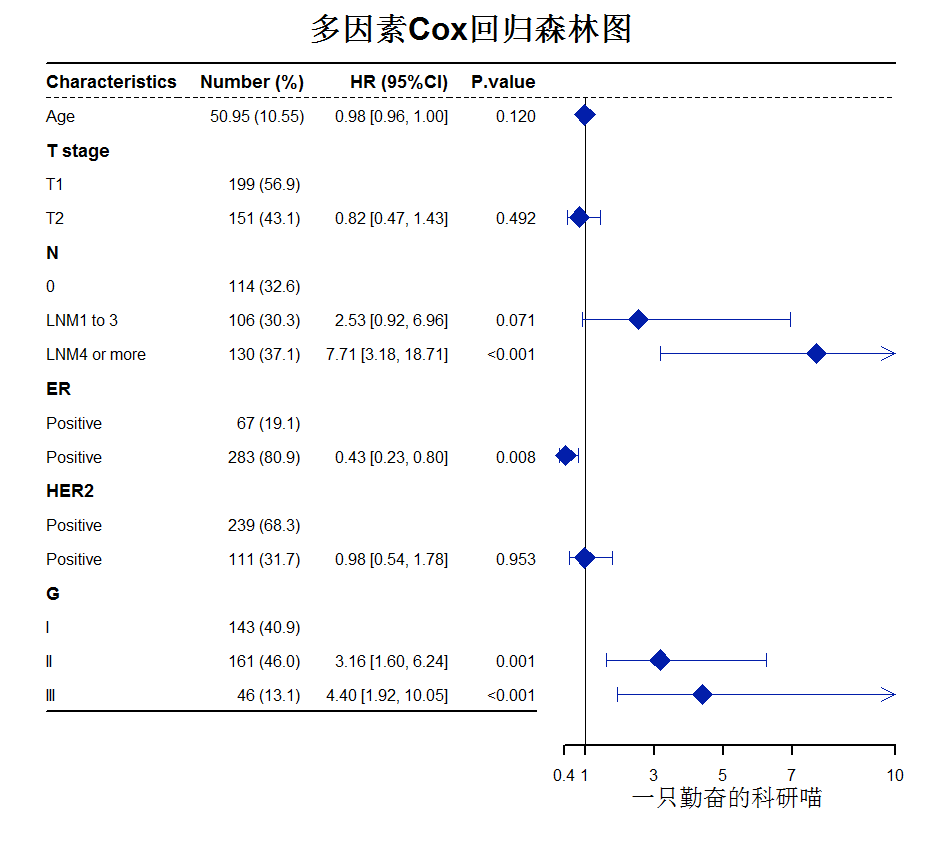
fig3_1<-forestplot(result1[,c(1,2,6,7)], mean=result1[,3], lower=result1[,4], upper=result1[,5], zero=1, boxsize=0.6, graph.pos= "right" , hrzl_lines=list("1" = gpar(lty=1,lwd=2), "2" = gpar(lty=2), "20"= gpar(lwd=2,lty=1,columns=c(1:4)) ), graphwidth = unit(.25,"npc"), xlab="一只勤奋的科研喵", xticks=c(0.4,1,3,5,7,10) , is.summary=c(T,F,T,F,F,T,F,F,F,T,F,F,T,F,F,T,F,F,F), txt_gp=fpTxtGp( label=gpar(cex=1), ticks=gpar(cex=1), xlab=gpar(cex=1.5), title=gpar(cex=2)), lwd.zero=1, lwd.ci=1.5, lwd.xaxis=2, lty.ci=1.5, ci.vertices =T, ci.vertices.height=0.2, clip=c(0.1,8), ineheight=unit(8, 'mm'), line.margin=unit(8, 'mm'), colgap=unit(6, 'mm'), fn.ci_norm="fpDrawDiamondCI", title="多因素Cox回归森林图", col=fpColors(box ='#021eaa', lines ='#021eaa', zero = "black")) 细节优化
代码的详细解释及其本期数据:R语言|12. 森林图-1: 多因素Cox回归模型森林图 (基于forestplot包)
#多个基因批量cox 多因素回归分析 mySurv=with(phe,Surv(time, event)) cox_results <-apply(exprSet , 1 , function(gene){ #把每个基因 分为高低表达两组 进行多因素回归分析 group=ifelse(gene>median(gene),'high','low') survival_dat <- data.frame(group=group, sex=phe$sex, age=phe$age,stringsAsFactors = F) m=coxph(mySurv ~ sex + age + group, # 代码核心句 感兴趣的因素放在最后,这里和deseq2的design正好位置相反 data = survival_dat) beta <- coef(m) se <- sqrt(diag(vcov(m))) HR <- exp(beta) #提取风险因子 HRse <- HR * se #signif(summary(m)$conf.int[,"lower .95"], 5) #lower .95 下限值HR (95% CI for HR) #signif(summary(m)$conf.int[,"upper .95"], 5) #lower .95 上限值HR (95% CI for HR) #summary(m) tmp <- round(cbind(coef = beta, se = se, z = beta/se, p = 1 - pchisq((beta/se)^2, 1), HR = HR, HRse = HRse, HRz = (HR - 1) / HRse, HRp = 1 - pchisq(((HR - 1)/HRse)^2, 1), HRCILL = exp(beta - qnorm(.975, 0, 1) * se), HRCIUL = exp(beta + qnorm(.975, 0, 1) * se), lower_95=signif(summary(m)$conf.int[,"lower .95"], 5), upper_95=signif(summary(m)$conf.int[,"upper .95"], 5) ), 3) return(tmp['grouplow',]) } ) cox_results=t(cox_results) head(cox_results) table(cox_results[,4]<0.05) cox_results[cox_results[,4]<0.05,] head(cox_results) head(cox_results[cox_results[,4]<0.05,]) summary(m) #save(phe,phe_final_3,exprSet,cox_results,file = "G:/r/duqiang_IPF/GSE70866_metainformation_4_platforms/3_ipf_combined_cox_univariate_Adjuste_for_age_sex.RData") load("G:/r/duqiang_IPF/GSE70866_metainformation_4_platforms/3_ipf_combined_cox_univariate_Adjuste_for_age_sex.RData") #单个基因cox 回归分析 (多因素) #森林图绘制https://www.jianshu.com/p/fc3b library("survival") library("survminer") res.cox <- coxph(Surv(time, event) ~ KLHL13+HECW1+USP2+sex, data = phe_final_3) res.cox summary(res.cox) #一-2 multi1:用于提取:变量+HR+95%CI+95%CI,4列 mul_cox1<-summary(res.cox) colnames(mul_cox1$conf.int) multi1<-as.data.frame(round(mul_cox1$conf.int[,c(1,3,4)],2)) multi1 #multi2 提取 HR library(tableone) multi1<-ShowRegTable(res.cox, exp=TRUE, digits=2, pDigits =3, printToggle = TRUE, quote=FALSE, ciFun=confint) ##2 提取HR.P.95%CI cox1<-summary(res.cox) #3 提取回归系数、统计量等 library(broom) multi2<-tidy(res.cox);multi2 #4 将两次提取结果合并 multi3=cox1$conf.int;multi3 multi<-cbind(multi1,multi2,multi3);multi result=tibble::rownames_to_column(multi,var = "Characteristics");result #森林图的基本图形 #BiocManager::install("forestplot") library(forestplot) fig1<- forestplot(result[,c(1,2,3)], #告诉函数,合成的表格result的第1,2,3列还是显示数字 mean=result[,9], #告诉函数,表格第9列为HR,它要变成森林图的小方块 lower=result[,ncol(results)-1], #告诉函数表格第3列为5%CI, upper=result[,ncol(results)], #表格第5列为95%CI,它俩要化作线段,穿过方块 zero=1, #告诉函数,零线或参考线为HR=1即x轴的垂直线 boxsize=0.3, #设置小黑块的大小 graph.pos=2) #森林图应插在图形第2列今天的文章
R语言:多因素Cox回归森林图 (基于forestplot包) 森林图 cox可视化分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/60681.html