作者:Jason Brownlee 2020年3月16日
多分类问题是必须对标签进行预测且可以对两个以上的标签进行预测的分类问题。
这是一种具有挑战性的预测性建模问题,因为模型学习问题时需要每个类有足够的代表性的数据。 当每个类别中的数据数量不平衡即偏向一个或几个类别而其他类别的数据很少时,问题将比较有难度。
上述的问题被称为不平衡的多类分类问题,这类问题需要仔细设计评估指标和测试工具,准确地选择机器学习模型。 大肠杆菌蛋白质定位位点数据集是用于此类问题的标准数据集。
在本教程中,您将学习到如何对不平衡的多分类数据集E.coli建立和评估模型。
学习完本教程后,您将了解一下知识:
- 如何加载和探索数据集并为数据预处理和模型选择提供思路。
- 如何使用强大的测试工具系统地评估一套机器学习模型。
- 如何训练最终模型并使用它来预测特定数据的类别标签。
在我的新书SMOTE中可以了解到单类分类(one-class classification),成本敏感型学习,阈值移动等更多内容,其中包含30步循序渐进的教程和完整的Python源代码。
让我们开始吧。
教程概述
这篇教程分为以下五个部分:
- E.coli数据集
- 探索数据集
- 测试模型并获取基准结果
- 评估模型
评估机器学习算法
评估数据过采样 - 对新数据进行类别预测
大肠杆菌(E.coli)数据集
在本项目中,我们将使用标准的不平衡机器学习数据集,称为“大肠杆菌”数据集,也称为“蛋白质定位位点”数据集。
该数据集描述了在细胞定位位点中使用氨基酸序列对大肠杆菌蛋白进行分类的问题。 即通过蛋白质在折叠之前的化学组成来预测蛋白质如何与细胞结合。
该数据集由Kenta Nakai创造,并由Paul Horton和Kenta Nakai在其1996年的论文“用于预测蛋白质细胞定位位点的概率分类系统”(A Probabilistic Classification System For Predicting The Cellular Localization Sites Of Proteins)中确定为当前形势。 在论文中,他们实现了81%的准确率。
336种大肠杆菌蛋白质被分为8类,准确性为81%…
—预测蛋白质细胞定位位点的概率分类系统,1996年。
该数据集由336个大肠杆菌蛋白质数据组成,每个数据均使用从蛋白质氨基酸序列计算出的七个输入变量进行描述。
忽略序列名称,输入变量的功能描述如下:
- mcg:McGeoch的信号序列识别方法。
- gvh:von Heijne的信号序列识别方法。
- lip:冯·海涅的Signal Peptidase II共有序列得分。
- chg:预测脂蛋白N端上存在电荷。
- aac:外膜和周质蛋白氨基酸含量的判别分析得分。
- alm1:ALOM跨膜区域预测程序的分数。
- alm2:从序列中排除假定的可裂解信号区域后,ALOM程序的得分。
总共有八个类,介绍如下:
- cp:细胞质
- im:没有信号序列的内膜
- pp:周质
- imU:内膜,不可切割的信号序列
- om:外膜
- omL:外膜脂蛋白
- imL:内膜脂蛋白
- imS:内膜,可裂解信号序列
数据集在各个类别中的分布不相等,甚至某些类别下严重失衡。
例如,“ cp”类有143个示例,而“ imL”和“ imS”类各只有两个示例。
下面,让我们深入地探索一下数据集。
作者提供的免费不平衡数据集分类问题课程
探索数据集
首先,下载并解压缩数据集,将其保存在当前工作目录中,名称为“ ecoli.csv”。
请注意,此版本的数据集删除了第一列(序列名称),因为它不包含建模的可推广信息。
- 下载数据集(ecoli.csv)
查看文件内容。
文件的前几行应如下所示:
0.49,0.29,0.48,0.50,0.56,0.24,0.35,cp
0.07,0.40,0.48,0.50,0.54,0.35,0.44,cp
0.56,0.40,0.48,0.50,0.49,0.37,0.46,cp
0.59,0.49,0.48,0.50,0.52,0.45,0.36,cp
0.23,0.32,0.48,0.50,0.55,0.25,0.35,cp
...
我们可以看到所有输入变量都显示为数字,并且类标签是字符串值,在建模我们之前需要对类标签进行编码。
可以使用Pandas中的read_csv()函数将数据集作为DataFrame加载,并在参数中指定文件的位置以及数据集没有标题行没有标题行。
...
# define the dataset location
filename = 'ecoli.csv'
# load the csv file as a data frame
dataframe = read_csv(filename, header=None)
加载数据集后,我们可以通过打印DataFrame的形状来确定行数和列数。
...
# summarize the shape of the dataset
print(dataframe.shape)
接下来,我们可以为每个输入变量截取一个拥有五个精确数字的近似值。
...
# describe the dataset
set_option('precision', 3)
print(dataframe.describe())
最后,我们还可以使用Counter对象总结每个类中的数据数量。
...
# summarize the class distribution
target = dataframe.values[:,-1]
counter = Counter(target)
for k,v in counter.items():
per = v / len(target) * 100
print('Class=%s, Count=%d, Percentage=%.3f%%' % (k, v, per))
结合在一起,下面列出了加载和汇总数据集的完整代码。
# load and summarize the dataset
from pandas import read_csv
from pandas import set_option
from collections import Counter
# define the dataset location
filename = 'ecoli.csv'
# load the csv file as a data frame
dataframe = read_csv(filename, header=None)
# summarize the shape of the dataset
print(dataframe.shape)
# describe the dataset
set_option('precision', 3)
print(dataframe.describe())
# summarize the class distribution
target = dataframe.values[:,-1]
counter = Counter(target)
for k,v in counter.items():
per = v / len(target) * 100
print('Class=%s, Count=%d, Percentage=%.3f%%' % (k, v, per))
首先运行上述代码,将加载数据集并确认行和列的数量,即336行,7个输入变量和1个目标变量。
查看每个变量的近似值,似乎变量已居中,即已调整为均值等于0.5。 似乎变量已被归一化,这意味着所有值都在大约0到1之间; 至少没有变量的值超出此范围。
然后汇总类别分布,确认每个类别的观察结果均存在严重偏斜。 我们可以看到“ cp”类占主导地位,约有42%的示例,少数类(例如“ imS”,“ imL”和“ omL”)占数据集的约1%或更少。
可能没有足够的数据可以概括这些少数群体。 一种处理方法是简单地删除带有这些类的数据。
(336, 8)
0 1 2 3 4 5 6
count 336.000 336.000 336.000 336.000 336.000 336.000 336.000
mean 0.500 0.500 0.495 0.501 0.500 0.500 0.500
std 0.195 0.148 0.088 0.027 0.122 0.216 0.209
min 0.000 0.160 0.480 0.500 0.000 0.030 0.000
25% 0.340 0.400 0.480 0.500 0.420 0.330 0.350
50% 0.500 0.470 0.480 0.500 0.495 0.455 0.430
75% 0.662 0.570 0.480 0.500 0.570 0.710 0.710
max 0.890 1.000 1.000 1.000 0.880 1.000 0.990
Class=cp, Count=143, Percentage=42.560%
Class=im, Count=77, Percentage=22.917%
Class=imS, Count=2, Percentage=0.595%
Class=imL, Count=2, Percentage=0.595%
Class=imU, Count=35, Percentage=10.417%
Class=om, Count=20, Percentage=5.952%
Class=omL, Count=5, Percentage=1.488%
Class=pp, Count=52, Percentage=15.476%
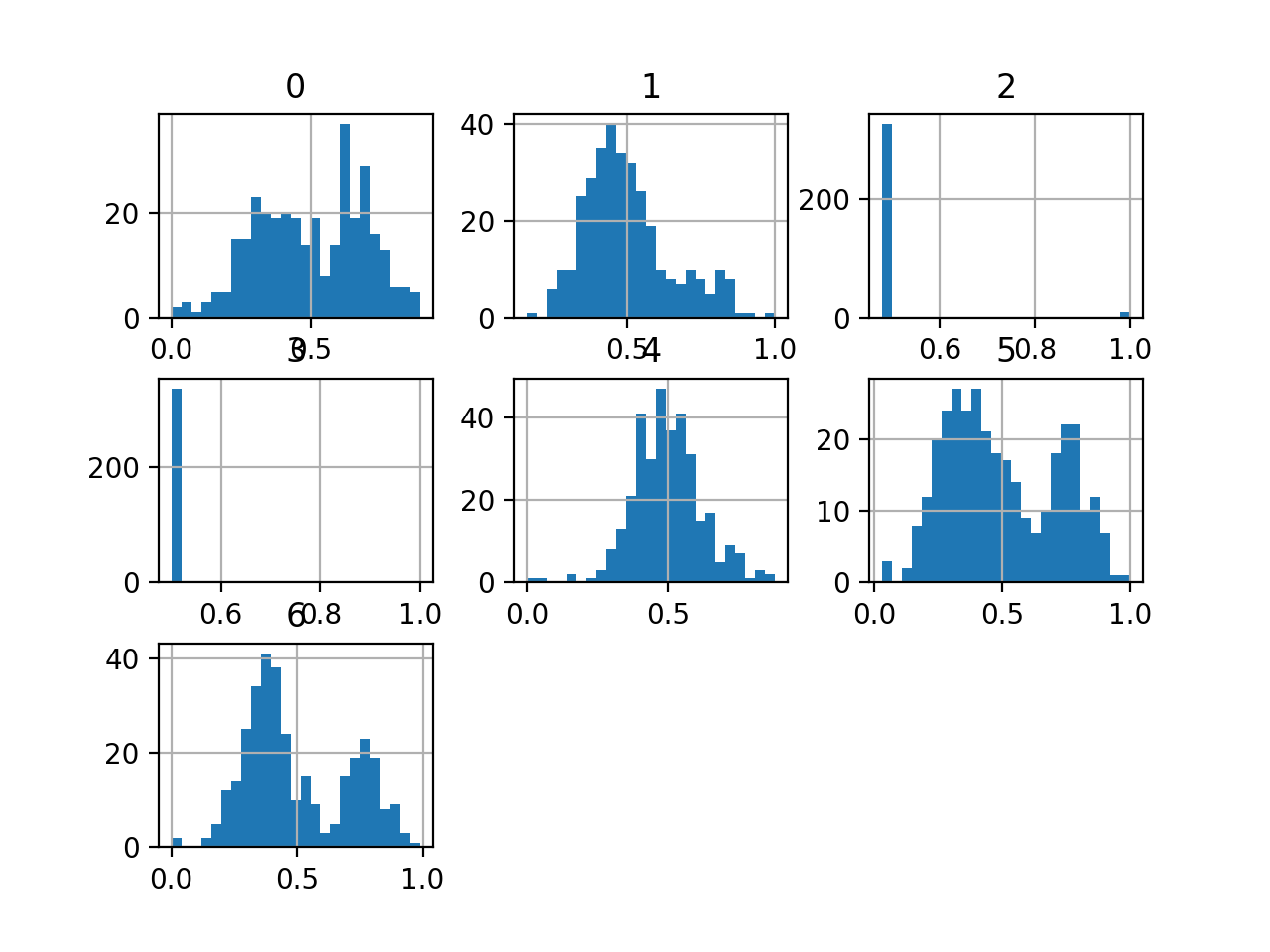
我们还可以通过为每个类别创建一个直方图来查看输入变量的分布。
下面列出了创建所有输入变量的直方图的完整代码。
# create histograms of all variables
from pandas import read_csv
from matplotlib import pyplot
# define the dataset location
filename = 'ecoli.csv'
# load the csv file as a data frame
df = read_csv(filename, header=None)
# create a histogram plot of each variable
df.hist(bins=25)
# show the plot
pyplot.show()
我们可以看到诸如0、5和6之类的类别似乎具有多峰分布。 类别2和3似乎具有二进制分布,类别1和4似乎具有高斯分布。
大肠杆菌数据集中的变量直方图
现在,我们已经检查了数据集,下面让我们来看一下如何开发用于评估候选模型的测试工具。
测试模型并获取基准结果
k折叠交叉验证(k-fold cross-validation)过程与单次训练并测试的过程相比,可以提供对模型性能的更加良好的总体估计,而不会限于局部最优。 我们将使用k = 5,这意味着每个折叠将包含大约336/5即67个示例。
分层意味着每个折叠的目标是将数据按照与训练集相同的类别比例进行混合。 重复表示评估过程将执行多次,以避免偶然结果并更好地获得所选模型之间的差异。 我们将重复三次。
这意味着将对每个模型进行拟合并评估5 * 3即15次,并得到这些运行结果的平均值和标准差。
以上过程可以使用scikit-learn中的RepeatedStratifiedKFold类来实现。
所有类别都同样重要。 因此,我们将使用分类准确性来评估模型。
首先,我们可以定义一个函数来加载数据集并将输入变量分为输入变量和输出变量,然后使用标签编码器确保对类标签进行顺序编号。
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
data = read_csv(full_path, header=None)
# retrieve numpy array
data = data.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable to have the classes 0 and 1
y = LabelEncoder().fit_transform(y)
return X, y
我们可以定义一个函数,该方法使用分层重复的5倍交叉验证来评估候选模型,然后返回每个折叠重复的在模型上计算出的分数列表。
下面的validate_model()函数实现了这一点。
# evaluate a model
def evaluate_model(X, y, model):
# define evaluation procedure
cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1)
# evaluate model
scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1)
return scores
然后,我们可以调用load_dataset()函数来加载数据集。
...
# define the location of the dataset
full_path = 'ecoli.csv'
# load the dataset
X, y = load_dataset(full_path)
# summarize the loaded dataset
print(X.shape, y.shape, Counter(y))
在这种情况下,我们将所有情况下的预测。
这可以使用DummyClassifier类自动设置,并将strategy设置为most_frequent,以预测训练数据集中最常见的类(例如“ cp”类)。由于这是训练集中最常见的类别分布,因此我们希望该模型的分类精度约为42%。
...
# define the reference model
model = DummyClassifier(strategy='most_frequent')
然后,我们可以通过调用validate_model()函数来评估模型,并获得结果的均值和标准差。
...
# evaluate the model
scores = evaluate_model(X, y, model)
# summarize performance
print('Mean Accuracy: %.3f (%.3f)' % (mean(scores), std(scores)))
综上所述,下面列出了使用分类精度评估大肠杆菌数据集模型的完整代码。
# baseline model and test harness for the ecoli dataset
from collections import Counter
from numpy import mean
from numpy import std
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.dummy import DummyClassifier
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
data = read_csv(full_path, header=None)
# retrieve numpy array
data = data.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable to have the classes 0 and 1
y = LabelEncoder().fit_transform(y)
return X, y
# evaluate a model
def evaluate_model(X, y, model):
# define evaluation procedure
cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1)
# evaluate model
scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1)
return scores
# define the location of the dataset
full_path = 'ecoli.csv'
# load the dataset
X, y = load_dataset(full_path)
# summarize the loaded dataset
print(X.shape, y.shape, Counter(y))
# define the reference model
model = DummyClassifier(strategy='most_frequent')
# evaluate the model
scores = evaluate_model(X, y, model)
# summarize performance
print('Mean Accuracy: %.3f (%.3f)' % (mean(scores), std(scores)))
&esmp;&esmp;首先运行代码将加载数据集,并获得数据数量——336,之后按照我们的预期获得类别标签的分布。
然后,使用重复的分层k倍交叉验证对DummyClassifier进行评估,获得分类准确度的平均值和标准差约为42.6%。
(336, 7) (336,) Counter({
0: 143, 1: 77, 7: 52, 4: 35, 5: 20, 6: 5, 3: 2, 2: 2})
Mean Accuracy: 0.426 (0.006)
在评估模型期间会报告警告; 例如:
Warning: The least populated class in y has only 2 members, which is too few. The minimum number of members in any class cannot be less than n_splits=5.
&smep;这是因为某些类没有足够数量的5倍交叉验证数据,例如 类“ imS”和“ imL”。
在这种情况下,我们将从数据集中删除这些示例。 这可以通过更新load_dataset()来删除具有这些类的那些行来实现。
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
df = read_csv(full_path, header=None)
# remove rows for the minority classes
df = df[df[7] != 'imS']
df = df[df[7] != 'imL']
# retrieve numpy array
data = df.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable to have the classes 0 and 1
y = LabelEncoder().fit_transform(y)
return X, y
然后,我们可以重新运行该代码以建立分类精度基准。
下面列出了完整的代码。
# baseline model and test harness for the ecoli dataset
from collections import Counter
from numpy import mean
from numpy import std
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.dummy import DummyClassifier
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
df = read_csv(full_path, header=None)
# remove rows for the minority classes
df = df[df[7] != 'imS']
df = df[df[7] != 'imL']
# retrieve numpy array
data = df.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable to have the classes 0 and 1
y = LabelEncoder().fit_transform(y)
return X, y
# evaluate a model
def evaluate_model(X, y, model):
# define evaluation procedure
cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1)
# evaluate model
scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1)
return scores
# define the location of the dataset
full_path = 'ecoli.csv'
# load the dataset
X, y = load_dataset(full_path)
# summarize the loaded dataset
print(X.shape, y.shape, Counter(y))
# define the reference model
model = DummyClassifier(strategy='most_frequent')
# evaluate the model
scores = evaluate_model(X, y, model)
# summarize performance
print('Mean Accuracy: %.3f (%.3f)' % (mean(scores), std(scores)))
运行代码可以确认数据数量减少了4个,从336个减少到332个。
我们还可以看到,类的数量从八种减少到了六种(从0类到5类)。
基准确定为43.1%。 该分数为该数据集提供了基线,通过该基线可以比较所有其他分类算法。 达到约43.1%以上的分数表示模型对该数据集具有分类能力,而分数等于或低于此值则表明该模型对该数据集不具有良好的分类能力。
(332, 7) (332,) Counter({
0: 143, 1: 77, 5: 52, 2: 35, 3: 20, 4: 5})
Mean Accuracy: 0.431 (0.005)
现在我们有了一个测试工具和一个性能基准,我们可以开始评估该数据集上的一些模型。
评估模型
在本节中,我们将使用上一节中开发的测试工具对数据集进行评估。
结果的性能很好,但尚未高度优化(例如,未调整超参数)。
你能做得更好吗? 如果您可以使用相同的测试工具来获得更好的分类精度,那么我也很想知道。 请在下面的评论中发表自己的看法。
评估机器学习算法
首先,评估数据集上各种机器学习模型。
最好对数据集进行抽查,以检查出一组不同的非线性算法,以快速分辨出那些算法比较有效,值得进一步注意,哪些算法无效。
我们将在数据集上评估以下机器学习模型:
- 线性判别分析(LDA)
- 支持向量机(SVM)
- 决策树(BAG)
- 随机森林(RF)
- Extra Trees(ET)
我们将主要使用默认的模型超参数,但集成算法中的树的数量除外,我们将其设置为合理的默认值1000。
我们将依次定义每个模型,并将它们添加到列表中,以便我们可以依次评估它们。 下面的get_models()函数定义了用于评估的模型列表,以及用于稍后绘制结果的模型,我们将它简称列表。
# define models to test
def get_models():
models, names = list(), list()
# LDA
models.append(LinearDiscriminantAnalysis())
names.append('LDA')
# SVM
models.append(LinearSVC())
names.append('SVM')
# Bagging
models.append(BaggingClassifier(n_estimators=1000))
names.append('BAG')
# RF
models.append(RandomForestClassifier(n_estimators=1000))
names.append('RF')
# ET
models.append(ExtraTreesClassifier(n_estimators=1000))
names.append('ET')
return models, names
然后,我们可以依次列举列表汇总的模型并评估每个模型,并存储得分以供以后评估。
...
# define models
models, names = get_models()
results = list()
# evaluate each model
for i in range(len(models)):
# evaluate the model and store results
scores = evaluate_model(X, y, models[i])
results.append(scores)
# summarize performance
print('>%s %.3f (%.3f)' % (names[i], mean(scores), std(scores)))
在运行结束时,我们可以将每个得分样本绘制为具有相同比例的箱型图,以便我们可以直接比较分布。
...
# plot the results
pyplot.boxplot(results, labels=names, showmeans=True)
pyplot.show()
综上所述,下面列出了评估数据集上一组机器学习算法的完整示例。
# spot check machine learning algorithms on the ecoli dataset
from numpy import mean
from numpy import std
from pandas import read_csv
from matplotlib import pyplot
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.svm import LinearSVC
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import ExtraTreesClassifier
from sklearn.ensemble import BaggingClassifier
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
df = read_csv(full_path, header=None)
# remove rows for the minority classes
df = df[df[7] != 'imS']
df = df[df[7] != 'imL']
# retrieve numpy array
data = df.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable
y = LabelEncoder().fit_transform(y)
return X, y
# evaluate a model
def evaluate_model(X, y, model):
# define evaluation procedure
cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1)
# evaluate model
scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1)
return scores
# define models to test
def get_models():
models, names = list(), list()
# LDA
models.append(LinearDiscriminantAnalysis())
names.append('LDA')
# SVM
models.append(LinearSVC())
names.append('SVM')
# Bagging
models.append(BaggingClassifier(n_estimators=1000))
names.append('BAG')
# RF
models.append(RandomForestClassifier(n_estimators=1000))
names.append('RF')
# ET
models.append(ExtraTreesClassifier(n_estimators=1000))
names.append('ET')
return models, names
# define the location of the dataset
full_path = 'ecoli.csv'
# load the dataset
X, y = load_dataset(full_path)
# define models
models, names = get_models()
results = list()
# evaluate each model
for i in range(len(models)):
# evaluate the model and store results
scores = evaluate_model(X, y, models[i])
results.append(scores)
# summarize performance
print('>%s %.3f (%.3f)' % (names[i], mean(scores), std(scores)))
# plot the results
pyplot.boxplot(results, labels=names, showmeans=True)
pyplot.show()
运行代码依次评估每种算法,并报告分类准确性的均值和标准差分类。
鉴于算法的随机性,您的具体结果会有所不同; 可以多运行几次。
在这种情况下,我们可以看到所有经过测试的算法都具有不错的结果,可以达到高于默认值43.1%的精度。
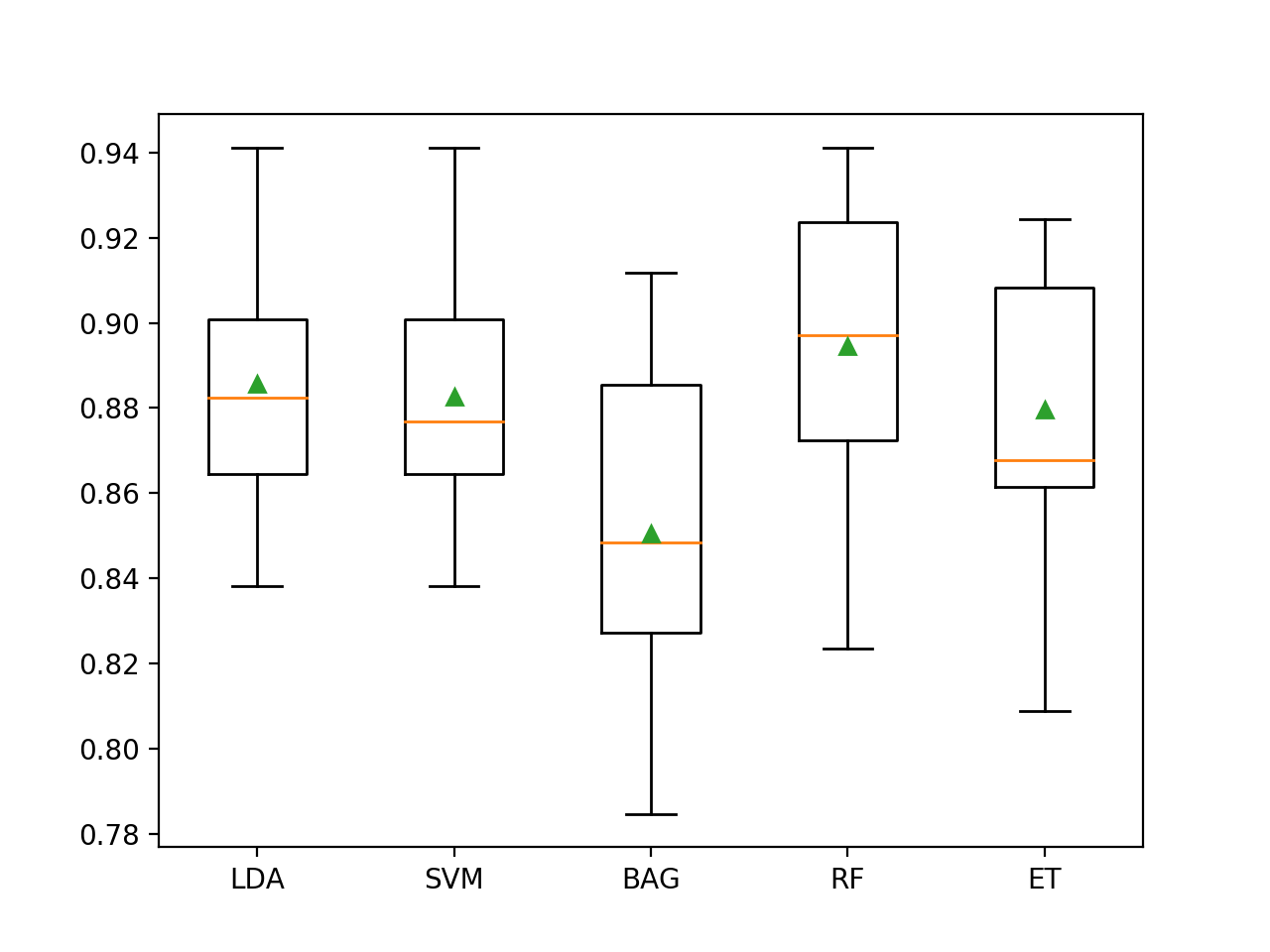
结果表明,大多数算法在该数据集上效果都很好,决策树的组合表现最佳,其中Extra Trees达到88%的准确度,Random Forest达到89.5%的准确度。
>LDA 0.886 (0.027)
>SVM 0.883 (0.027)
>BAG 0.851 (0.037)
>RF 0.895 (0.032)
>ET 0.880 (0.030)
下面的代码将创建箱型图,显示每个算法的结果。 该框显示数据的中间50%,每个框中间的橙色线显示样本的中位数,每个框中的绿色三角形显示样本的平均值。
&emps;我们可以看到,决策树集合的分数分布与其他测试算法是分开的。 在大多数情况下,平均值和中位数在图中接近,表明分数的分布大概对称,这可以表明模型是稳定的。
基于不平衡大肠杆菌数据集的机器学习模型的箱型图
评估数据过采样
虽然有了这么多的类,但许多类中的样本却很少,数据集可能会受益于过采样。
我们可以测试适用于除多数类(cp)以外的所有样本的SMOTE算法,从而提高性能。
通常,SMOTE似乎无法帮助决策树的集成,因此我们将算法更改为以下内容:
- 多项逻辑回归(LR)
- 线性判别分析(LDA)
- 支持向量机(SVM)
- k近邻(KNN)
- Gaussian Process(GP)
下面列出了定义这些模型的get_models()函数的更新版本。
# define models to test
def get_models():
models, names = list(), list()
# LR
models.append(LogisticRegression(solver='lbfgs', multi_class='multinomial'))
names.append('LR')
# LDA
models.append(LinearDiscriminantAnalysis())
names.append('LDA')
# SVM
models.append(LinearSVC())
names.append('SVM')
# KNN
models.append(KNeighborsClassifier(n_neighbors=3))
names.append('KNN')
# GP
models.append(GaussianProcessClassifier())
names.append('GP')
return models, names
我们可以使用来自不平衡学习库的SMOTE实现。首先将SMOTE应用于训练集,然后将给定模型拟合为交叉验证过程的一部分。
SMOTE将使用训练数据集中的k个最近数据来合成新数据,默认情况下,k设置为5。
对于我们的数据集中的某些类来说,这太大了。 因此,我们尝试将k值设为2。
...
# create pipeline
steps = [('o', SMOTE(k_neighbors=2)), ('m', models[i])]
pipeline = Pipeline(steps=steps)
# evaluate the model and store results
scores = evaluate_model(X, y, pipeline)
结合在一起,下面列出了在数据集上使用SMOTE过采样的完整代码。
# spot check smote with machine learning algorithms on the ecoli dataset
from numpy import mean
from numpy import std
from pandas import read_csv
from matplotlib import pyplot
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.svm import LinearSVC
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.neighbors import KNeighborsClassifier
from sklearn.gaussian_process import GaussianProcessClassifier
from sklearn.linear_model import LogisticRegression
from imblearn.pipeline import Pipeline
from imblearn.over_sampling import SMOTE
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
df = read_csv(full_path, header=None)
# remove rows for the minority classes
df = df[df[7] != 'imS']
df = df[df[7] != 'imL']
# retrieve numpy array
data = df.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable
y = LabelEncoder().fit_transform(y)
return X, y
# evaluate a model
def evaluate_model(X, y, model):
# define evaluation procedure
cv = RepeatedStratifiedKFold(n_splits=5, n_repeats=3, random_state=1)
# evaluate model
scores = cross_val_score(model, X, y, scoring='accuracy', cv=cv, n_jobs=-1)
return scores
# define models to test
def get_models():
models, names = list(), list()
# LR
models.append(LogisticRegression(solver='lbfgs', multi_class='multinomial'))
names.append('LR')
# LDA
models.append(LinearDiscriminantAnalysis())
names.append('LDA')
# SVM
models.append(LinearSVC())
names.append('SVM')
# KNN
models.append(KNeighborsClassifier(n_neighbors=3))
names.append('KNN')
# GP
models.append(GaussianProcessClassifier())
names.append('GP')
return models, names
# define the location of the dataset
full_path = 'ecoli.csv'
# load the dataset
X, y = load_dataset(full_path)
# define models
models, names = get_models()
results = list()
# evaluate each model
for i in range(len(models)):
# create pipeline
steps = [('o', SMOTE(k_neighbors=2)), ('m', models[i])]
pipeline = Pipeline(steps=steps)
# evaluate the model and store results
scores = evaluate_model(X, y, pipeline)
results.append(scores)
# summarize performance
print('>%s %.3f (%.3f)' % (names[i], mean(scores), std(scores)))
# plot the results
pyplot.boxplot(results, labels=names, showmeans=True)
pyplot.show()
运行代码依次评估每种算法,并获得分类准确率的均值和标准差。
鉴于算法的随机性,您的具体结果会有所不同。可以多运行几次。
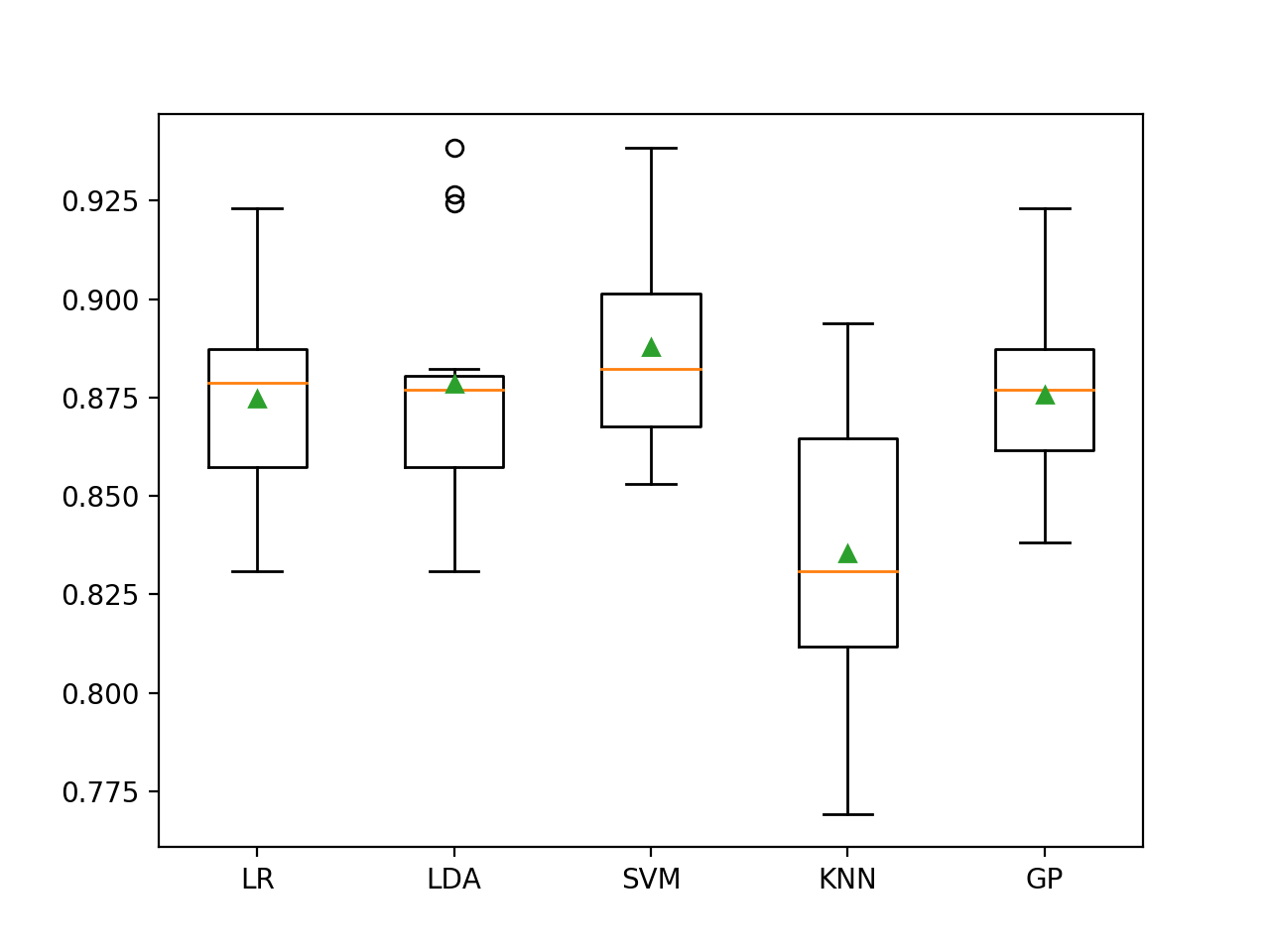
在这种情况下,我们可以看到带有SMOTE的LDA从88.6%小幅下降到大约87.9%,而带有SMOTE的SVM则从大约88.3%小幅增长到大约88.8%。
在这种情况下,使用SMOTE时,SVM似乎也是性能最好的方法,尽管与上一节中的随机森林相比,它没有改进。
>LR 0.875 (0.024)
>LDA 0.879 (0.029)
>SVM 0.888 (0.025)
>KNN 0.835 (0.040)
>GP 0.876 (0.023)
为每种算法创建分类准确度得分的箱型图。
我们可以看到,LDA有许多性能异常值,最高有90%,这很有趣。 这可能表明,如果专注于较多的类别,LDA可能会表现更好。
不平衡大肠杆菌数据集上具有机器学习模型的SMOTE的箱型图
现在,我们已经了解了如何在此数据集上评估模型,让我们看看如何使用最终模型进行预测。
在新数据上进行预测
在本节中,我们可以拟合最终模型,并使用它对单行数据进行预测。
我们将使用随机森林模型作为最终模型,该模型可实现约89.5%的分类精度。
首先,我们对模型进行定义:
...
# define model to evaluate
model = RandomForestClassifier(n_estimators=1000)
我们就可以将模型适用于整个训练集。
...
# fit the model
model.fit(X, y)
拟合后,我们就可以通过调用prepare()函数将其用于新数据的预测。 这将为每个数据返回类别标签的编码。
然后,我们可以使用标签编码器进行逆变换以获得字符串类标签。例如:
...
# define a row of data
row = [...]
# predict the class label
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
为了证明这一点,我们可以使用已经拟合的模型对一些我们知道结果的数据进行标签预测。
下面列出了完整的示例。
# fit a model and make predictions for the on the ecoli dataset
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.ensemble import RandomForestClassifier
# load the dataset
def load_dataset(full_path):
# load the dataset as a numpy array
df = read_csv(full_path, header=None)
# remove rows for the minority classes
df = df[df[7] != 'imS']
df = df[df[7] != 'imL']
# retrieve numpy array
data = df.values
# split into input and output elements
X, y = data[:, :-1], data[:, -1]
# label encode the target variable
le = LabelEncoder()
y = le.fit_transform(y)
return X, y, le
# define the location of the dataset
full_path = 'ecoli.csv'
# load the dataset
X, y, le = load_dataset(full_path)
# define model to evaluate
model = RandomForestClassifier(n_estimators=1000)
# fit the model
model.fit(X, y)
# known class "cp"
row = [0.49,0.29,0.48,0.50,0.56,0.24,0.35]
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
print('>Predicted=%s (expected cp)' % (label))
# known class "im"
row = [0.06,0.61,0.48,0.50,0.49,0.92,0.37]
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
print('>Predicted=%s (expected im)' % (label))
# known class "imU"
row = [0.72,0.42,0.48,0.50,0.65,0.77,0.79]
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
print('>Predicted=%s (expected imU)' % (label))
# known class "om"
row = [0.78,0.68,0.48,0.50,0.83,0.40,0.29]
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
print('>Predicted=%s (expected om)' % (label))
# known class "omL"
row = [0.77,0.57,1.00,0.50,0.37,0.54,0.0]
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
print('>Predicted=%s (expected omL)' % (label))
# known class "pp"
row = [0.74,0.49,0.48,0.50,0.42,0.54,0.36]
yhat = model.predict([row])
label = le.inverse_transform(yhat)[0]
print('>Predicted=%s (expected pp)' % (label))
首先运行代码使得模型适合整个训练集。
然后,使用模型来预测从六个类别的每个类别中抽取的一个样本的标签。
我们可以看到,模型为每个选择的样本预测了正确的类标签。 但是,平均而言,我们预计每10个预测中就有1个是错误的,并且这些错误可能不会在各个类中平均分配。
>Predicted=cp (expected cp)
>Predicted=im (expected im)
>Predicted=imU (expected imU)
>Predicted=om (expected om)
>Predicted=omL (expected omL)
>Predicted=pp (expected pp)
总结
在本教程中,您了解了如何为不平衡的多类大肠杆菌数据集开发和评估模型。
具体来说,您了解到:
- 如何加载和探索数据集并为数据准备和模型选择提供思路。
- 如何使用强大的测试工具系统地评估一套机器学习模型。
- 如何拟合最终模型并使用它来预测特定示例的类标签。
您有任何问题吗?
请在下面的评论中提出您的问题,我会尽力回答。
原文链接:https://machinelearningmastery.com/imbalanced-multiclass-classification-with-the-e-coli-dataset/
今天的文章(翻译)机器学习:E.coli数据集的不平衡多类分类分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/86543.html