R语言下多组学因子分析MOFA ——单细胞甲基化分析
前言
提问:什么是MOFA?
MOFA分析可以在常规组学分析基础上,进一步将数据进行整合,从因子分析的角度解析影响不同组学数据背后异质性因素,随后可对解析出的因子进行注释及其他扩展分析。MOFA相比于传统联合分析有比较明显的优势:
MOFA优势
- 样本量:既适用于大样本量数据,也适用于小样本量数据(每个组学具有一一对应关系的样本至少15个以上);
- 缺失值估计:对于有缺失值的样本,可以进行缺失值的估计;
- 适用的数据类型广泛:可对离散型数据、连续型数据和二进制数据进行分析;
- 应用范围广:适用于两个及两个以上组学,因子分析结果还可以继续进行非线性降维聚类(t-SNE,UMAP)和预测临床反应(Cox模型)等分析;
- 分析角度独特:从因子贡献度角度解析数据异质性,为多组学研究提供了新思路。
提示:以下是本篇文章正文内容,下面案例可供参考
一、MOFA与单细胞、甲基化数据?
为了在处理大量缺失值的同时进行降维,并分析异质性。
二、分析步骤
数据来源:“Multi-omics profiling of mouse gastrulation at single cell resolution”
数据介绍:来自同一时期三个胚层的胚胎干细胞甲基化数据
1.导入数据
代码如下(示例):
library(data.table)
library(purrr)
library(ggplot2)
#确定数据的读取路径
io <- list()
io$basedir <- "scnmt_gastrulation"
io$data.dir <- "scnmt_gastrulation/met/feature_level"
io$sample.metadata <- "scnmt_gastrulation/sample_metadata.txt"
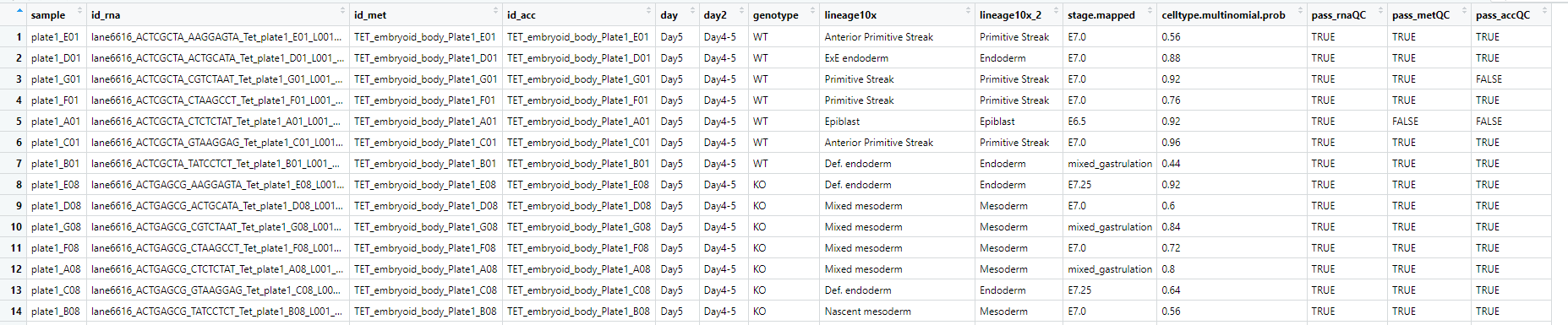
##我们可以先导入metadata看一下
sample_metadata <- fread(io$sample.metadata)
大致是这个样子,然后根据我们的要求去进行细胞筛选
前期一些不重要的环境,条件筛选设置。可根据各自文章要求来自定义设置。
opts <- list()
# 选择annotations
opts$annos <- c("ESC_DHS"="DHS")
# 选择细胞的时期
opts$stage_lineage <- c("E7.5_Endoderm","E7.5_Mesoderm","E7.5_Ectoderm")
# 选择符合标准的细胞
opts$cells <- fread(io$sample.metadata) %>%
.[,stage_lineage:=paste(stage,lineage10x_2,sep="_")] %>%
.[pass_metQC==T & stage_lineage%in%opts$stage_lineage,id_met]
# 选择符合标准的细胞的metadata
sample_metadata <- fread(io$sample.metadata) %>%
.[id_met%in%opts$cells] %>%
.[,c("id_met","stage","lineage10x_2")] %>%
.[,stage_lineage:=paste(stage,lineage10x_2,sep="_")]
# 数据筛选的一些标准
opts$min.CpGs <- 1 # minimum number of CpG sites per feature and cell
opts$min.coverage <- 0.10 # minimum coverage (fraction of cells with at least min.CpG measurements)
opts$nfeatures <- 5000 # number of features per view (filter based on variance)
# 选择颜色
opts$colors <- c( Ectoderm="steelblue",
Mesoderm="#CD3278",Endoderm="#43CD80")
导入数据并根据筛选标准过滤
##导入数据
met_dt <- lapply(names(opts$annos), function(n)
fread(sprintf("%s/%s.tsv.gz",io$data.dir,n), select=c(1,2,3,5,6)) %>%
setnames(c("id_met","id","anno","N","rate")) %>% .[N>=opts$min.CpGs] %>% .[,N:=NULL]
) %>% rbindlist %>% merge(sample_metadata, by="id_met")
# Calculate M value from Beta value
met_dt[,m:=log2(((rate/100)+0.01)/(1-(rate/100)+0.01))]
# 按coverage划分的过滤标准
nsamples <- length(unique(met_dt$id_met))
met_dt <- met_dt[,cov:=.N/nsamples,by=c("id","anno")] %>% .[cov>=opts$min.coverage] %>% .[,c("cov"):=NULL]
# 按方差划分的过滤标准
keep_hv_sites <- met_dt %>% split(.$lineage10x_2) %>% map(~ .[,.(var = var(rate)), by="id"] %>% .[var>0] %>% setorder(-var) %>% head(n = opts$nfeatures) %>% .$id)
met_dt <- met_dt %>% split(.$lineage10x_2) %>% map2(.,names(.), function(x,y) x[id %in% keep_hv_sites[[y]]]) %>% rbindlist
2.在 R 中训练模型
library(MOFA2)
#矩阵列表,其中每个条目对应一个视图。样本存储在列中,特征存储在行中。
dmatrix_list <- met_dt %>% split(.$lineage10x_2) %>%
map(~
dcast(.[,c("id_met","id","m")], formula=id_met~id, value.var="m") %>% as.data.frame %>% tibble::column_to_rownames("id_met") %>% as.matrix %>% t)
lapply(dmatrix_list,dim)
# 创建 MOFA 对象:
MOFAobject <- create_mofa(df)
plot_data_overview(MOFAobject)
# 设置参数
ModelOptions <- get_default_model_options(MOFAobject)
ModelOptions$num_factors <- 2
head(ModelOptions)
TrainOptions <- get_default_training_options(MOFAobject)
TrainOptions$seed <- 42
# Prepare
MOFAobject <- prepare_mofa(MOFAobject,
model_options = ModelOptions,
training_options = TrainOptions
)
# Train the model
model <- run_mofa(MOFAobject, io$outfile)
3. plot model
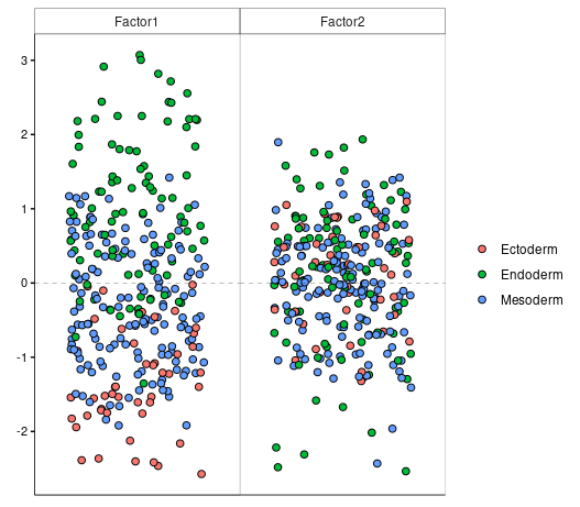
p <- plot_factor(
model,
factors=c("Factor1", "Factor2"),
color_by=sample_metadata_filt$lineage10x_2
)
p <- p +
scale_colour_manual(values=opts$colors) +
labs(x=sprintf("Factor 1 (%.2f%%)",r2["LF1",]*100), y=sprintf("Factor 2 (%.2f%%)",r2["LF2",]*100))
print(p)
图中显示了前两个潜在因素的散点图,这些因素是用来自指定阶段的细胞训练的模型。
今天的文章r语言及bioconductor在基因组分析中的应用_已知初等因子组求不变因子分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/86934.html