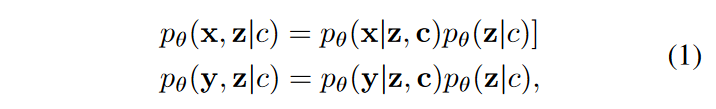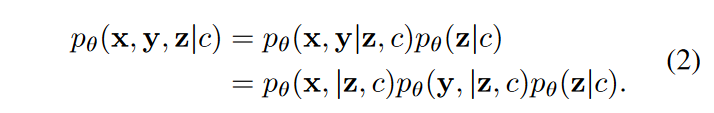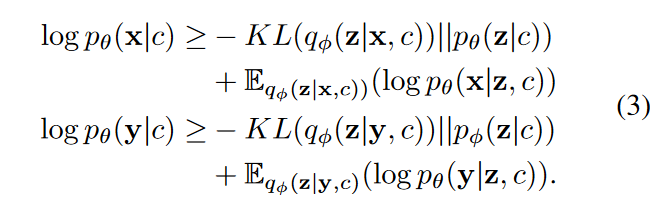ReadingNotes|COEM: Cross-Modal Embedding for MetaCell Identification – Echo的文章 – 知乎 https://zhuanlan.zhihu.com/p/545899495
Title:COEM: Cross-Modal Embedding for MetaCell Identification
links:https://arxiv.org/pdf/2207.07734
The 2022 ICML Workshop on Computational Biology.
Abstract
元细胞是不相交和均匀的单细胞组,代表离散和高度颗粒状的细胞状态。现有的元细胞算法往往只使用一种模态来推断元细胞,即使单细胞多组学数据描述了同一细胞的多种分子模态。
第一次接触metacells,对此概念不是很理解,原文如下:
Metacells are disjoint and homogeneous groups of single-cell profiles, representing discrete and highly granular cell states.
本文提出了一种基于跨模态嵌入的元细胞识别算法(cross-modal Embedding for MetaCell Identification, COEM),该算法在嵌入空间中同时利用了scATAC-seq和scRNA-seq的信息来进行聚类,在高分辨率和高测序覆盖率之间取得了平衡。COEM方法能够在连续和离散细胞类型的数据集上高效地识别出精确且分离良好的元细胞,其性能优于目前最先进的SEACells方法。此外,COEM显著改善了峰-基因(peak-to-gene)关联分析,并促进了复杂的基因调控推断任务。
Discussion
实验表明,整合来自scRNA-seq和scATAC-seq的信息有利于从单细胞数据中准确和稳健的识别元细胞。特别是联合嵌入的低维表示可以识别 细胞类型纯度高和分离良好的元细胞 的细胞状态。其他优势:
-
faster running time
-
避免了大量错误的负的峰到基因链接(peak-to-gene links)
Hence,联合分析的序列数据能够描绘细胞阶段的全面景观 (conprehensive lanscape)。此外,(Bilous et al.,2021) 已经证明从scRNA-seq推断的元细胞与RNA速度模型(velocity model)兼容。因此,COEM也可以用于估计表观基因组和转录组动力学的多组学速度模型。
Future work:探索更多用于多模态序列数据集成(multi-modal data integration)和元细胞识别(metacell identification)的机器学习技术。因为scRNA-seq和scATAC-seq具有自然的因果关系,utilizing this relation to learn more robust, causally sufficient, and efficient representations is interesting.
Besides, the results are promising for use of an optimal transport approach to model cellular stage changes across different modalities on the metacell level (Schiebinger et al., 2019).
Schiebinger, G., Shu, J., Tabaka, M., Cleary, B., Subramanian, V., Solomon, A., Gould, J., Liu, S., Lin, S., Berube, P., et al. Optimal-transport analysis of single-cell gene expression identifies developmental trajectories in reprogramming. Cell, 176(4):928–943, 2019. Bilous, M., Tran, L., Cianciaruso, C., Gabriel, A., Michel, H., Carmona, S., Pittet, M., and Gfeller, D. Metacells untangle large and complex single-cell transcriptome networks (preprint). 2021.
注:可以理解为(scRNA-seq和scATAC-seq)data integration后的一个下游任务?
Introduction
-
Background:
单细胞技术的最新进展使在单细胞分辨率下同时测量染色质可及性和基因表达水平成为可能。各种测序技术,如Sci-CAR、SNARE-seq、SHARE-seq和10X Genomics Multiome,已被开发并应用于细胞异质性、发育动力学和顺式调控(cis-regulatory)元件(CRE)的综合研究。
然而,在scRNA-seq和scATAC-seq数据的单细胞测序测定中观察到极端的数据稀疏性(sparsity)。这阻碍了单细胞水平的顺式调控推断。
生物样本的高通量单细胞分析通常会导致高度相似的和统计等效的细胞被重复抽样。元细胞的概念(Baran等人,2019年)已被提出以保持统计置信度。元细胞是一组scRNA-seq细胞图谱,代表不同的、高度粒状的细胞状态。元细胞聚集体具有足够的测序覆盖率,有助于减少下游分析中的稀疏性问题。
Baran, Y., Bercovich, A., Sebe-Pedros, A., Lubling, Y., Giladi, A., Chomsky, E., Meir, Z., Hoichman, M., Lifshitz, A., and Tanay, A. Metacell: analysis of single-cell rnaseq data using k-nn graph partitions. Genome biology, 20 (1):1–19, 2019.
cis-regulatory-顺式调控:基因启动子发生突变,使调控蛋白不能识别启动子结构,基因不能表达,这种只影响基因本身表达、不影响其它等位基因调控的突变,称顺式调控。
-
Related works:
MetaCells Algorithm 仅基于scRNA-seq数据上的k最近邻(KNN)相似性图的划分推断元细胞,但它在scATAC-seq数据上失败。
SEACell方法(Single-cell aggregation of cell-states)旨在以RNA或ATAC模态识别元细胞。它显著改善了峰到基因的关联,即染色质可及性峰和基因表达之间的关联。然而,SEACell仅基于一种模式识别元细胞:提出严格的生物学假设,即可及染色质与活性转录一致相关,尽管不同的数据模式可能在基因的染色质重塑与其转录之间表现出时间差。.这种“时滞”的发生表现在: 1.染色质在转录开始前打开;2.染色质关闭后mRNA降解。
Persad, S., Choo, Z.-N., Dien, C., Masilionis, I., Chalign ́ e, R., Nawy, T., Brown, C. C., Pe’er, I., Setty, M., and Pe’er, D. Seacells: Inference of transcriptional and epigenomic cellular states from single-cell genomics data. bioRxiv, 2022.
-
Main work:
这篇文章的目的是研究scATAC-seq和scRNA-seq数据的综合分析,以提高发现和表征细胞状态的能力,同时避免有偏差的关联。
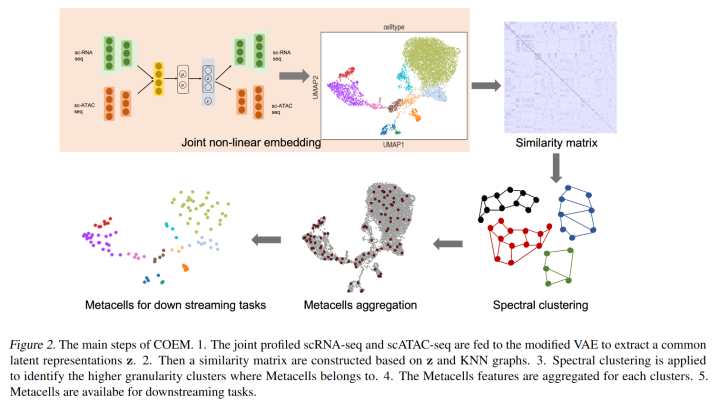
文章提出的新算法COEM旨在综合分析多组测序单细胞数据(例如,在同一细胞中测量的基因表达和染色质可及性)。COEM首先通过多视图、多模态变分自编码器(VAE)模型学习scRNA-seq和scATAC-seq数据的联合低维潜在表示来解决数据整合问题(Baltru saitis等人,2018年);进一步利用基于图连通性的谱聚类方法来识别RNA和染色质水平上代表高度粒度、不同细胞状态的元细胞。发现COEM在具有离散细胞类型和连续发育轨迹的数据集中对元细胞的识别中比SEACell更准确。此外,COEM识别的元细胞大大改善了峰到基因的关联分析,并支持CRE预测。
注:peak-to-gene 暂时还是不清楚,关联分析我认为可以理解为,之前数据整合中会将峰矩阵转化为基因表达矩阵后再与scRNA-seq进行整合。
Method
1. 多模态联合嵌入Multi-Modal Joint Embedding
COEM从同一细胞中测量的scRNA-seq和scATAC-seq数据中联合学习低维表征,采用改进的多视图(multi-view)VAE来提取两种数据模态的公共潜在空间。VAEs的生成模型如下:
Given a cell, the scRNA gene expression x x\in P_{x}, scATAC chromatin accessibility data y\in P_{y} , cell type c , and the common latent representation z , we have the following probability densities,
其中 \theta 表示生成模型的参数。
x 和 y 的似然函数取决于 共同潜在空间 z 和细胞类型 c ,当给定 z 和 c , x 和 y 的分布是独立的。所以可以得到联合生成模型:
编辑切换为居中
添加图片注释,不超过 140 字(可选)
对于具有条件变分近似 q_{\phi} 的两个模态,我们有以下证据下界(ELBO, evidence lower bound),
编辑切换为居中
添加图片注释,不超过 140 字(可选)
通常使用推断网络inference network(编码器)来学习变分近似q_{\phi},和生成网络generative network(解码器)来学习 p_{\theta}。特别地,我们假设 p_{\theta}(x|z,c) 是零膨胀泊松分布,p_{\theta}(y|z,c)是负二项式(NB)分布,p_{\theta}(z|c)是高斯混合模型。
给定联合嵌入生成式模型,我们采用scMVP(Li et al.,2022)架构对生成网络和推理网络进行建模。具体来说,对于scRNA-seq和scATAC-seq输入,存在一个基于双通道注意力的编码器网络,然后将它们连接以导出后验分布p_{\phi}(z|x,y,c)。接下来,通过基于注意力的双通道解码器网络重建估算的scRNA和scATAC profiles,以计算似然p_{\theta}(x,y|z,c)。
2. 元细胞认定Metacells Identification
COEM是一种基于图,采用谱聚类来计算元细胞的方法。更具体地说,COEM由以下四个步骤组成:
-
应用跨模态联合嵌入VAE来学习低维公共表征z,如何编码scRNA-seq和scATAC-seq的公共信息。
-
基于欧式距离在低维嵌入空间z上构建k-近邻(KNN)图。
-
通过连通性从KNN图构建相似性矩阵。相似性矩阵基于径向基函数(radial basis function-RBF)核来编码z中的非线性关系。
-
利用谱聚类来识别簇,并随后聚合元细胞,如(Persad,2022)中所述。
Persad, S., Choo, Z.-N., Dien, C., Masilionis, I., Chalign ́ e, R., Nawy, T., Brown, C. C., Pe’er, I., Setty, M., and Pe’er, D. Seacells: Inference of transcriptional and epigenomic cellular states from single-cell genomics data. bioRxiv, 2022.
COEM与SEACells都基于graph,区别是元细胞认定COEM采用谱聚类,而SEACells采用原型分析(archetypal analysis)。
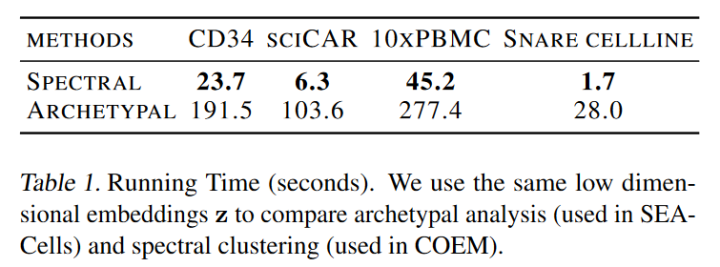
COEM的谱聚类纯粹利用图的连通性信息,运行速度更快。且文章验证了谱聚类可以提高元细胞识别和运行时间。
采用不同的低维嵌入空间,比较原型分析和谱聚类的运行时间
Experiment & Results
正如在SEACells论文中一样,我们使用致密性、分离度和细胞类型纯度作为元细胞基准。致密性衡量元细胞内细胞的同质程度。分离评估元细胞之间的差异。相反,细胞类型纯度衡量构成元细胞的细胞之间细胞类型的一致性。
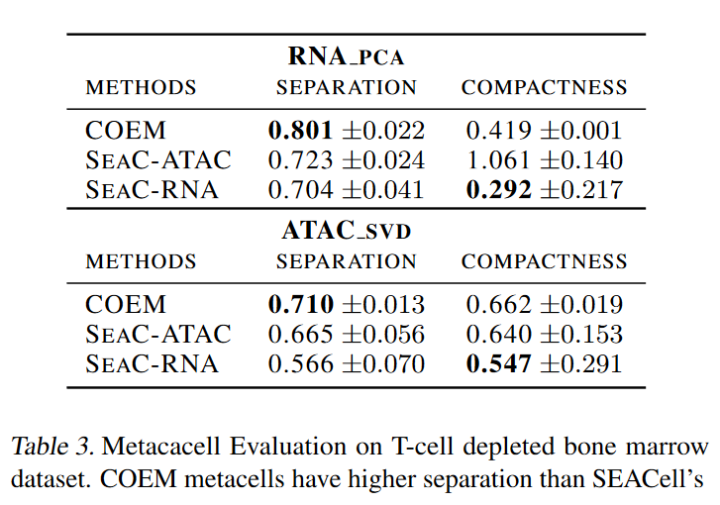
我们使用来自RNA测定(RNA_PCA)的主成分、或来自ATAC测定(ATAC_SVD)的奇异值计算扩散成分,然后量化元细胞的致密性和分离度。由于SEACells从一个模态识别元细胞,我们将它们表示为SeaC-ATAC和SeaC-RNA。
我们评估了sci-CAR数据集中的细胞类型纯度,来自COEM算法的元细胞比SeaCATAC(0.67)和SeaC-RNA(0.74)具有更高的纯度(0.91),这表明利用两种模式提高了我们定义细胞状态的能力。
(sci-CAR 是有标签的数据集)
添加图片注释,不超过 140 字(可选)
然后测量了具有离散细胞类型的数据集之间的致密性和分离度。通常,基于COEM的方法比表2、3和4至6中所示的SeaC-ATAC和SeaC-RNA更好地分离元细胞。但是它们可能仅基于一种模态具有更高的紧致度,因为COEM同时平衡了两种模态的细胞可变性。SEACells识别的元细胞具有较低的紧致度,因为元细胞识别和紧致度评估基于来自单个模态的扩散成分。但这些元细胞可能不能很好地分离和压实,因为它们的扩散成分来自于其他模态。
因此,使用仅从一种模态推断的元细胞来分析基因表达和峰值可及性之间的关联可能会有偏差。此外,基于低维嵌入z,谱聚类在识别高细胞类型纯度和分离度的元细胞方面优于原型分析。我们还测量了原型分析和谱聚类使用相同的低维嵌入z的运行时间,如表1所示。我们可以看到,谱聚类在四个数据集中的运行时间通常要短一个数量级。
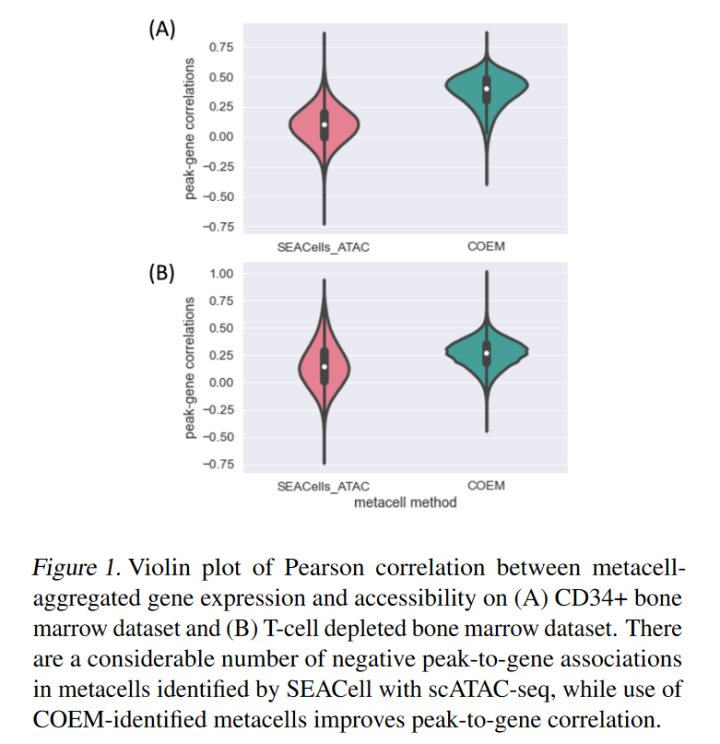
通过分析染色质可及性和基因表达,我们可以推断开染色质峰和转录活性之间的关系,这有助于COEM在CRE的发现。元细胞可以作为一种明智的策略,具有高分辨率和足够的测序覆盖范围,用于从单细胞数据进行基因调控推断。我们通过计算每个峰值在基因+/- 100kb范围内的Pearson相关性来评估跨元细胞染色质可及性和基因表达的共变异。我们观察到SeaC-ATATC metaccells (CD34+骨髓:25.1%;t细胞耗尽骨髓:23.0%)(图1),而在骨髓中发现一小部分负性峰值与基因关联(CD34+骨髓:1.3%;t细胞耗尽骨髓:3.0%),这与单细胞水平上报道的负峰与基因关联比率(1.2 ~ 11%)一致。这表明分化过程中“时间滞后”现象的偏差在SEACells metacells中被夸大,而在COEM中没有。此外,基于COEM元细胞,我们可以观察到更强的核心基因峰-基因相关性。例如,GATA2基因是造血干细胞和祖细胞增殖和维持的主要调控因子。来自CD34+骨髓数据集的COEM元细胞中,GATA2的峰值与基因的相关性为0.73,而在SeaC-ATAC元细胞中,其相关性为0.62。(在单细胞水平上,GATA2峰峰与基因的相关性为0.1)
注:数据分析这里不是很明白
个人观点:
这篇文章篇幅很短,用的模型和方法原创性不高,主要是结合了VAE和谱聚类,所做的metacells identification,因为我对metacell的定义和意义暂时不是很清楚,所以有点迷糊,不知道这样做的意义和优势?我之前主要看的是单细胞数据整合的内容,最多做到聚类这样的下游任务,来判断方法的性能,还没有看到针对元细胞聚类的文章,感觉是个不错的可以结合的下游任务(可以用来丰富文章内容)。
今天的文章COEM: Cross-Modal Embedding for MetaCell Identification「建议收藏」分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/85791.html