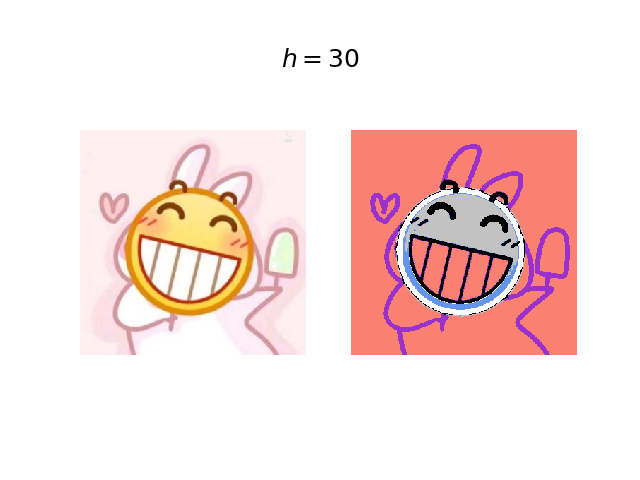前言
前面几篇博客已经介绍过了基于距离的聚类算法 ( ( (K-Means、K-Means++和MeanShift ) ) )和基于密度的聚类算法 ( ( (DBSCAN ) ) ),当然,除此之外还有像层次聚类、谱聚类等这些聚类算法还没有学习到,以后若涉及到再做记录。本篇博客就主要借助机器学习中常用的一个库——scikit-learn来实现图像分割,没错,就是我的头像b( ̄▽ ̄)d
这张图片 ( 320 ∗ 320 ) (320*320) (320∗320)是三通道的,相当于数据的维度为3,由此就根据像素值 ( ( (颜色 ) ) )来实现聚类,即分割图像。
1. K-Means分割图像
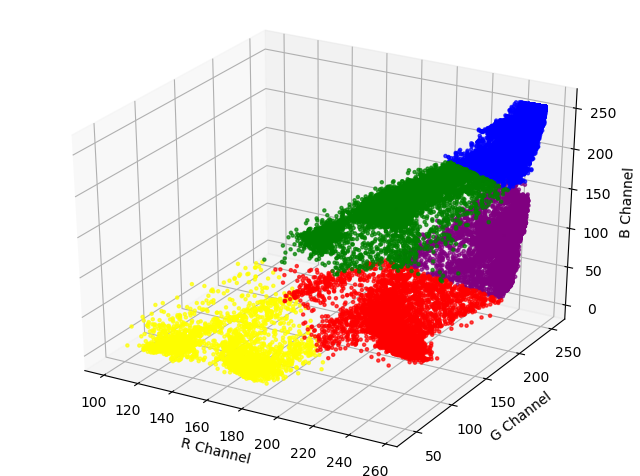
为了与前面几篇博客保持一致,先在坐标轴上简单看一下聚类的结果,只不过是将前面的二维坐标变成了三维坐标 ( R , G , B ) (R,G,B) (R,G,B),代码如下:
import numpy as np
from sklearn.cluster import KMeans
import imageio
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
def draw_picture(data_arr, cluster_centers, labels):
dots1 = data_arr[labels == 0]
dots2 = data_arr[labels == 1]
dots3 = data_arr[labels == 2]
dots4 = data_arr[labels == 3]
dots5 = data_arr[labels == 4]
fig = plt.figure()
ax = Axes3D(fig)
ax.scatter(dots1[:, 0], dots1[:, 1], dots1[:, 2], marker='o',
color='blue', alpha=0.7, label='dots1 samples', s=5)
ax.scatter(dots2[:, 0], dots2[:, 1], dots2[:, 2], marker='o',
color='green', alpha=0.7, label='dots2 samples', s=5)
ax.scatter(dots3[:, 0], dots3[:, 1], dots3[:, 2], marker='o',
color='red', alpha=0.7, label='dots3 samples', s=5)
ax.scatter(dots4[:, 0], dots4[:, 1], dots4[:, 2], marker='o',
color='purple', alpha=0.7, label='dots4 samples', s=5)
ax.scatter(dots5[:, 0], dots5[:, 1], dots5[:, 2], marker='o',
color='yellow', alpha=0.7, label='dots5 samples', s=5)
ax.scatter(cluster_centers[:, 0], cluster_centers[:, 1], cluster_centers[:, 2], marker='x',
color='black', alpha=0.7, label='centroids', s=5)
ax.set_xlabel('R Channel')
ax.set_ylabel('G Channel')
ax.set_zlabel('B Channel')
plt.savefig('./result_3d_s.png')
plt.show()
if __name__ == '__main__':
img = imageio.imread('./touxiang.png')
img_flatten = img.reshape(-1, 3)
kmeans = KMeans(n_clusters=5, init='k-means++', n_init=10, max_iter=300,
tol=1e4, verbose=0, random_state=1024, n_jobs=1)
kmeans.fit(X=img_flatten)
cluster_centers = kmeans.cluster_centers_
labels = kmeans.labels_
draw_picture(img_flatten, cluster_centers, labels)
上面的参数在这篇博客中有介绍,在这里就不过多解释了,结果如下:
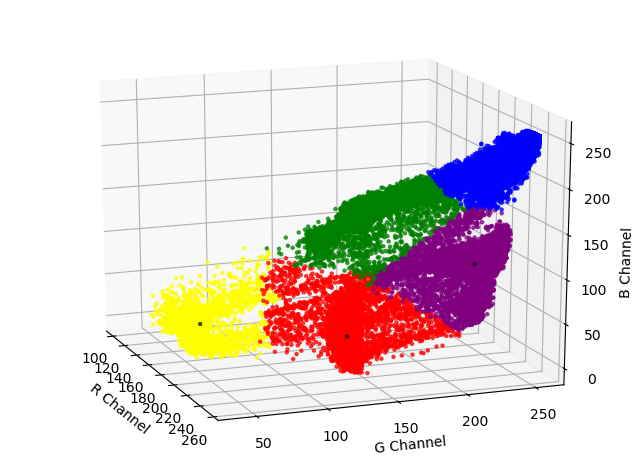
在Pycharm中默认显示的3D图像是不可以旋转的,可以通过如下设置:File − − > –> −−>Settings − − > –> −−>Tools − − > –> −−>Python Scientific − − > –> −−>Show plots in tool window ( ( (前面的√去掉 ) ) ) − − > –> −−>Apply − − > –> −−>OK,然后通过旋转就可以看到聚类中心了 ( ( (即图中黑色圆点 ) ) )。
OK,fine,其实图像的结果也和上面一样了,只不过不把它放到坐标轴上,而是以数组的形式存起来,将下面的seg_img函数替换掉上面的draw_picture即可,代码如下:
def seg_img(img, labels):
labels = labels.reshape(320, 320)
img_seg = np.array(np.zeros(shape=(320, 320, 3), dtype=np.uint8))
img_seg[labels == 0] = [250, 128, 114]
img_seg[labels == 1] = [194, 194, 194]
img_seg[labels == 2] = [153, 50, 204]
img_seg[labels == 3] = [255, 255, 255]
img_seg[labels == 4] = [100, 149, 237]
plt.subplot(121)
plt.axis('off')
plt.imshow(img)
plt.subplot(122)
plt.axis('off')
plt.imshow(img_seg)
plt.savefig('./result_5k.png')
plt.show()
如果划分为四个类别,就是这样子的:
如果划分为五个类别,就是这样子的:
注:上面的颜色会随机分给各个类别。
2. Mean Shift分割图像
实现流程和上述一样,只不过函数变成了MeanShift,这里只贴出改动的部分,代码如下:
MeanShift函数的部分参数已注释,更详细的说明,请参考官方文档。
from sklearn.cluster import MeanShift
if __name__ == '__main__':
img = imageio.imread('./touxiang.png')
img_flatten = img.reshape(-1, 3)
# bandwidth: 半径(或带宽), float型, 默认None
# 如果没有给出, 则使用sklearn.cluster.estimate_bandwidth计算出半径(带宽)(可选)
# seeds: 圆心(或种子), 数组类型, 即初始化的圆心, 默认None(可选)
# 如果未设置, 则通过clustering.get_bin_seeds计算种子, 并使用带宽作为网格大小以及其他参数的默认值
# bin_seeding: 布尔值. 如果为真, 初始内核位置不是所有点的位置, 而是点的离散版本的位置, 其中点被分类到其粗糙度对应于带宽的网格上
# 将此选项设置为True将加速算法, 因为较少的种子将被初始化. 默认值: False, 如果种子参数(seeds)不为None则忽略
# n_jobs: 任务使用的CPU数量
# 如果bandwidth不设置, 代码运行会消耗较多时间
meanshift = MeanShift(bandwidth=20, seeds=None, bin_seeding=True,
min_bin_freq=10, cluster_all=True, n_jobs=1)
meanshift.fit(X=img_flatten)
cluster_centers = meanshift.cluster_centers_
labels = meanshift.labels_
seg_img(img, labels)
不同的半径会有不同的结果,可以去尝试不同的设置,部分结果如下:
搞错了,再来(づ。◕ᴗᴗ◕。)づ(感觉还是蛮像的!哈哈哈哈哈哈哈哈哈)
3. DBSCAN分割图像
实现流程和上述一样,只不过函数变成了DBSCAN,这里只贴出改动的部分,代码如下:
DBSCAN函数的部分参数已注释,更详细的说明,请参考官方文档。
from sklearn.cluster import DBSCAN
if __name__ == '__main__':
img = imageio.imread('./touxiang.png')
img_flatten = img.reshape(-1, 3)
# eps: 两个样本之间的最大距离, 即半径, 默认为0.5
# min_samples: 作为核心点的邻域(即以其为圆心, eps为半径的圆, 含圆上的点)中的最小样本数(包括点本身)
# 即MinPts, 默认为5
# 其他参数可参考官方文档
dbscan = DBSCAN(eps=0.5, min_samples=5)
dbscan.fit(X=img_flatten)
labels = dbscan.labels_
seg_img(img, labels)
运行结果如下:
对于图片来说,运行较占内存,可能上面的参数还要再配置一下。然后我又多尝试了几次,结果MemoryError了∑(っ°Д°;)っ卧槽,无情。官方是这样说的:
大样本量的内存消耗
默认情况下,此实现的内存效率不高,因为它在无法使用kd树或球树的情况下 ( ( (例如,稀疏矩阵 ) ) )构造了完整的成对相似度矩阵。该矩阵将消耗 n 2 n^2 n2个浮点数。解决此问题的几种机制是:
- 将OPTICS聚类与该extract_dbscan方法结合使用 。OPTICS集群还可以计算完整的成对矩阵,但是一次只能在内存中保留一行 ( ( (内存复杂度 n ) n) n)。
- 可以用内存有效的方式预先计算稀疏半径邻域图 ( ( (假定缺少的条目不在eps之内 ) ) ),而dbscan可以使用metric =’precomputed’在其上运行。
- 可以压缩数据集,方法是删除精确重复项 ( ( (如果数据中出现重复项 ) ) ),或者使用BIRCH。这样,对于大量的点,就只有相对较少的代表。然后,可以在拟合DBSCAN时提供一个sample_weight。
我尝试用官方的方法去设置,然而并没有有效解决,然后受到上面第三条的启发,我就把图像缩放到了 128 ∗ 128 128*128 128∗128,问题得以解决。
为了方便调参,将数据进行了归一化处理,修正代码如下:
img_flatten = img.reshape(-1, 3) / 255.0
调整后的运行结果如下:
eps越大,聚类的类别就越少;MinPts越大,聚类的类别也越少。根据经验,最好将MinPts设置为数据中特征的数量,eps使用小值通常是首选。
结束语
本篇博客在使用DBSCAN时出了一些问题,图像的尺寸有些大,处理不了,然后就将图像进行放缩了。另外,由此也可以看出DBSCAN算法的弊端,就是参数需要不断去调整,相对来说还是较麻烦的,不过我看网上有说可以借助k-distance图来找最佳的eps,当然了官方也说有优化的方法,这些还有待探索。继续加油ヾ(◍°∇°◍)ノ゙
今天的文章大黄脸头像_图像分类算法分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/67422.html