AutoDock分子对接教程 小分子对接
文章目录
前言
提示:软件安装以及软件安装报错这里就不多说了, 大家可以查看之前写好的文章
-
AutoDock与Vina的区别 如何选择AutoDOCK与Vina对接 AutoDock对接系列 run autogrid出现[Error 2]从而得不到.map文件 AutoDock安装闪退
-
Linux系统安装AutoDockTools、AutoGrid和AutoDock并实现分子对接 分子对接软件 CentOs Ubuntu系统适用(详细讲解)
下面讲一下AutoDock Tools的对接:
一、蛋白获取
使用PubChem搜索需要的小分子,在对应结果的structure处下载即可,这里推荐下载sdf格式文件
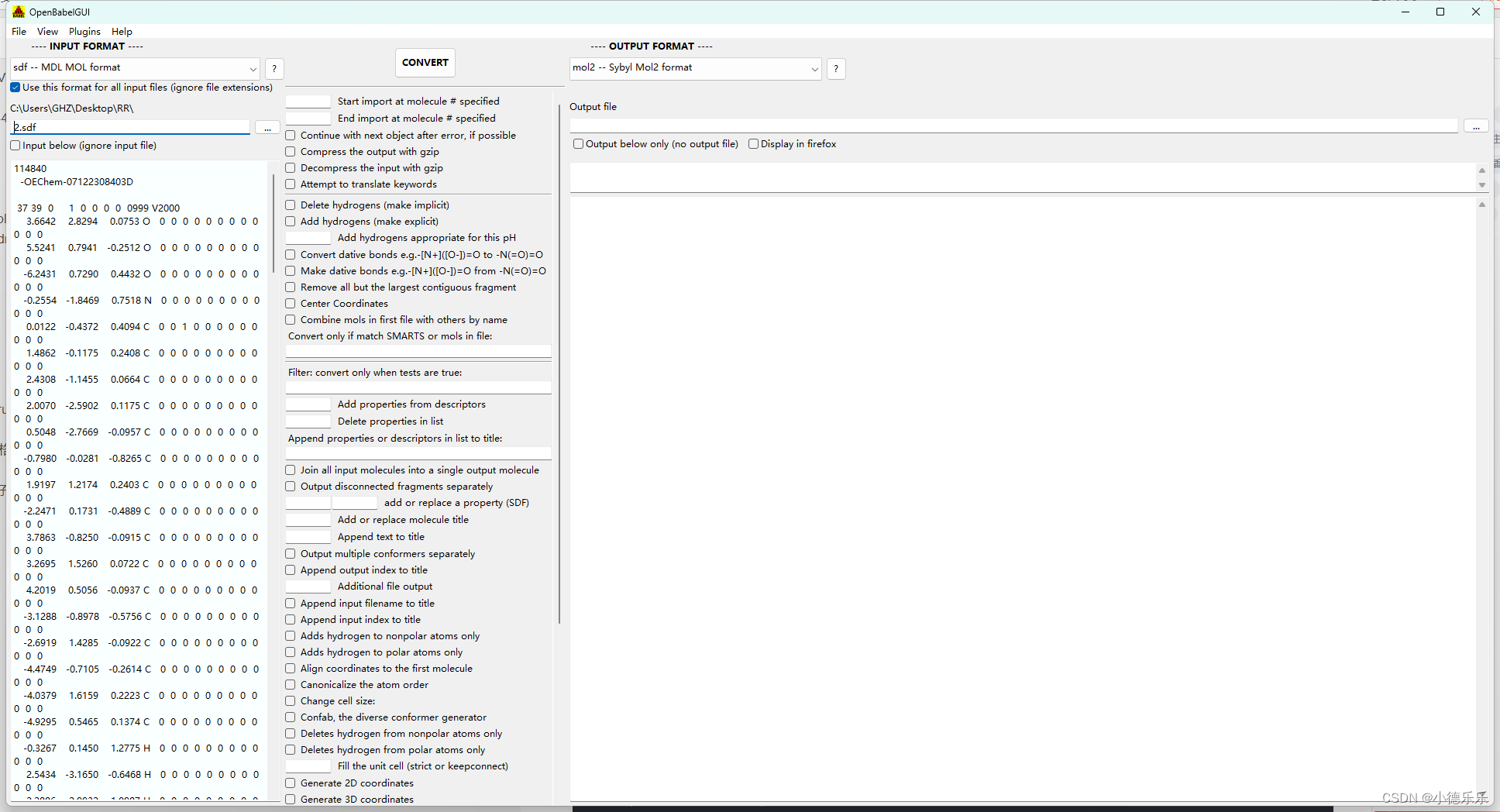
下载得到小分子三维结构后,使用OpenBabel将sdf格式文件转换为Mol2格式文件。
OpenBabel的使用如下面的图,左右两边分别选择好输入输出文件的格式和对应路径,然后点中间的CONVERT即可:
二、使用步骤
软件预处理:
-
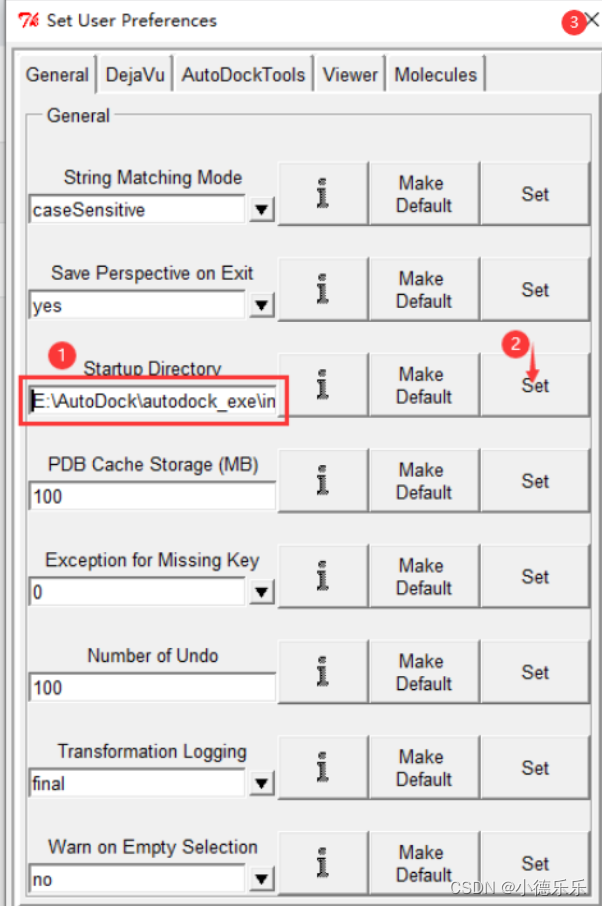
双击打开后,首先会弹出一个黑色窗口,这个窗口在运行autodock的过程中由始至终都会存在,注意不要关闭,否则将会自动关闭autodock
- 将蛋白质pdb文件和小分子pdb文件放在同一个目录下,注意路径中不能包含中文!!! 将软件运行目录设置在蛋白质和小分子所在目录中
- 在All Molecules处点击右键,选择Read molecule,打开蛋白质pdb文件
提示:到这里初步工作就完了
去水:
随后需要将pdb文件中的水分子移除(该操作只能移除水分子,如果pdb文件中还有其他小分子的建议使用pymol手动删除)
注意:只有从PDB数据库中下载的实验结构才会包含水分子或其他小分子,使用alphafold预测的蛋白质结构不包含水分子,可跳过该步
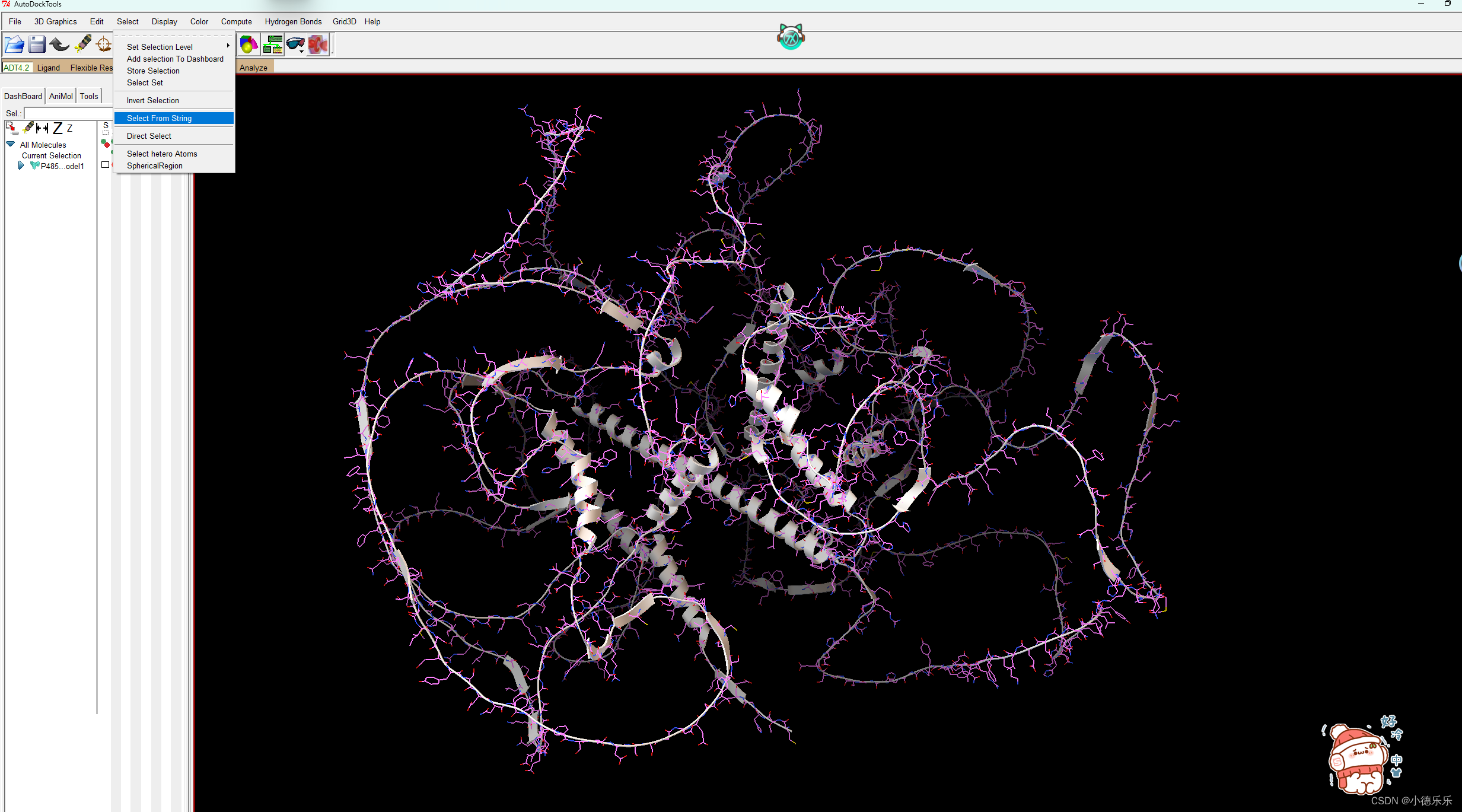
Select->Select From String
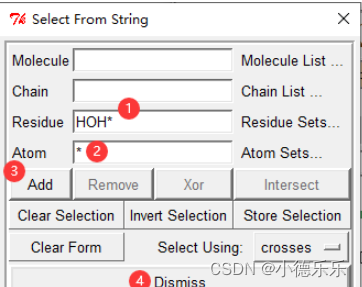
在弹出窗口中,Residues中键入HOH*,Atoms中键入*,然后分贝点击Add和Dismiss即可
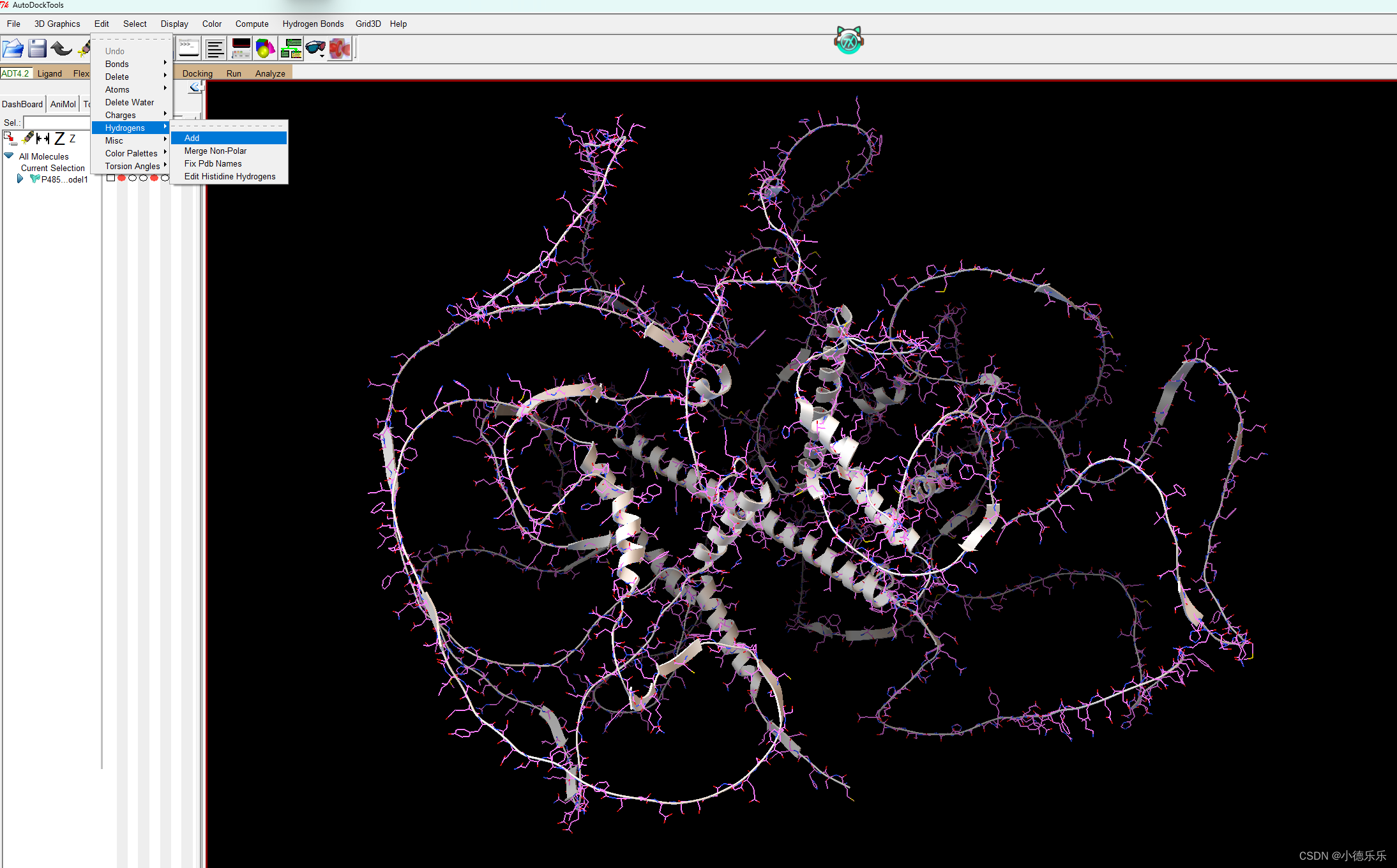
加氢:
无论是PDB还是Alphafold,下载得到的蛋白质PDB结构都只包含碳链骨架,因此需要把氢原子给补上
依次点击Edit->Hydrogens->Add
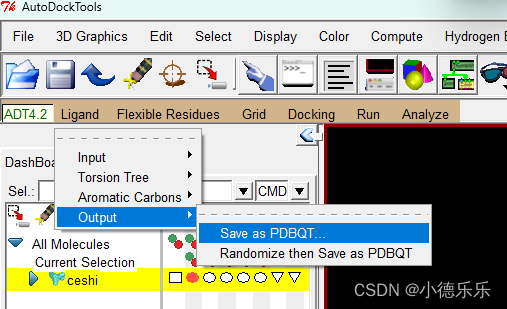
配体小分子准备
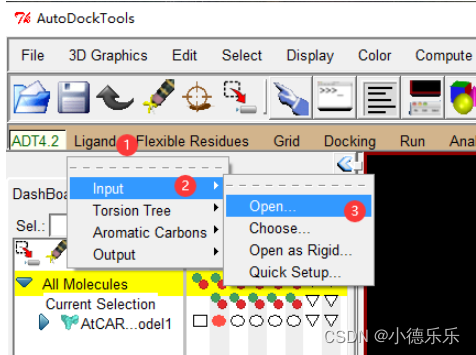
打开配体小分子pdb文件
注意这里文件默认后缀名是.pdbqt,我们输入的是.pdb文件,需要选择一下可用文件类型,all files或者.pdb都可以
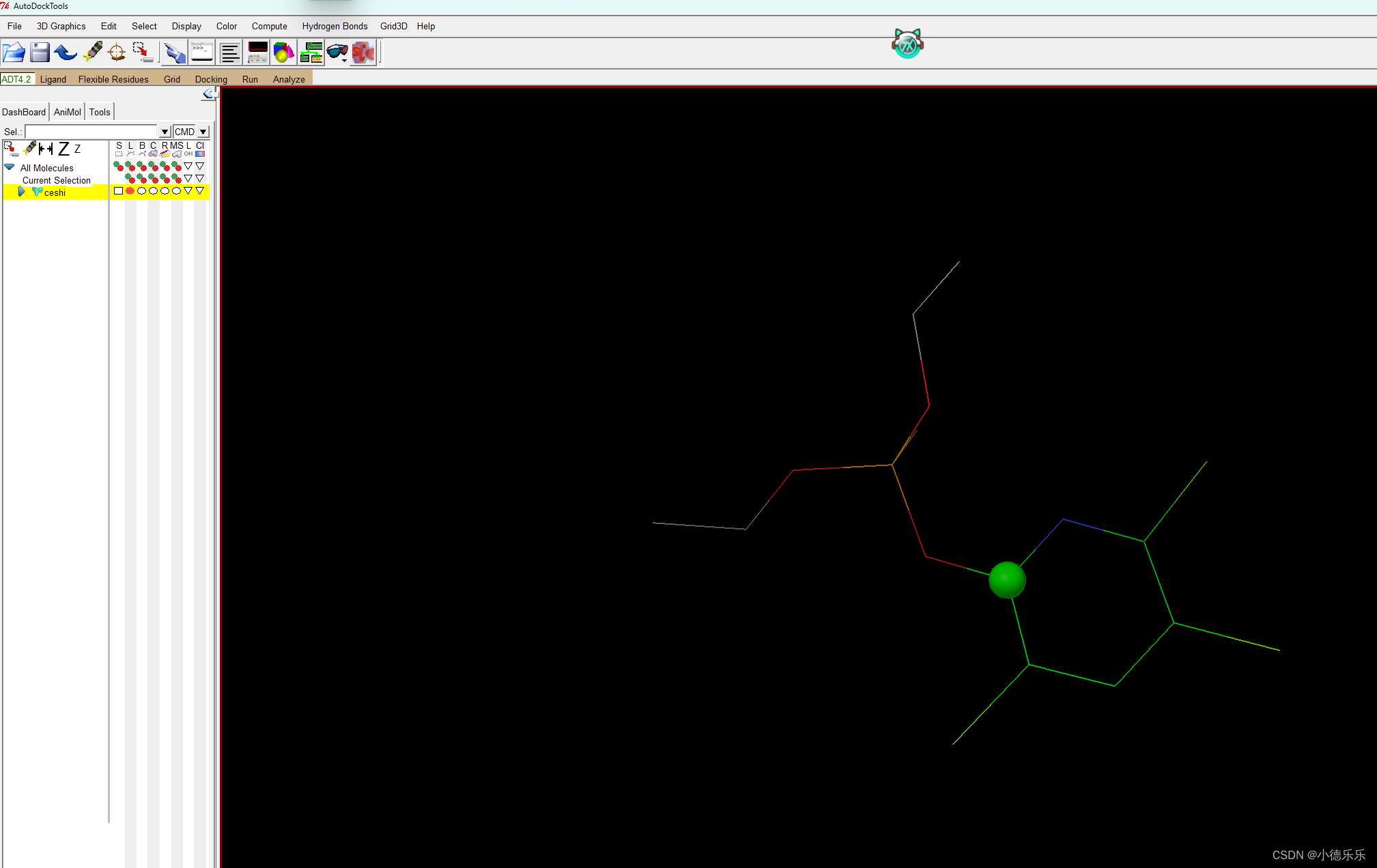
接下来需要设置小分子扭转
点击后将小分子放大可以看到有一个原子被绿色球所包裹,该原子即为中心
按住左键移动鼠标为旋转;按住鼠标右键移动鼠标为放大缩小;按住鼠标中键即滚轮移动鼠标为移动位置
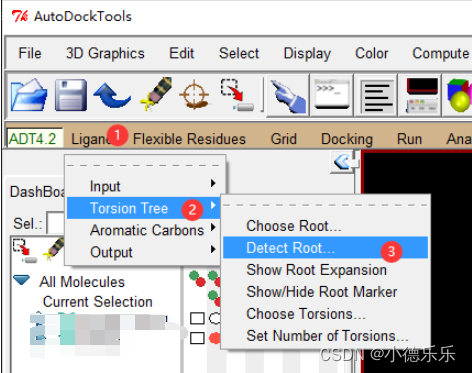
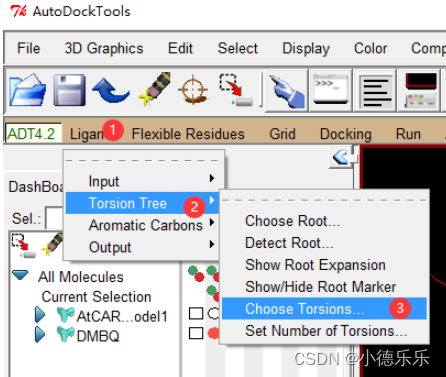
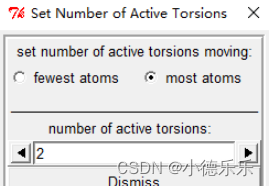
通过如下方法可以设置分子中哪些键是可以扭曲的
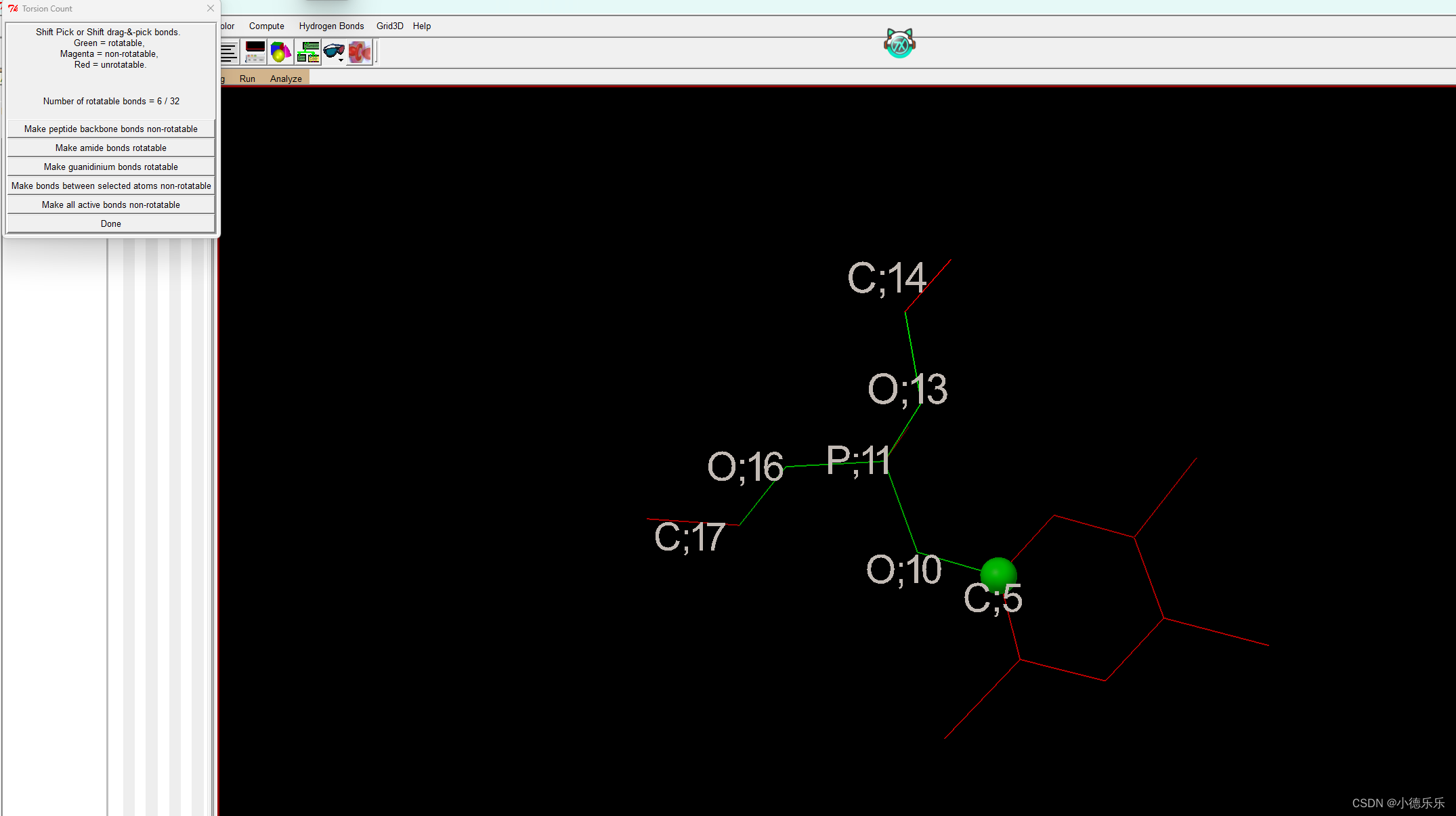
扭曲规则是根据化学性质来的,当然你可以可以根据个人需要设定哪些键可以扭曲哪些不能扭曲,如果你啥也不懂那就不用动了。选择完毕后就Done即可。
这里可以扭曲的原子越多后面需要消耗的计算资源也越多,时间越长。设置好后Dismiss就行。
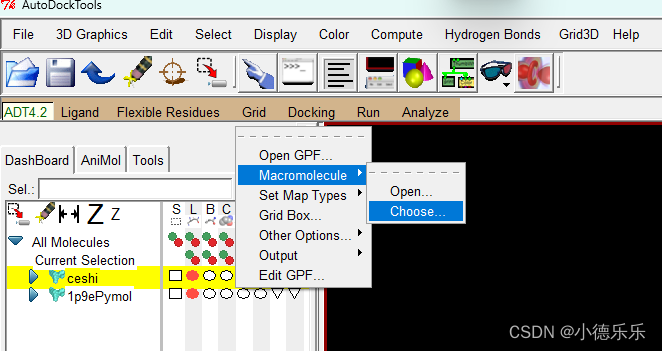
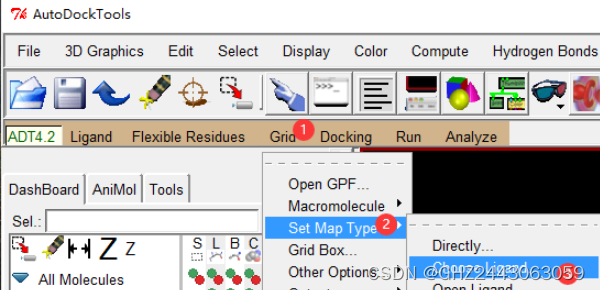
受体分子准备
选择目标蛋白质区域进行分子对接
加氢去水后:
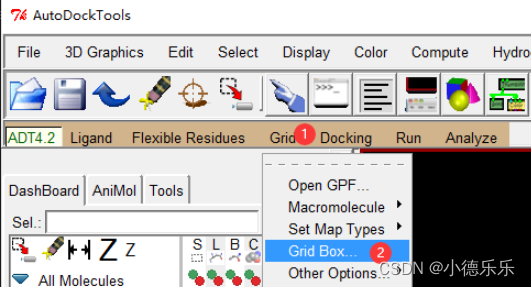
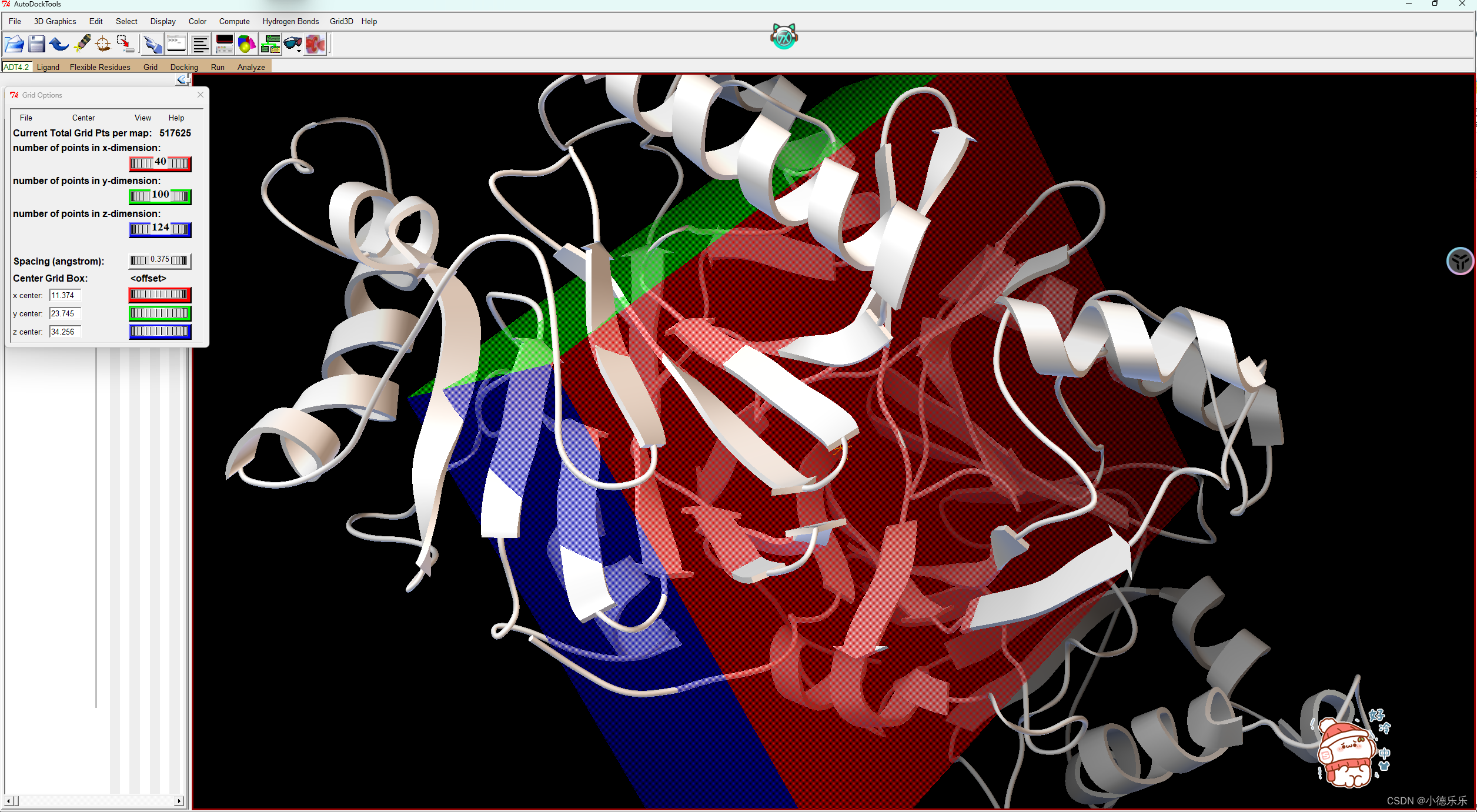
然后打开Grid Box,选择目标蛋白质区域
知道分子对接可能的作用口袋,那你只需要选择该口袋进行对接就可以了。如果不了解的话选择全蛋白即可。
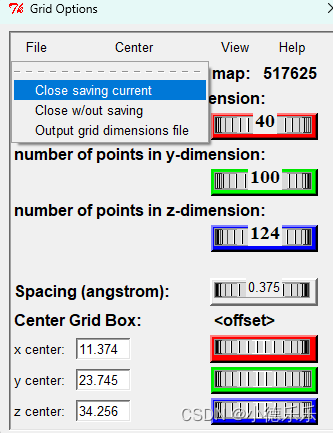
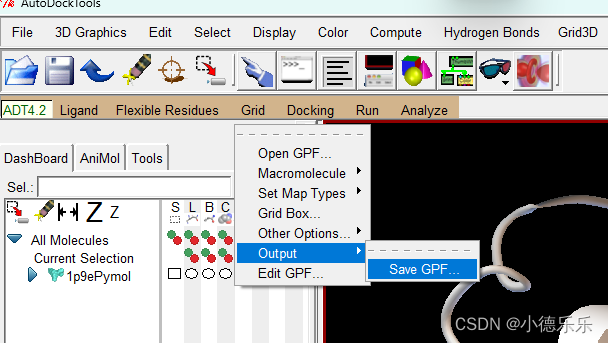
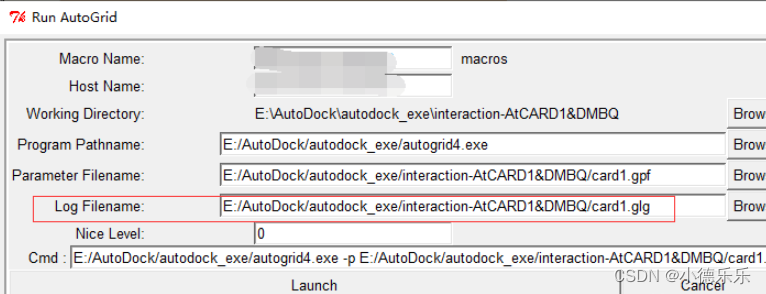
输出grid设置文件。建议保存位置就在跟大分子和小分子同一目录下,不要放其他地方。路径不能有中文。同时在输出文件名的时候记得手动给他把后缀名.gpf加上
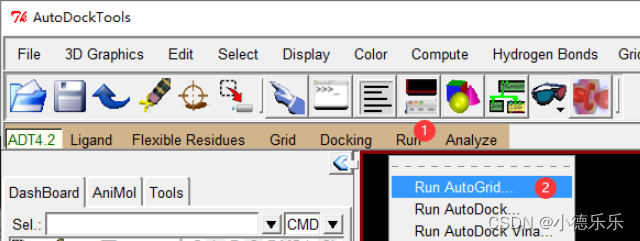
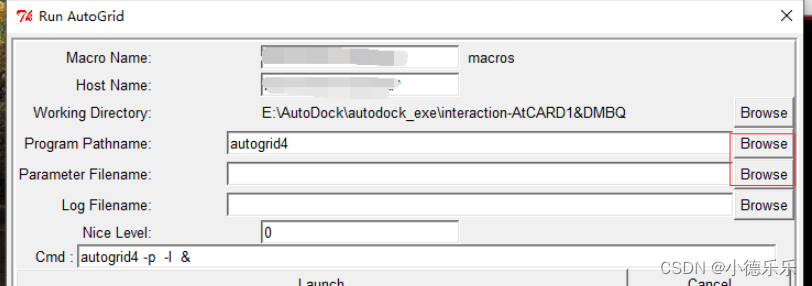
运行AutoGrid
此时点Launch即可。如果前面的.gpf文件没有后缀名这里可能会出错
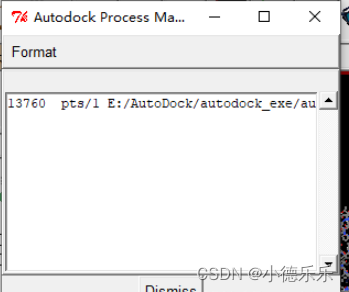
开始运行后会出现这样一个小窗,说明程序正在运行,小窗消失说明运行完成。
如果你的这个小窗在十秒内消失了那你可能要反思一下是不是之前有哪步做错了,比如在刚开始的时候软件的运行路径设置错了,没有设置在大小分子的目录下。
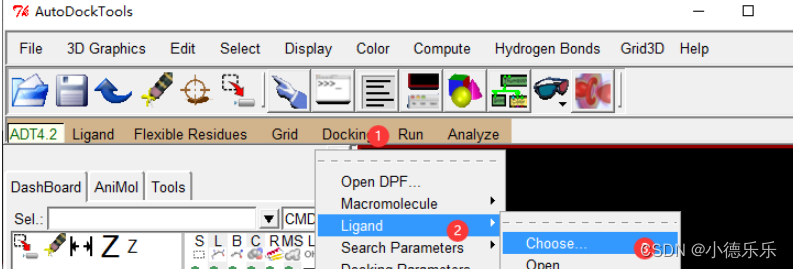
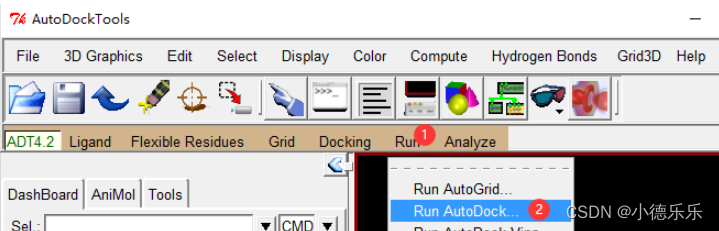
运行AutoDock
选择小分子配体文件,弹出的窗口accept就行:
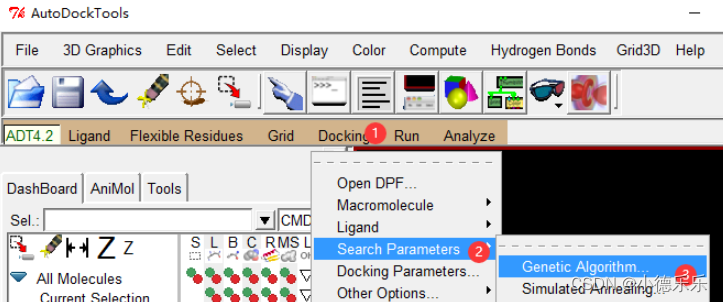
选择对接算法,我个人一般会用遗传算法,读者也可以自行尝试其他算法:
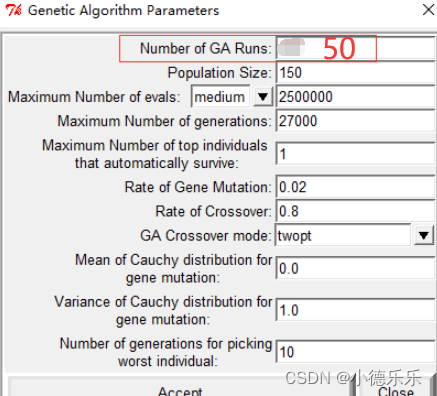
在弹出的窗口中修改Number of GA Runs,这里指的是后面要重复对接多少次,次数越多消耗时间越长,结果相对更可靠,这个数字一般不要低于50,设置好后accept:
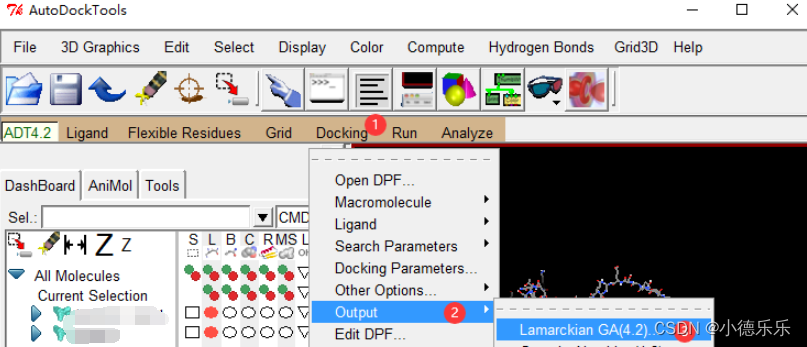
设置完毕后输出.dpf文件,注意这里选择的是Lamarckian GA而不是刚刚的Genetic Algorithm。
跟gpf一样,dpf的后缀名也需要自己加一下。
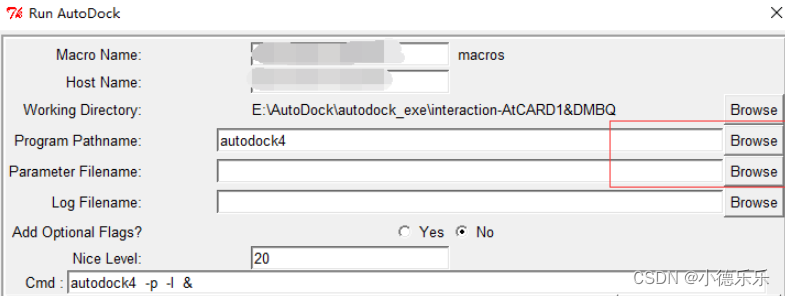
设置好autodock4.exe的路径和.dpf文件路径,log filename一行即可自动生成,然后Launch
同样会出现一个小窗,小窗消失即运行完成,前面设置的重复次数越多这一步越耗时
对接结果查看测试
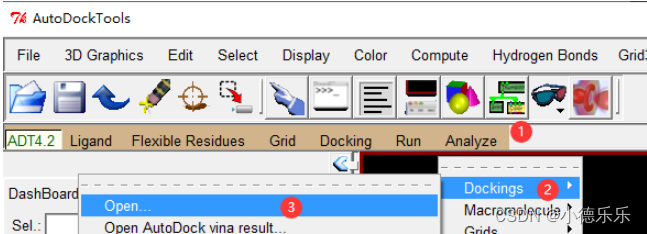
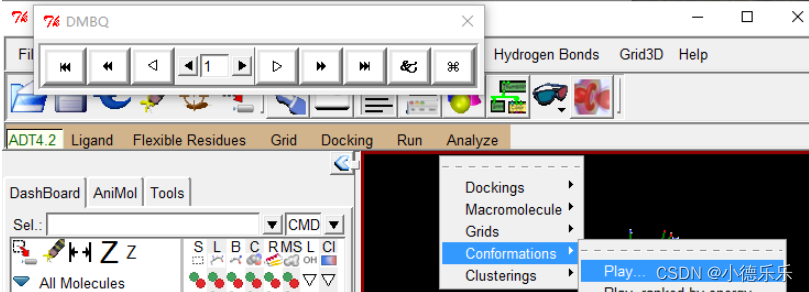
打开刚刚生成的.dlg文件
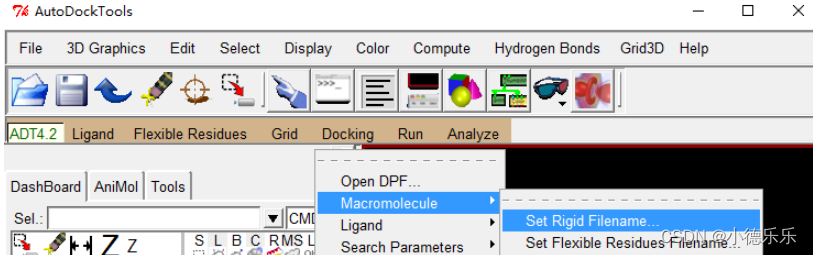
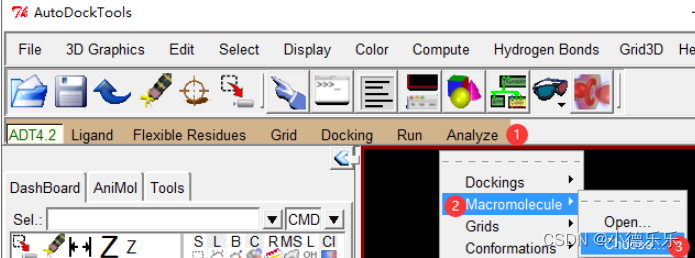
选择大分子文件:
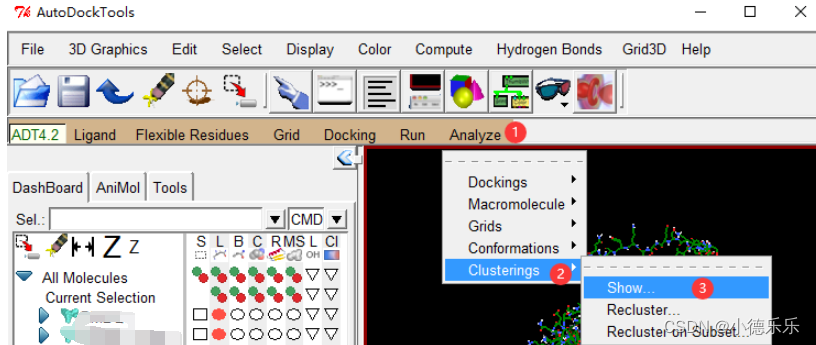
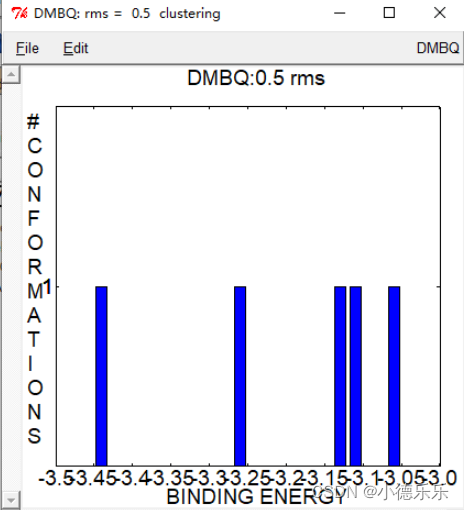
对接结果构象进行聚类分析:
该柱状图表示某一类构象有多少个相似的对接结果,如果在数次重复中小分子都对接到了同一口袋即使小分子扭曲构象略有区别,也会被聚类到一起,纵轴表示频数,数目越多表示该构象越稳定,横轴则是能量,能量越低越稳定
像这里展示的这个构象如果在对接次数足够多的情况下依然每种都只有一个那就说明对接结果不太可靠
(因为Autodock功能比较简单,对接结果较差不能说明该分子与蛋白就一定没有互作)
单击柱状图某一根柱子该柱子会变为红色,相应构象也会显示
可以选择合适的构象进行保存,输出的文件记得加后缀名.pdbqt,使用Openbabel转为.pdb文件,可以使用pymol对.pdb文件进行可视化,并进一步分析相互作用:
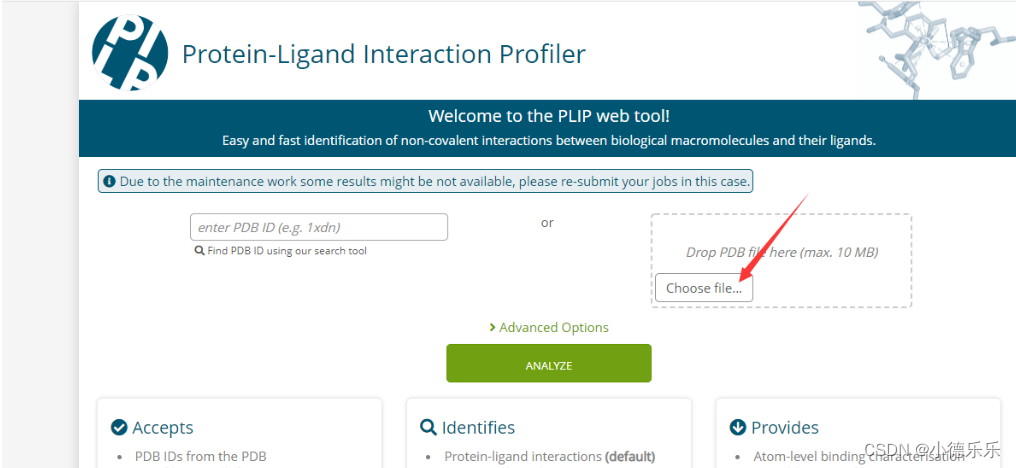
三、使用PLIP分析相互作用
使用PLIP分析对接结果,该网站使用方法非常简单,只需要直接输入对接结果的pdb文件,然后点击Analyze:
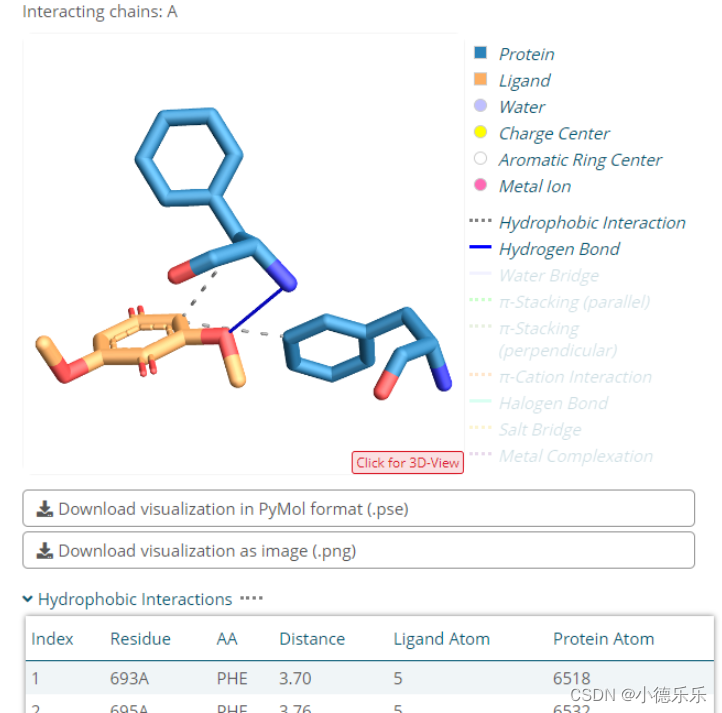
在输出结果中,会展示包括氢键等各种常见的相互作用力,并注明蛋白质中具体哪个氨基酸残基与小分子互作,方便用户后续进行点突变等操作
四、与我联系——解决AutoDock对接报错以及闪退,提供对接教学服务。
PC端电脑通过
点击PC端分子对接软件合集——“能看到某宝对应的分子对接软件商品!!!。
手机淘宝通过:
点击手淘分子对接软件合集 “——能看到某宝对应的分子对接软件商品!!!
五、总结
这一篇文章为参考别人的文章给对方改的,因为看了以后发现对方写的顺序和一些细节错误太多,就顺手改了一下。
注意:对方的包括我改的,我并没详细到每一步都截图给出来,改了一些对方的错误,还有的错误大家遇到了再来问我就行。因为要每一步细节的给出来,不如来购买我这边录制好的视频,截图加文字是在描述不来,请大家看得懂的看,看不懂的见谅哈
参考:Eumenidus这个博主的文章:https://blog.csdn.net/Eumenidus/article/details/124064090
今天的文章autodock对接结果分析_我的世界模组工具mcpE.A分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/82002.html