DOI: 10.3389/fmicb.2022.1032186
通讯作者:孙燕妮
主要单位:香港城市大学电机工程系
Department of Electrical Engineering, City University of Hong Kong, Hong Kong, China
– 背景与研究目的 –
噬菌体是感染细菌的病毒,种类十分丰富且广泛存在于不同生态环境中,包括海洋、土壤和人体等。然而,尽管噬菌体在各种生态系统中非常重要,我们对噬菌体的了解仍然有限。将噬菌体归入不同的分类组是噬菌体相关研究的一个基本步骤。官方的分类法由国际病毒分类委员会(ICTV)建立,将病毒分为几个分类层次,包括界(kingdom),门(phylum),纲(class),目(order),科(family),属(genus),种(species)等等。事实上,噬菌体分类不是一个小问题。随着今年来大量新噬菌体的发现,ICTV的分类标准也在不断变化。最近,ICTV在2022年8月更新了噬菌体分类系统,删除了有尾噬菌体目中多个主要的科,如Siphoviridae、Podoviridae和Myoviridae,这些变化会大大影响到科级分类的性能。据我们所知,目前还没有文章对这些分类变化进行定量评估。作者使用Dashing比较新旧ICTV分类系统中最大的四个科的平均相似度后发现,更新后的科更加保守,这可能使科级分类比以往更加容易。
近年来发布了很多噬菌体分类工具,并被证明有较好的分类表现。然而,现有的分类工具通常具有不同的设计,并由其开发者在不同的数据集上进行测试。如果没有在相同的训练/参考数据集和测试集上的综合比较,用户很难选择最符合他们需求的解决方案。因此,本文的主要工作是根据最新的ICTV标准,对噬菌体分类工具在有尾噬菌体的科级分类上进行全面的基准测试。文章首先介绍现有噬菌体分类工具的主要方法和模型,并讨论它们在新的ICTV分类标准下被重新训练使用的可行性。然后我们在不同难度的数据集上对四个可以进行再训练的工具进行测试评估。用到的数据集包括:完整的序列(complete genomes)、序列短片段(short contigs)、模拟宏基因组数据集(simulated metagenomic dataset)和低相似度数据集(low-similarity dataset)。此外,我们还进行了 “排除某一科类(leave-one-family-out)”实验,以测试这些工具是否能识别已知分类之外的序列。通过比较它们的实验结果并分析内在原因,我们为用户提供了在不同情况下选择最合适的工具的指导。
– 实验结果 –
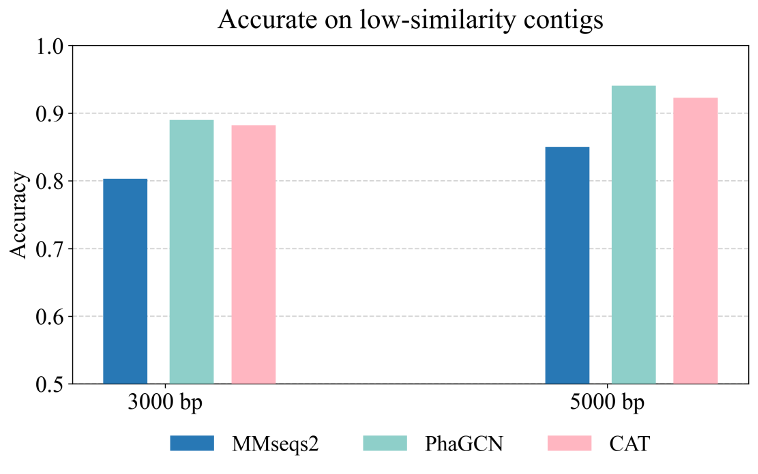
虽然目前有不少工具可以进行病毒分类,但是大部分工具不支持用新的ICTV分类标准进行重新的训练。基于这个要求,文中选择PhaGCN, vConTACT 2.0, CAT, 和 MMSeqs2 四个工具进行测试。PhaGCN是基于机器学习的分类工具,将DNA序列特征和蛋白质序列相似性相结合构建网络,利用模型来推断各分类的序列特征从而进行节点分类。其他工具则利用序列比对方法将测试数据与参考数据库进行比较来进行分类。考虑到属于同一科内会存在差异度较大的序列,我们设置了难度较高的实验条件,降低训练集和测试集的相似度并观察工具在该测试集上的分类表现。实验结果表明,PhaGCN的分类性能受到的影响较小,准确率仅下降2%-3%,相比之下,差异度较大的数据集对MMseqs2和CAT的分类效果影响更为明显,整体准确率下降分别为10%和6%左右。具体的结果请见图1。因为MMseqs2和CAT是基于序列比对的分类工具,分类效果更依赖于参考数据集的质量,难以分类差异度较大的数据。而PhaGCN使用机器学习方法提取分类特征,并不完全依赖于参考数据库,因此性能在分类与参考基因组相似度低的序列时受影响较小。从结果可以看出,基于机器学习方法的工具在对新的或高度差异的噬菌体序列进行分类方面具有内在的优势,尤其是对于那些参考数据数量不足的噬菌体类别更加容易分类。
图1:MMseqs2, PhaGCN和CAT三种工具分别在3000 bp和5000 bp长度的低相似度数据集上的整体分类准确率表现
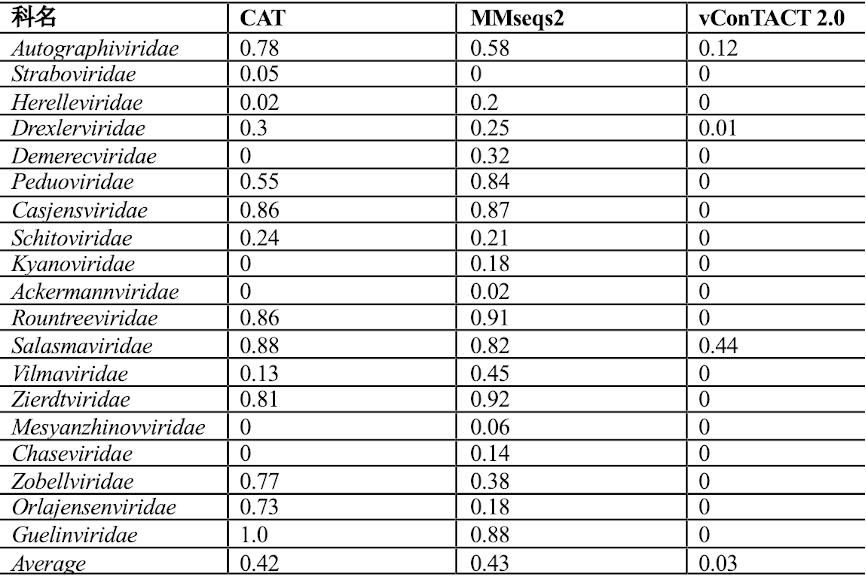
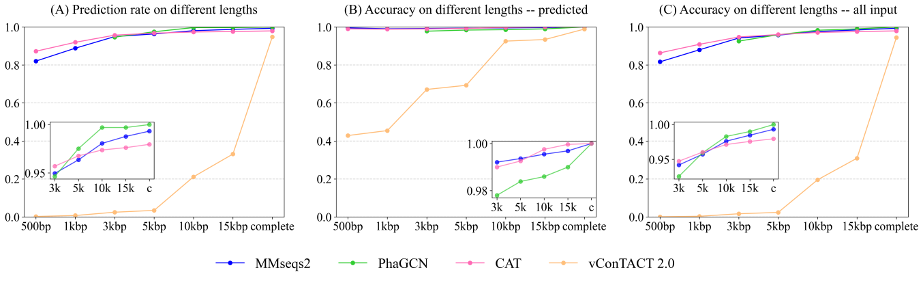
除了测试工具在低相似度数据上的表现,我们还在其他数据集上对四种工具的分类性能进行测试。在“排除某一科类”的实验中(实验一),我们把一个科的所有噬菌体序列从训练数据中移到测试数据中,然后重新训练所有工具。理想情况下,这个被删除的家族中的测试序列不应该被归入任何现有的家族标签。实验结果表明(参见表1),CAT和MMseqs2将更多的测试序列错误地分配给参考数据库中的其他科,而vConTACT 2.0 测试的绝大多数序列都未被分配分类结果。根据进一步的观察,错误分类的可能性较大程度取决于科之间的相似性。因此,vConTACT 2.0在识别未定义分类的噬菌体方面比其他三种工具表现得更好。随后在评估分类性能的实验(实验二)中对比了四个工具在不同长度的测试序列上的分类表现,结果详见图2。由图可见,所有工具在完整序列上的表现都较好,准确率和预测率均高于95%。在短序列片段的测试中,PhaGCN和MMseqs2在短序列上的表现更好。但由于vConTACT 2.0主要是为完整或长序列设计的,当输入的测试序列短于15, 000 bp时,其预测率有大幅度下降。而CAT更倾向于提供较高的精度,并不具有极高的预测率。因此,当用户选择分类工具时,如果序列片段长度超过2,000 bp,建议使用PhaGCN和MMseqs2以获得高预测率。否则,如果精度是首要考虑因素,CAT是更好的选择。
表1:在“排除某一科类”实验中三种工具对于每个科的误分类率
图2:工具在RefSeq数据集上的分类表现。(A): 工具在不同长度序列上的预测率 (B): 工具在不同长度序列上的分类准确率(不包括未输出分类结果的数据) (C)工具在不同长度序列上的总分类准确率(包括未输出分类结果的数据)
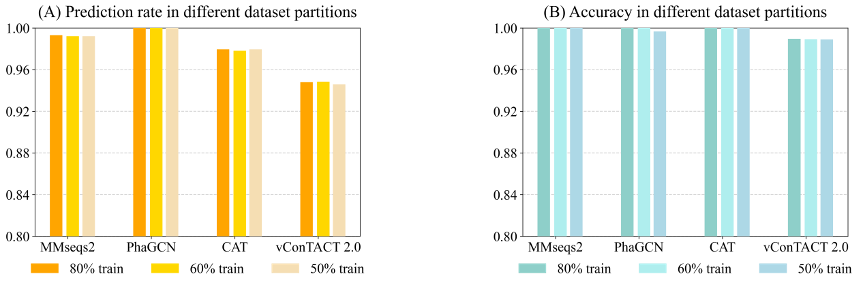
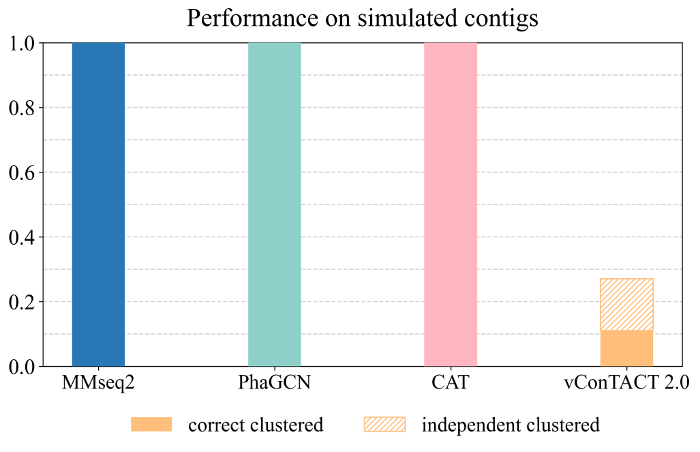
随后进行了调整训练数据集大小的实验(实验三,图3),但实验结果表明四个工具在不同大小的参考数据库中的预测率和准确率没有明显差异。并且四个工具在模拟宏基因数据集上的分类表现(实验四, 图4)也与在实验一中的分类表现具有较高的一致性。除此之外,文中还比较了四个工具的运行时间(表2)。在处理相同数量的数据时,CAT和MMseqs2的运行速度更快。
图3:四种工具分别将80%,60%和50%的RefSeq数据作为训练数据集的分类表现。(A): 预测率 (B): 准确率
图4:四种工具在模拟宏基因组序列数据集上的分类准确率
表2:四种工具分类500条序列数据的所需时间
– 总结 –
综上,根据实验结果以及工具本身的特性,文章可以对用户选择分类工具时提供一些具有参考性的建议:PhaGCN只能在科级(family)对序列进行分类。其他三个工具可以分类的最低级别是属级(genus)或以下。vConTACT 2.0对属于未定义科的序列更敏感,但代价是它对短片段的预测率较低。这四个工具在完整序列上都表现良好,没有较大差异。PhaGCN在对短片段(>3,000 bp)进行分类时具有最高的预测率,CAT的预测率稍低但具有更高的准确率。因此,在对大于3,000bp的不完整片段进行分类时,PhaGCN、CAT和MMseqs2都可以被考虑在内,但PhaGCN的整体性能更好。此外,CAT和MMseqs2可以用来对短于2, 000bp的序列片段进行分类,因为PhaGCN不能处理这个长度。而训练数据的数量减少对这四种工具几乎没有影响。并且,PhaGCN的性能在分类与参考基因组相似度低的高分化序列时表现更好,这也说明基于机器学习的分类方法在噬菌体分类问题中具有一定的内在优势。
参考文献:
Zhu Y, Shang J, Peng C and Sun Y (2022) Phage family classification under Caudoviricetes: A review of current tools using the latest ICTV classification framework. Front. Microbiol. 13:1032186.
– 作者简介 –
香港城市大学
朱益林
研究助理
朱益林:香港城市大学研究助理,主要研究方向是病毒分类工具开发等。目前在Frontiers in Microbiology 有发表相关文章。
香港城市大学
商家煜
博士研究生
商家煜:香港城市大学博士研究生,主要研究方向是深度学习在宏基因组学中的应用。目前在Bioinformatics,Frontiers in Microbiology,BMC Biology,Briefings in Bioinformatics等期刊和ISMB/ECCB会议有发表相关文章。
香港城市大学
彭诚
博士研究生
彭诚:香港城市大学博士研究生,主要研究方向是深度学习在宏基因组中的应用等。
香港城市大学
孙燕妮
副教授,博士生导师
孙燕妮:香港城市大学电机工程系(Electrical Engineering)副教授,博士生导师。在美国圣路易斯的华盛顿大学(Washington University in Saint Louis)取得计算机系博士学位后在密西根州立大学计算机系担任助理教授和副教授(with tenure)。2018年加入香港城市大学。主要研究方向是生物信息学,序列分析,宏基因组学,和病毒基因组学。
具体的研究课题,发表的论文,以及实验室的招募请参加作者个人主页:https://yannisun.github.io/
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读
今天的文章Front Microbiol | 有尾噬菌体的科级分类:使用最新的ICTV分类标准评估当前分类工具…分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/88638.html