相关性分析
相关性是指两个变量的关联程度,两个随机变量之间的关系可以用相关系数来描述。相关系数的大小表示两者关系的强弱程度(若系数等于0则完全不相关,系数绝对值等于1表明完全相关),相关系数的正负值表示两者关系呈正相关或负相关。 衡量随机变量相关性的方法主要有三种:pearson相关系数,spearman相关系数和kendall相关系数。在本案例中,主要涉及的是pearson相关系数。笔者接下来将讲解如何通过R语言软件来实现变量间的相关性的统计计算,并绘制几种时下较为常用的相关性图形供大家学习参考,希望各位老师同行可以多多指点。
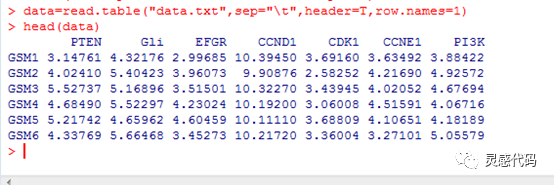
跟往常一样,我们在进行相关性分析之前,首先先准备好我们所需的数据。本次我们下载了GEO数据库的数据,该数据库也是一个常用的公共数据库。在R语言中,我们读取并查看数据前6行,我们可以看到,该数据以行对应每个样本,以列对应每个基因的表达, 如下:
在R语言本身是就带有计算相关性的函数cor.test,因此我们可以直接用该函数计算任意两个基因之间的相关性,结果如下:
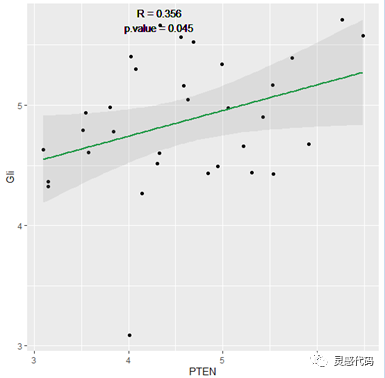
我们从以上结果得知,PTEN和Gli基因的相关性系数达到0.356,呈正相关,并且p值小于0.05,表明显著相关。以此类推,我们也可以计算其他基因之间的相关性。
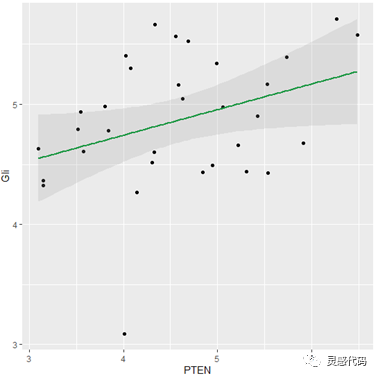
图形能够更加直观地表达两者之间的关系,因此我们可以选择相关性散点图,鉴于ggplot2在绘图方面的表现能力独一档,本次就使用该包来绘制相关性散点图。代码如下:
上图中,我们可以看到两种基因的表达情况之间的相关性,随着PTEN的表达升高,Gli的表达也同样呈上升趋势,因此两者具有正相关性。但我们也发现,图中缺乏相关性系数和p值,因此我们通过以下代码可以实现对两种参数的添加。代码如下:
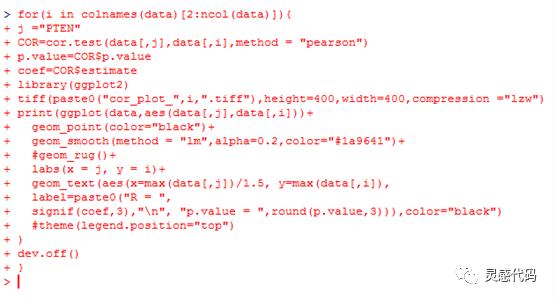
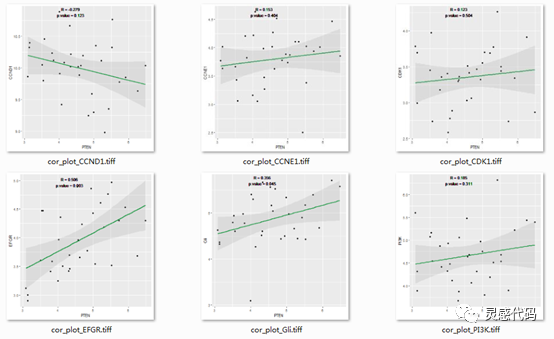
以上只是提到如何进行其中两种基因的相关性分析,但如果我们想探索PTEN与其他基因之间的相关性分析,就要改代码进行进一步的计算,我们想到,R语言中可以进行循环计算。在此,我们编写for循环函数,对每个基因与PTEN的表达相关性进行分析并绘制相关性散点图,代码如下:
我们对每个基因都与PTEN进行了相关性分析,并将相关性散点图输出到工作目录中。
其实,以上也不是最简单的方法,数据科学家早就想到了大家的需求。以下将介绍在R语言中两种更加简单的方法:
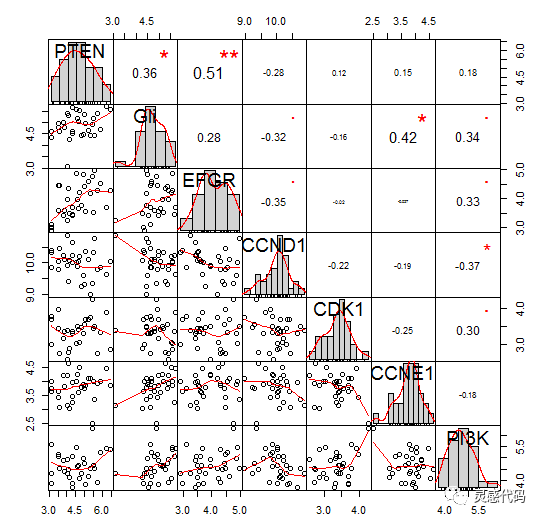
1. PerformanceAnalytics包
通过以上,简单的代码,我们即可得到如下结果,其中数字表示相关性系数,星号代表统计学显著性,星号越多,p值越小。
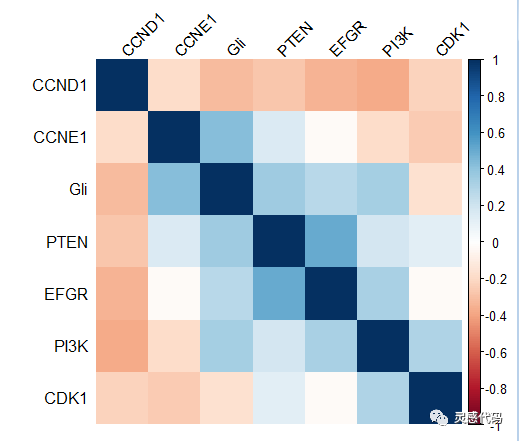
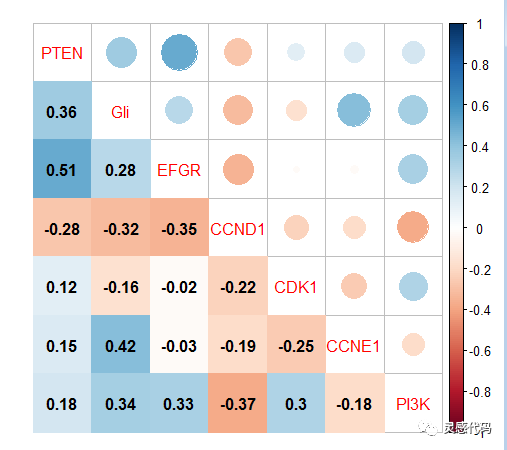
2.Corrplot包
Corrplot是另一种进行批量分析的方法,我们可以得到相关性热图,其中颜色的深浅代表相关性系数的大小,结果如下:
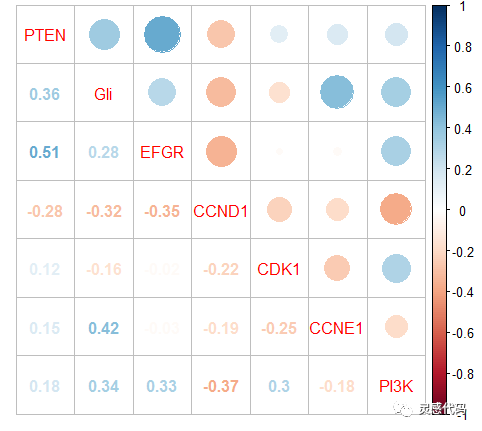
当然,我们也可以对其中的参数进行进一步修改,使图形更具可读性,如下:
这样的图形可能会更加美观一点。或者,我们还可以试着其他调整:
总之,在R语言中,我们可以利用多种方法对数据中的变量进行相关性分析。如果我们对其中某两个变量的相关性感兴趣,就可以利用先前讲到的ggplot2,如果我们想探索所有基因的相关性,那我们就可以利用corrplot或者PerformanceAnalytics了。
————————————————————————————————————————————
好了,今天的内容就跟大家分享到这里吧,如果大家感兴趣的话,可以加笔者的企鹅获取完整代码或做进一步的交流,谢谢!
或者关注我们的微信订阅号,关注即可获得我们完整的代码,我们后续还会不定时发布最新的内容供大家学习参考,谢谢支持!!!
今天的文章r语言相关性分析的结果怎么看_R语言如何检测数据的相关性分享到此就结束了,感谢您的阅读。
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 举报,一经查实,本站将立刻删除。
如需转载请保留出处:https://bianchenghao.cn/85346.html